染色质免疫共沉淀测序技术(Chromatin Immunopreciptation with Sequencing,ChIP-Seq),作为传统的研究细胞内蛋白质与DNA互作的重要工具,技术成熟,已被广泛应用于动植物的表观遗传学研究。但是由于ChIP的交联作用/免疫共沉淀的局限性(如:需要大量细胞,实验重复性差,低信号、高背景等),往往导致大量的精力投入却得不到预期的结果。CUT&Tag解除了传统ChIP-Seq的众多限制因素,势必将引领蛋白质—DNA互作新革命。
CUT&Tag(Cleavage Under Targets and Tagmentation)技术首次应用于哺乳动物细胞,并于2019年4月29日《Nature Communications》上公布[1]。
什么是CUT&Tag?
CUT&Tag是一种全新的研究DNA与蛋白质互作的技术,可以替代传统的ChIP-Seq,主要用于研究目标蛋白在染色质上的定位,在表观遗传学研究等领域至关重要。
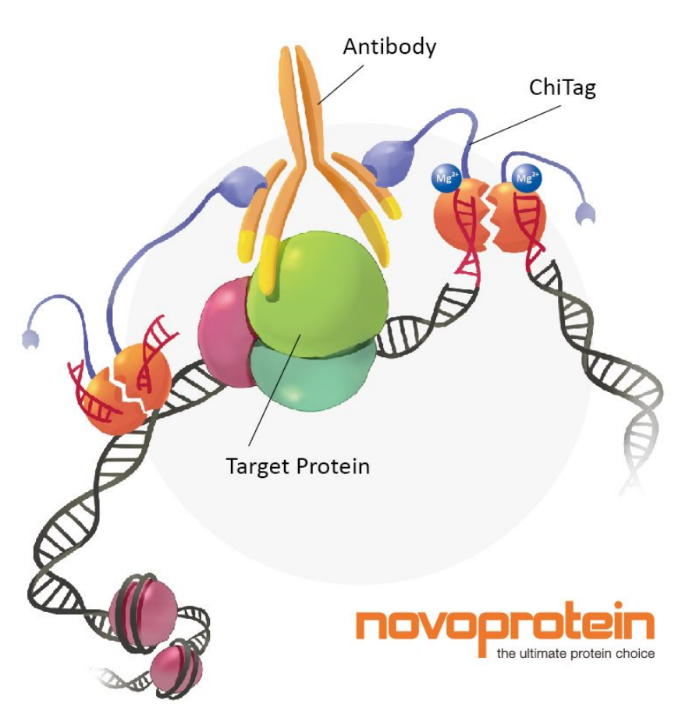
图1 CUT&Tag的基本原理
CUT&Tag使用Protein A与Tn5转座酶的融合蛋白(ChiTag),在抗体的引导下,Protein A与染色质上的目的组蛋白修饰标记、转录因子或染色质调控蛋白结合,连带的Tn5转座酶对目的蛋白附近的DNA序列进行切割,并将测序接头连接到切割片段的两端(图1),并释放到细胞外,PCR扩增后直接用于高通量测序。
与ChIP-Seq相比,CUT&Tag具有哪些优势?
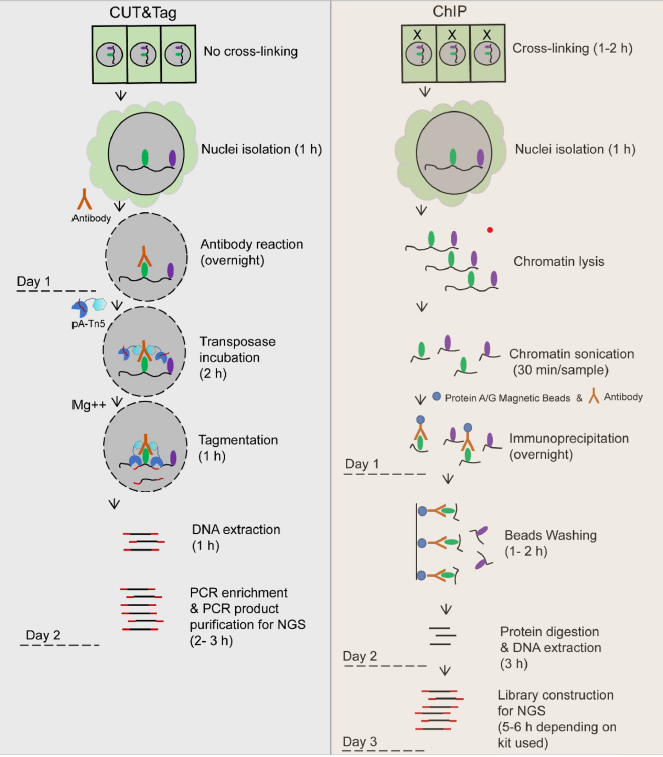
图2 CUT&Tag与ChIP-Seq实验流程对比[2]
(1)无需交联、超声打断等步骤,节约时间;
(2)不受ChIP交联引起的表位遮蔽影响;
(3)高分辨率和低背景信号(这是由于转座酶原位激活切割染色质);(4)转座酶切割片段两边序列能同时加上测序接头,为PCR扩增做准备;
(5)细胞需求量从10,000个降至60个甚至单细胞;
(6)需要的测序深度低,节省成本。
CUT&Tag在植物上很难成功?
植物细胞细胞壁、大液泡及一些次生代谢产物的存在,使得这项技术在植物中的应用多具挑战性。但是,Tao等于2020年8月31日在《Plant Methods》上发表CUT&Tag和ChIP-Seq技术在棉花应用的比较[2],种种结果表明,CUT&Tag技术能够在植物中成功应用,且所得结果明显优于ChIP-Seq。
H3K4me3是一个通用的基因表达活性标记。Tao等用棉花叶片为材料,以H3K4me3抗体设置了2个生物学重复,以IgG抗体作为对照用于CUT&Tag实验;用H3K4me3抗体做一个ChIP,不加H3K4me3抗体的ChIP mock作为对照。分别从以下几点对CUT&Tag和ChIP-Seq进行比较。
一、 重复性
对CUT&Tag和ChIP数据进行相关性分析,发现CUT&Tag的两个生物学重复(CUT&Tag_H3K4me3_rep1和CUT&Tag_H3K4me3_rep2)与对照CUT&Tag_IgG相关性非常低(r=0.01,Pearson’s correlation),表明CUT&Tag实验组和对照组(背景噪音)存在显著性差异(图3,a),然而,ChIP_H3K4me3实验组与ChIP_mock具有很高的相关性(r=0.89,Pearson’s correlation),这表明,ChIP分析中信号-噪音比例很高(图3,a),而CUT&Tag实验组的两个生物学重复相关性近乎完美(r=0.97,Pearson’s correlation),说明不同的生物学重复之间重复性非常好(图3,b)。
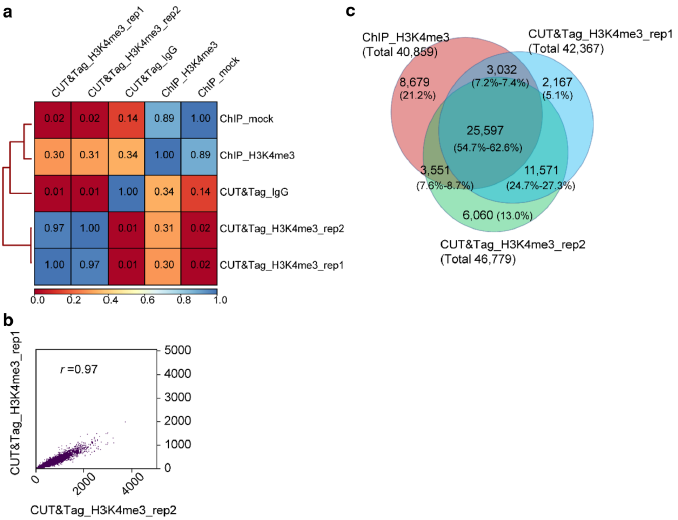
图3 CUT&Tag与ChIP样品的相关性分析。
二、分辨率(测序深度)
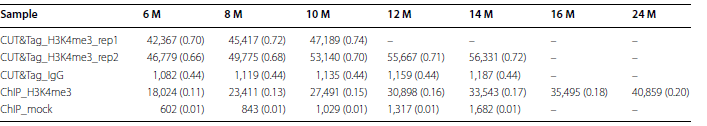
为了分析CUT&Tag和ChIP的数据的信号分辨率,每个样品随机选取6M-24M的clean data,分别call peak后发现CUT&Tag实验组的两个生物学重复在clean data为6M时,peaks数目分别为42,367和46,779,ChIP仅有18,024条,当ChIP的 clean data达到24M时,peaks数才达到40,859条(表1),这意味着CUT&Tag 6M clean data产生的信号分辨率相当于ChIP 24M clean data产生的。这些peaks中的25,597(54.7-62.6%)个是CUT&Tag与ChIP共有的,两个CUT&Tag生物学重复的共有peaks为37,168(79.5-87.7%;图3,c),这也说明CUT&Tag的高重现性。
表1 相同测序深度下CUT&Tag及ChIP产生的 peaks数及FRiP值
三、 信噪比
FRiP(fraction of reads in peaks)values calculated the ratio of mapped reads that fall into peaks among all mapped reads. FRiP值越高,信噪比越高,结果也越可靠。结果表明CUT&Tag产生的信噪比较高(FRiP=0.7;表1)。然而,从H3K4me3信号热图上可以看出,在整个gene body区域CUT&Tag的信号比ChIP-Seq更强(图4,a)。从相关性分析的结果可以看出,基因附近的peaks在CUT&Tag两个生物学重复之间相关性很高(r=0.94,pearson’s correlation;图4,b),CUT&Tag和ChIP之间的相关性也较高(r=0.71,pearson’s correlation;图4,c)。
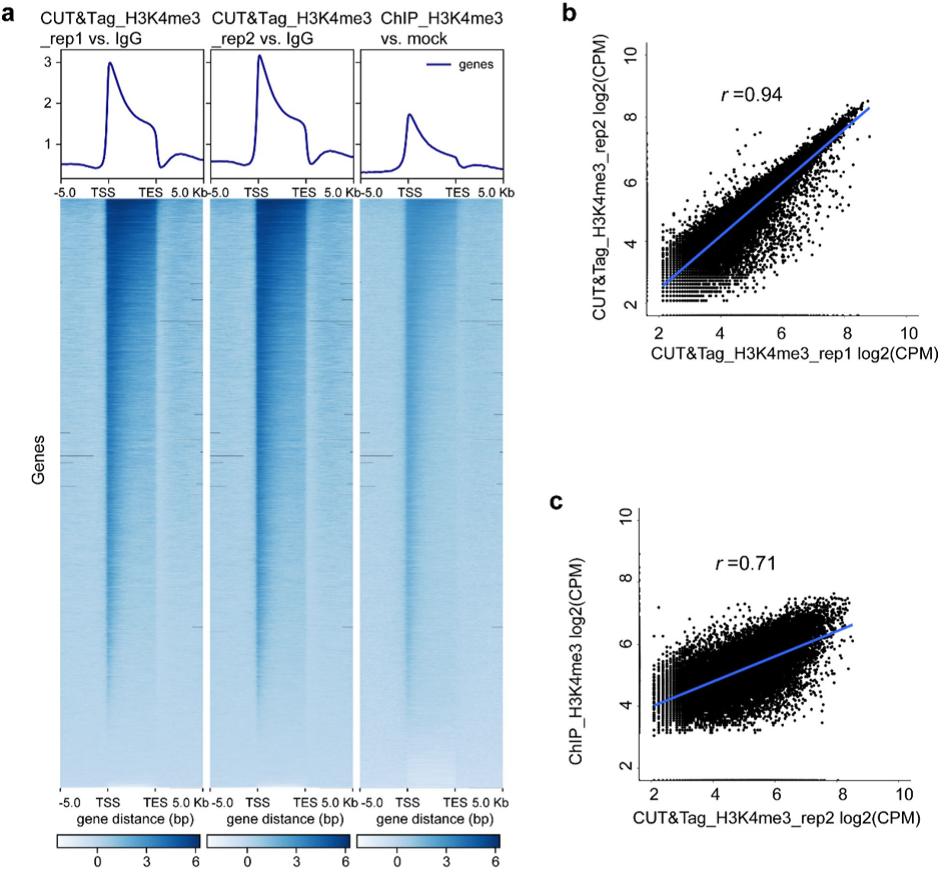
图4 CUT&Tag和ChIP分析蛋白质编码基因附近的H3K4me3信号
四、灵敏性
与热图显示的强度一致,IGV截图表明CUT&Tag信号在分辨率和灵敏度上的表现优于ChIP(图5,a),尤其在表达量相对低的基因中表现尤为明显(如GB_A11G1394, GB_D10G1774, 和 GB_A13G1872;图5,b)

图5 有代表性的H3K4me4信号的IGV截图
综上所述,Tao等对Steven Henikoff团队研发的CUT&Tag技术进行改进,打破植物细胞壁、大液泡和次生代谢产物的壁垒,使其能够广泛应用于植物染色质的释放。CUT&Tag与传统的ChIP-Seq相比,操作简单,所需时间短,且所需细胞少,能产生高分辨率信号,且背景噪音低,这些优点奠定了不久的将来该技术在表观遗传学研究中的“流量新秀”的地位。
参考文献:







 京公网安备 11011302003368号
京公网安备 11011302003368号