近日,佛罗里达大学的Meixia Zhao教授团队在国际顶尖期刊《Nature Communications》上发表了题为“mop1 affects maize recombination landscapes by modulating methylation of MITES near genes in open chromatin”的研究论文。
该研究揭示了玉米中一个关键的表观遗传调控因子mop1在塑造减数分裂重组图景中的核心作用。通过高分辨率的全基因组重组图谱分析,研究团队发现,mop1功能的缺失会以一种性别特异性的方式改变重组事件的发生位置,其背后的机制是通过调控一类被称为“微型反向重复转座子”(MITEs)的特殊DNA元件的甲基化水平,从而在特定的基因组位、尤其是遗传多样性高的区域“点燃”新的重组热点。百迈客生物为该研究提供了转录组测序及分析服务!

研究背景
减数分裂重组是地球上有性生殖生物多样性的基石。在细胞分裂形成配子(如花粉和卵细胞)的过程中,来自父母本的同源染色体像舞伴一样配对、交换遗传物质片段,这一过程被称为“重组交换”(Crossover, CO)。它不仅打乱并重排了基因,创造出新的遗传组合,还像“分子胶水”一样确保染色体在分裂时能被正确地分离,避免了毁灭性的遗传错误。然而,这个过程并非随机发生,而是受到一套极其精密的调控网络所控制。在玉米这样基因组庞大且复杂的作物中,重组事件往往集中在特定的“热点”区域,而广阔的基因组区域则鲜有重组发生,如同“冰封”的大陆,这极大地限制了育种家利用优异基因资源的能力。近年来,表观遗传学,尤其是DNA甲基化,被认为是调控重组的关键因素之一,但其具体机制仍充满谜团。
研究内容及结果
1、mop1改变雌雄减数重组格局
构建群体与CO定位
- 作者在B73×Mo17杂交背景下,做了4个BC1群体:雌WT、雌mop1、雄WT、雄mop1,共423个个体。
- 全基因组3×深度重测序,利用~1.9 SNP/kb的密度,HMM调用出4048个CO,其中60%的CO区间<5kb,分辨率很高。
CO 总数的性别差异(Fig.1d)
- 雌性:WT和mop1每减数平均CO数基本相同(约19条,差异不显著)。
- 雄性:mop1 CO数略低于WT(约18vs19.4,P=0.058,接近显著)。
- WT中雌雄CO数相似;但在mop1中,雄性显著低于雌性,说明mop1参与调控雌雄间的重组差异。
染色体尺度的CO分布(Fig.1c, 1e)
用Marey map画出遗传距离与重组率曲线,结果显示全基因组的重组率形状在WT与mop1间总体相似,也与雌雄之间大体相似。但放大单条染色体后,可以看到:尤其在雌mop1中,多条染色体(1,4,5,6,7,8,9)在臂区CO增加,在着丝粒周CO减少;雄mop1也在多条染色体呈现类似趋势。
结论:mop1对总CO数影响相对温和,但会在局部区域重排CO峰谷,且这种效应具性别特异性。
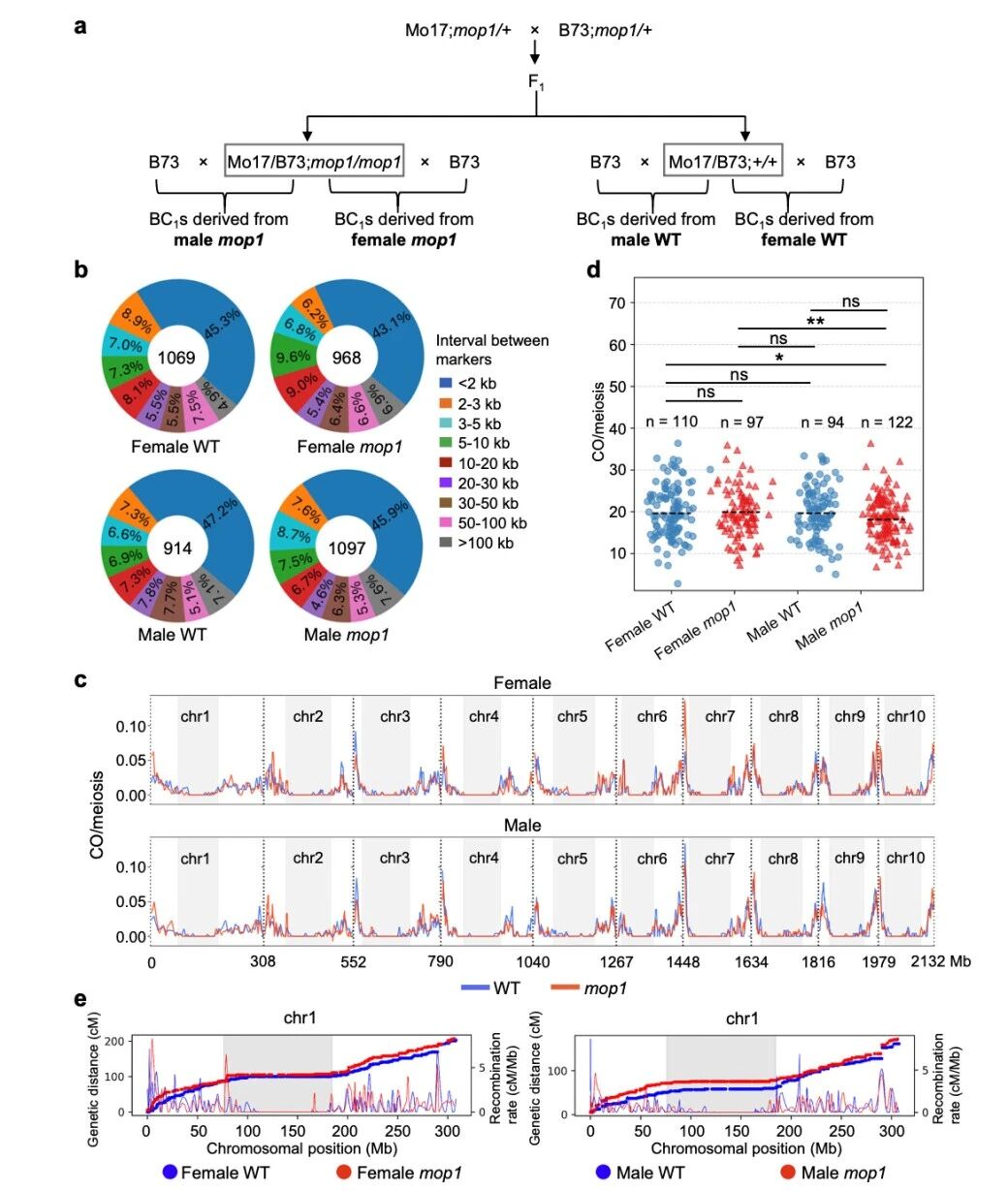
图1 雄性mop1突变体与雌性mop1突变体相比,交叉(CO)数量减少。
2、CO相关序列motif的变化
筛选“高分辨率CO”
作者只选CO区间<2 kb的1835个CO,分别来自四个群体(雌WT 484、雌mop1 417、雄WT 431、雄mop1 503),用来做motif分析。
主要发现(Fig.2)
- 前五大motif都是富C/G或富A/T的短重复序列,和之前在玉米、小麦、水稻中CO常见motif一致。
- 定量统计显示一个细节:富GC的motif(前三个)的出现频率在雄mop1中明显比雄WT低;富AT的motif(后两个)在雌mop1中的出现频率高于雌WT。
解读:mop1使雄性CO更少落在GC-rich区,而让雌性CO更偏向AT-rich(往往更开放)区域,体现出性别特异的序列/染色质偏好变化。
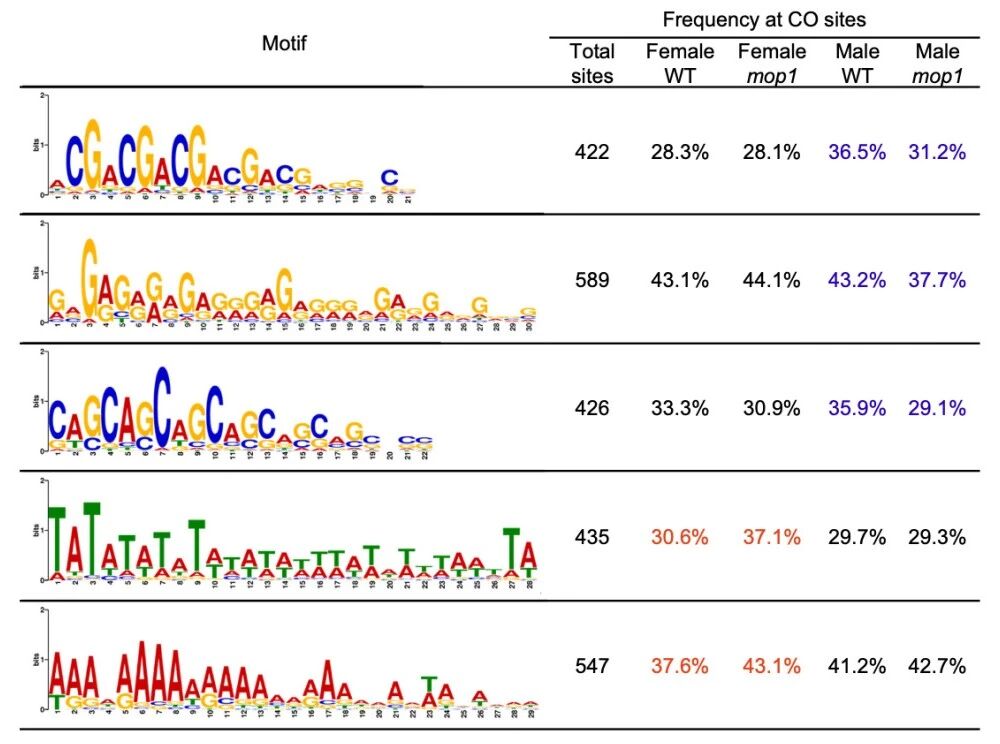
图2 在雄性mop1突变体中,G/C相关基序的频率降低,而在雌性mop1交叉互换(COs)中A/T相关基序的频率增加。
3、序列多态性与CO的关系
SNP密度与CO
- 在雌mop1的CO区间中,SNP密度明显高于雌WT;雄性则无显著差别。
- 把全基因组细分不同SNP密度区间,统计CO频率:
雌WT:CO 频率在~7 SNP/kb 达峰;
雌mop1:峰值移动到~13 SNP/kb;
SNP>20/kb 时CO频率接近0,呈明显“倒 U 型”,存在上限阈值。
InDel与CO
- mop1与WT之间,CO区域的InDel数量/长度无显著差别。
- CO对InDel的容忍度很低:4 InDel/kb左右达到峰值,达到12个 InDel/kb时几乎没有CO,说明InDel比SNP更干扰重组。
结论:存在一个“适中多态性”窗口,既不能太低(重组驱动力不足),也不能太高(同源配对困难)。在雌mop1中,这个SNP阈值被显著抬高(从7→13 SNP/kb),说明RdDM缺失让雌性减数更能容忍高多态背景下的CO。
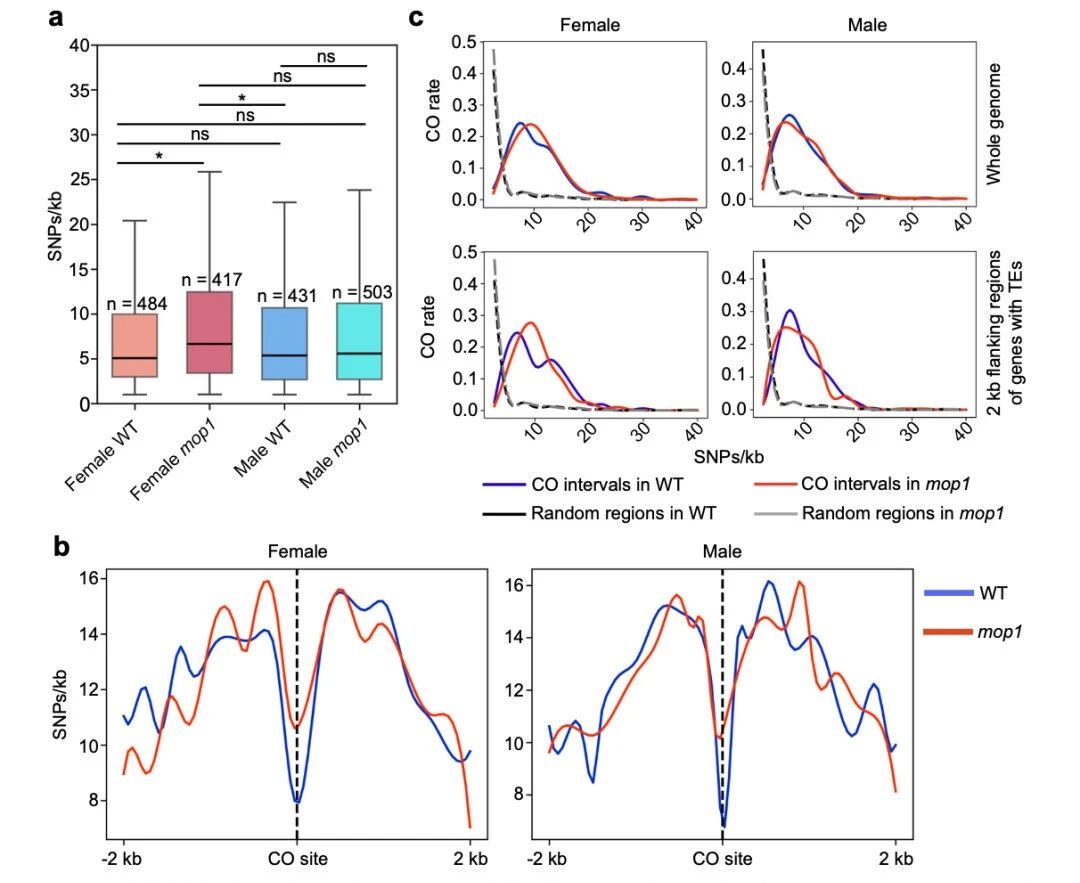
图3 mop1基因,尤其在雌性个体中,倾向于在遗传多样性较高的区域引入新的交叉(CO)位点。
4、CO与转座子,尤其MITEs的紧密关系
CO与基因/TE的位置关系(Fig.4a):1835个高分辨率CO中,约69%位于基因体或2kb上下游,其中44.3%同时与TE重叠。
不同TE类型与CO的相关性(Fig.4b–c):按1Mb窗口算CO数与TE覆盖度:CO与LTR/Gypsy强负相关(主要在周围冷区)。与LINE、TIR、Helitron、MITE均正相关,其中MITE的正相关最强。
MITEs与CO直接重叠的比例(Fig.4d):随机选1835个等长区:仅1.5%与MITE重叠;而CO区中:雌WT:33.8%CO 与MITE重叠;雌mop1:升到39.3%;雄WT:31.8%;雄mop1:34.7%。 说明:CO极度偏爱MITEs,且mop1尤其在雌性中进一步加强这种偏好。
哪类MITE更“吸CO”?(Fig.4e):在MITE子家族中,**DTA(hAT类)和DTH(Tourist/PIF-Harbinger类)**最常与CO重叠,占比明显高于其他子家族。
CO热点与冷点(Fig.5)
- CO热点:重组率≥全基因组均值5倍的5kb区间。总数在四个群体为391–525个。它们多数位于染色体两端:39.1% 在前10Mb,63.9%在前20Mb。热点2kb区内,~65%附近有MITEs,而附近是LTR的比例很低。
- CO冷点:≥1Mb无任何CO的大区间。四个群体分别有~1167–1310个,绝大多数位于着丝粒周,且在不同群体间高度保守。冷点几乎都与LTR retrotransposons重叠,与MITEs却很少重叠。
总结:MITEs≈重组热点的标志;LTR retrotransposons≈稳定冷点骨架。mop1通过调控MITEs,精细地改写了部分热点,而不改变那些“固有冷点”。
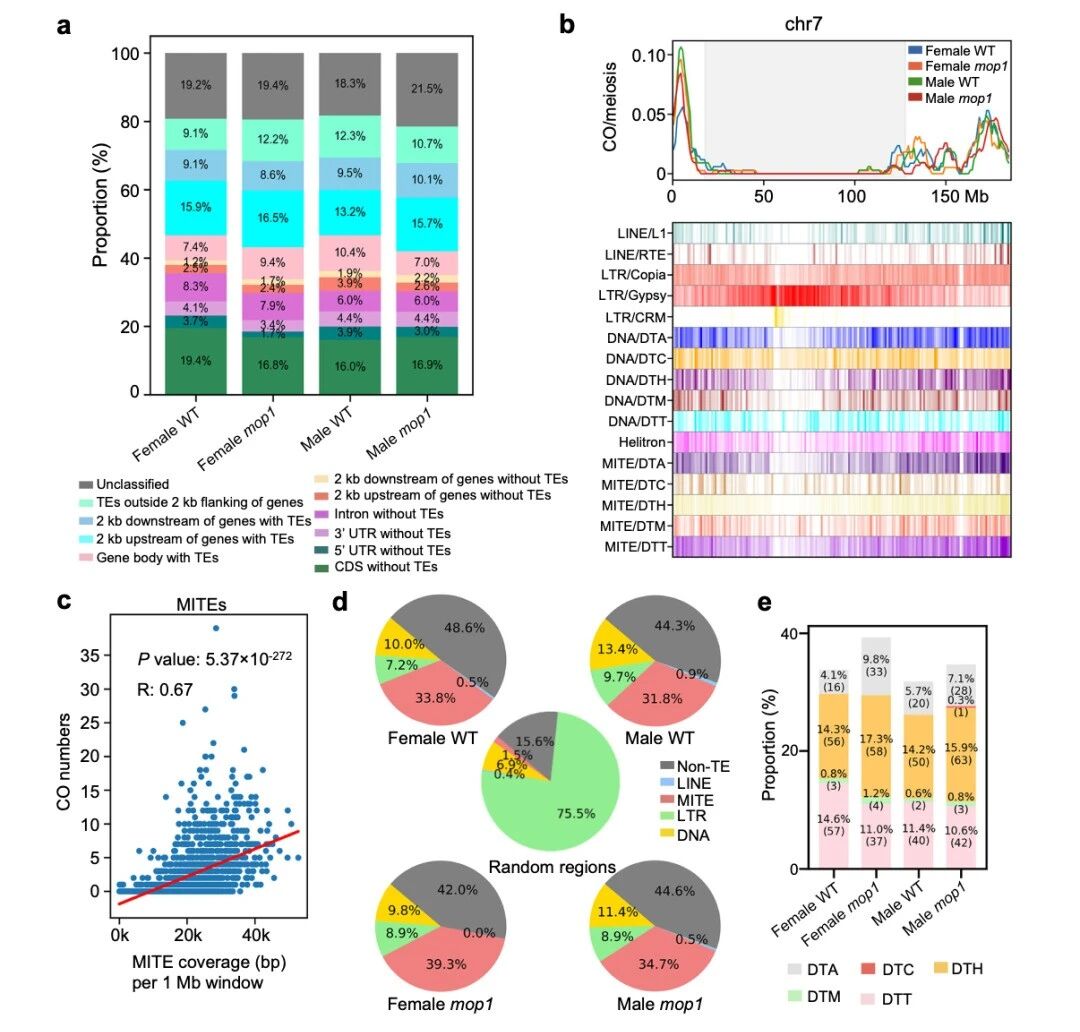
图4 交叉变异(COs)与微RNA元件(MITEs)相关联,在mop1突变体中观察到更高的富集程度
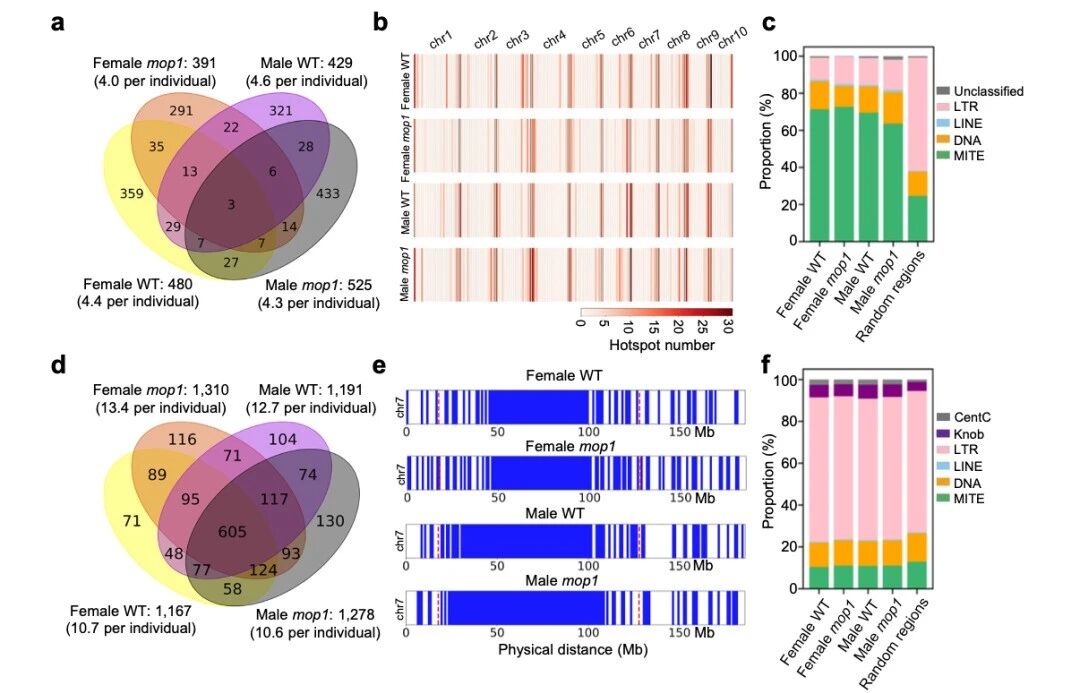
图5 交叉位点(CO)在MITE序列附近富集,而冷点则与LTR逆转座子相关联
5、mop1通过MITEs上的局部去甲基化+开放染色质引入新CO
CO区DNA甲基化总体特征(Fig.6a)
- 与随机区相比,CO附近的CG/CHG甲基化大幅降低(CG: 23.8%vs78.3%;CHG: 12.4%vs60.4%),说明CO天然偏向低甲基化区域。
- CG/CHG:mop1和WT在CO区没有显著差异。
- CHH:WT中CO区CHH与随机区相当,而在mop1中显著降低。也就是mop1主要在CO区进一步清空CHH甲基化。
DMR与CO的重叠情况(Fig.6b–c)
在雄mop1与WT花药甲基化数据中,作者找到3189个CG DMR、4800个CHG DMR、13864个CHH DMR,其中大多数是低甲基化(hypo)。这些成千上万DMR中,与CO±2kb重叠的不到1%,其中CG:9/2749;CHG:30/4110;CHH:101/12061。且这些与CO重叠的DMR,绝大部分都位于 基因2kb上下游区域。
这些DMR上到底是哪些TE?(Fig.6d)在“DMR+CO”双重重叠的TE中,MITEs占比最高,明显高于仅有DMR或无DMR/CO的TE。这说明:mop1引起的CO变化主要集中在MITEs,尤其是靠近基因的MITEs上。
MITEs上的甲基化&组蛋白标记分组分析(Fig.6e–f)
- 按是否与DMR/CO重叠,把MITEs分4类:既有DMR又有CO;有DMR无CO;无DMR有CO;都没有。
- 结果:在mop1中,类别1的MITEs CHG/CHH甲基化降低最明显;同时,类别1上H2A.Z、H3K27ac、H3K56ac、H3K9ac活性标记最高;完全没有DMR/CO的MITEs这些标记最低。即:“真正变成CO热点”的MITEs=低甲基化+高H2A.Z+高 H3乙酰化+位于基因附近。
49kb典型区域的可视化例子(Fig.6g)
- 染色体9上49 kb区间中有两个CHH低甲基化DMR:
- 红框:一个DMR 同时有 MITE+CO,且H2A.Z、H3 acetylation高,H3K27me3低,mop1中还伴随CG/CHG降低;
- 蓝框:另一个DMR也有MITE,却没有CO,CG/CHG一直高,且H3K27me3高、H3Ac低。
- 这对比说明:单靠CHH降低不够,还要配合整体开放染色质(低CG/CHG+高H2A.Z/H3Ac+低H3K27me3),MITEs才会真实变成CO热点。
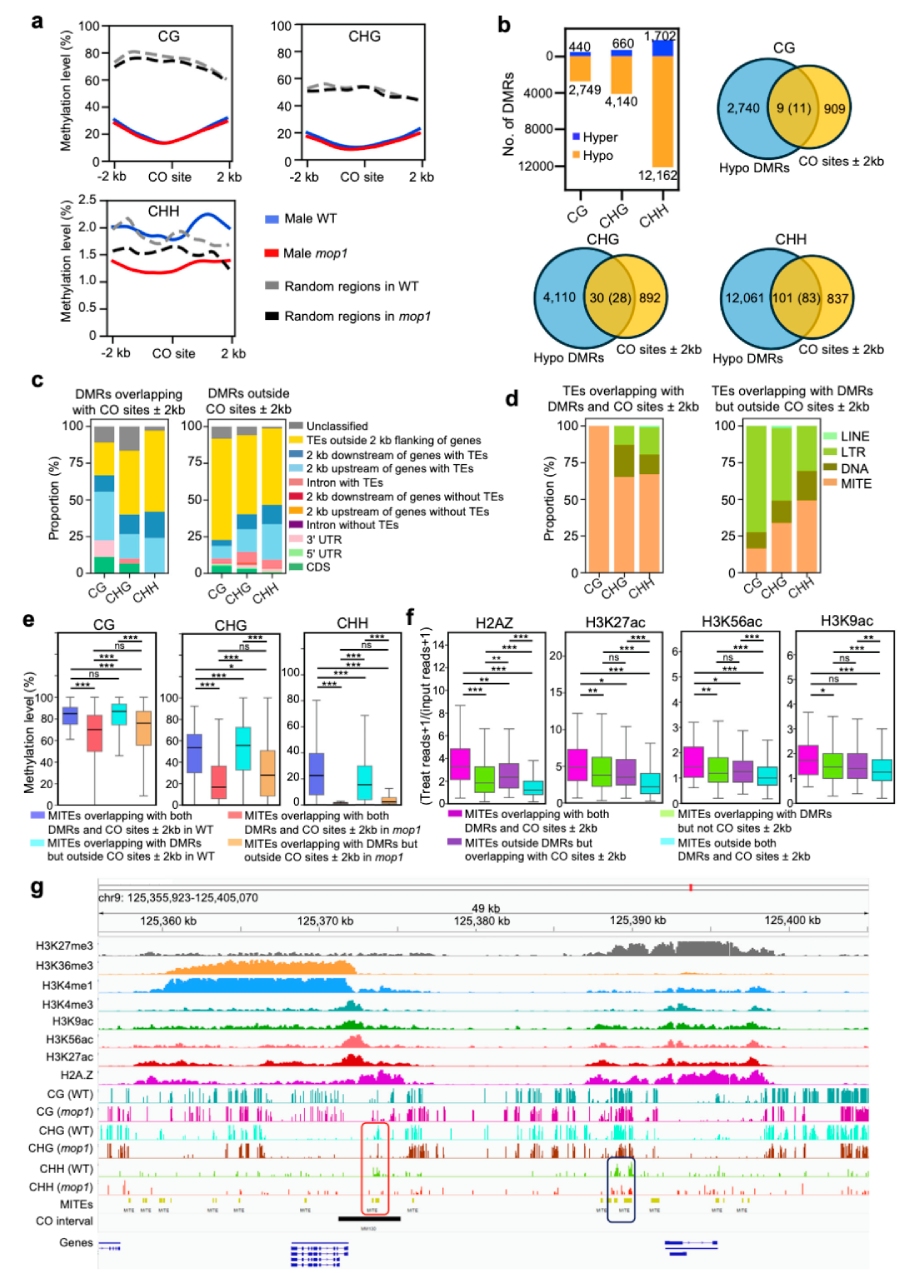
图6 mop1可能通过局部降低具有开放染色质标记的MITE序列上的DNA甲基化水平
引入新的交叉(CO)位点
6、基因表达和CO干涉:ASY4下调,但干涉基本不变
RNA-seq:减数基因表达变化
- 在幼花药中,mop1vsWT 共有186个上调、199个下调DEG。
- 若只看41个减数相关基因:只有轴蛋白ASY4显著下调;ZYP1、ZIP4接近显著下调。
- 这些基因负责轴/联会复合体结构和CO干涉,Arabidopsis中它们缺失会让CO向染色体臂端偏移。
DEG与DMR、CO的关系
只有极少部分DEG附近有 DMR,且没有任何CO与这些DEG的DMR 重叠,说明 MITEs去甲基化带来的新CO不必然改变附近基因的转录。
CO干涉分析
作者用crossover coefficient(CoC)分析干涉强度,发现:mop1雌雄的干涉与WT基本无显著差异,只是雌mop1干涉略有减弱趋势。因此,CO干涉变化不是驱动CO重排的主要原因。
解释:mop1的主要作用,是通过局部改变MITEs的表观状态+略微削弱轴/SC 组件来调整CO的位置,而不是大幅改变CO总数或经典的干涉模型。
研究总结
在机制层面,mop1通过RdDM通路主要清除基因附近MITEs的CHH(并部分 CG/CHG)甲基化,在本就偏开放的MITE区促进H2A.Z和H3 acetylation富集,从而让这些TE成为新的CO热点。性别差异:这一作用在雌性中更强——雌mop1 不仅保留CO总数,还把CO推向高SNP、MITE富集的开放区域,提高了对序列多态性的容忍度。结构蛋白贡献:mop1还伴随轴蛋白ASY4下调,可能进一步改变CO在染色体上的分布,而对CO干涉整体影响不大。
在应用前景方面,提示可以通过精细调控MITEs附近的甲基化/表观状态,而不是粗暴打掉全基因组甲基化,来在作物中局部提升重组率;有望用于玉米及其他TE-rich作物中打破连锁拖带、提高育种重组效率、利用“中度多态区域”的隐藏遗传变异。
以上内容来源于清同趣科研,侵删







 京公网安备 11011302003368号
京公网安备 11011302003368号