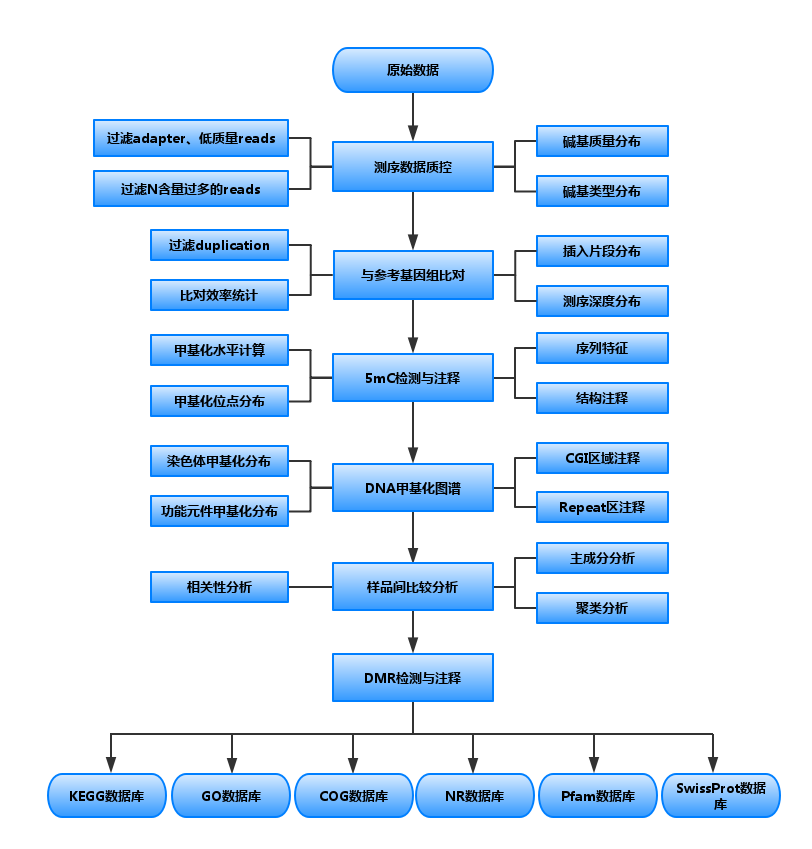
全基因组甲基化测序分析
产品介绍
DNA甲基化是重要的表观遗传学标记信息,获得全基因组范围内所有C位点的甲基化水平数据,对表观遗传学的时空特异性研究具有重要意义。Bisulfite甲基化测序是以新一代高通量测序平台为基础,结合全基因组Bisulfite处理和生物信息数据分析技术,进行低成本、高效率、高准确度的全基因组DNA甲基化水平图谱绘制。
分析内容流程
结果展示
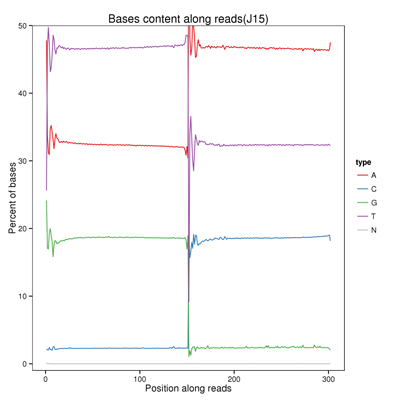
碱基类型分布
碱基类型分布检查用于检测有无AT、GC分离现象。若无bisulfite处理,高通量所测序列为基因组随机打断后的DNA片段,由于位点在基因组上的分布是近似均匀的,同时,G/C、A/T含量也是近似均匀的。全基因组甲基化项目中经过bisulfite处理后的基因组中大部分的C位点变成T位点,因此其中一条链出现T的含量增加而C的含量剧减,同时对应的反向链出现A的含量增加而G的含量减少。
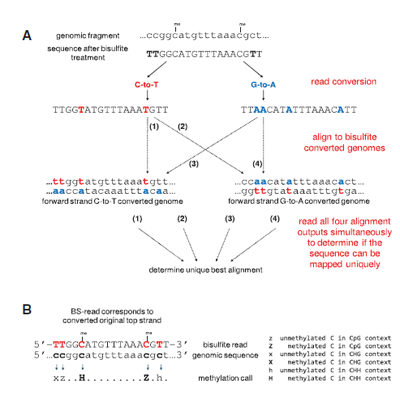
5mC检测
5mC的检测主要使用Bismark软件工具包实现。由于样品经过重亚硫酸处理,基因组上所有的未甲基化的C位点转换成了U;经过PCR扩增U->T;即基因组上未甲基化的C转化成了T;而原本甲基化的C;没有改变;相应的负链G->A;bismark是专门用于甲基化处理的比对软件;他会对reads进行转变;根据Clean Reads在参考基因组的最佳的比对结果,提取全基因组胞嘧啶(C)位点的比对碱基信息,最终得到5mC位点覆盖统计。
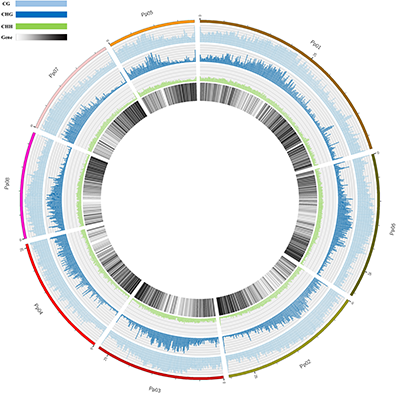
染色体水平甲基化分布图谱
在每条染色体上,以100K大小为一窗口来计算每个窗口的甲基化水平,继而从染色体水平描述甲基化 C 碱基的分布情况,绘制全基因组染色体水平甲基化图谱,可以很直观的呈现出染色体某个区域甲基化水平。
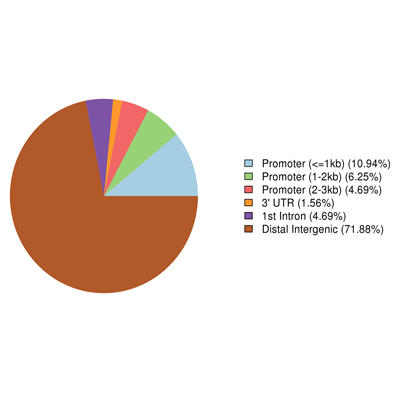
高甲基化CGI区域注释
对高甲基化密度的CGI区域进行注释,区域甲基化水平大于0.7我们认为是高甲基化的CGI区域;同时我们把覆盖度大于5X的C位点认为是可信度较高的位点,去除CGI 区域内可信度较高的C位点的比例小于0.1的CGI区域;对剩下的区域进行注释;取基因上游3000bp为promoter区,进行overlap注释。
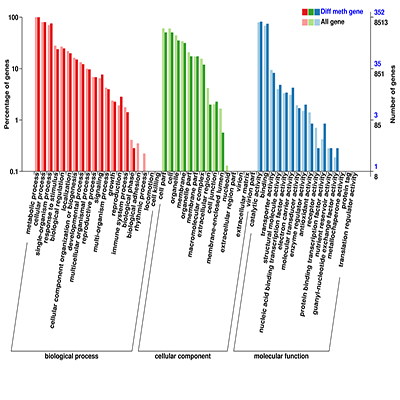
DMR相关GO基因分类注释
DMR相关基因GO注释分类统计图,直观的反映出在生物过程(biological process)、细胞组分(cellular component)
和分子功能(molecular function),所有基因和差异基因注释GO term的个数分布。可深入挖掘DMR相关基因的功能及所在的信号通路,筛选关注差异基因注释情况。
DMR关联基因代谢通路分析
生物体内,某些基因位点受到甲基化修饰之后,会影响该基因的表达,从而改变生物学表型。因此,对DMR关联基因分别进行GO和KEGG注释及富集分析,有助于深入挖掘甲基化修饰引起的生物学功能变化。
成功案例
- 《Nature Genetics》丨亚基因组的平行趋同选择是导致芸薹和甘蓝的形态型多样化以及趋同驯化的主要动力2016年8月15日,《自然-遗传学》(Nature Genetics)杂志在线发表了中国农业科学院蔬菜花卉研 […]阅读更多
常见问题
答:.DMR是用Bisulfighter软件检测的,详细可以参考Bisulfighter的相关文献(Bisulfighter:accuratedetectionofmethylatedcytosinesanddifferentiallymethylatedregions)。DMR是差异甲基化区域,长度不固定,长度为1KB的DMR中的任意一段不一定是DMR,如果取得的任意一段没有甲基化的差异就不是DMR,如果有多个DMR连在一起,我们便将这些DMR合并成一个大的DMR。
答:甲基化水平计算方法共分四种:
1)单个位点甲基化水平:
单个位点的甲基化水平=支持甲基化C的reads数/总的reads数
注: 对于参考基因组上的C位点,有些reads(reads上此位置处是C)支持此位点是甲基化的C位点,有些reads不支持(reads上此位置是T), 那么单个位点的甲基化水平就是支持甲基化的reads数/(支持甲基化的reads数+不支持甲基化的reads数)
计算得到单个位点的甲基化水平后,我们用二项分布检验判断某位点是否为甲基化C位点。
用从左开始第二个C计算单个位点甲基化水平。
对于某个区域的甲基化水平计算方法,不同的方法会得到不同的结果。
2)区域甲基化水平计算方法1-Fraction of methylated cytosines
即:甲基化C占区域内所有覆盖到的C位点的比例
对于图片中的a:区域甲基化水平=10/12;b)甲基化水平=11/12
3)区域甲基化水平计算方法2-Mean methylation level
即:所有C位点的平均甲基化水平,区域内所有甲基化的C的单个位点甲基化水平之和/区域内所有覆盖到的C位点的个数
a)甲基化水平=(2/2+3/3+20/21+4/30+4/25+3/17+3/20+6/7+4/6+5/5)/12
b)甲基化水平=(1/5+1/3+3/12+40/41+29/31+30/32+77/87+45/47+4/12+1/7+2/10)/12
4)区域甲基化水平计算方法3-Weighted methylation level
即:加权甲基化水平,区域内所有甲基化C位点总的reads数/区域内总的覆盖度
a)甲基化水平=(2+3+20+4+4+3+3+6+4+5)/(2+3+21+30+25+17+20+7+6+5+18+25)
b)甲基化水平=(1+1+3+40+29+30+77+45+4+1+2)/(5+3+12+16+41+31+32+87+47+12+7+10
相关文章
- NG|ONT重测序&甲基化应用案例|DNA甲基化reader vs eraser本研究使用一组基因敲除的人类胚胎干细胞(ESC)系,利用二代和nanopore三代DNA甲基化测序技术,分析了 […]阅读更多






 京公网安备 11011302003368号
京公网安备 11011302003368号