宏基因组(NGS)
宏基因组技术介绍
宏基因测序是通过高通量测序技术对环境样品中全部微小生物遗传物质,包含了可培养的和未可培养的微生物的基因组进行测序分析,以微生物多样性、种群结构、进化关系、功能活性、相互协作关系及与环境之间的关系为研究目的的新的微生物研究方法。
产品热点
无需分离培养,可鉴定环境样品中物种组成和功能组成;
分辨率高,可检测环境样品中极低丰度物种;
可获得环境样品中全面的功能基因信息,涵盖碳氮磷硫代谢、抗性基因等;
可组装获得环境中单菌基因组信息 ;
实验设计到信息分析一站式服务
提供全面的宏基因组研究的方案设计和售后服务,丰富的标准分析以及个性化分析多角度阐明环境微生物潜在的功能和作用机制。
-
方案确定
-
提取检测
-
建库测序
-
信息分析
-
售后服务
分析内容
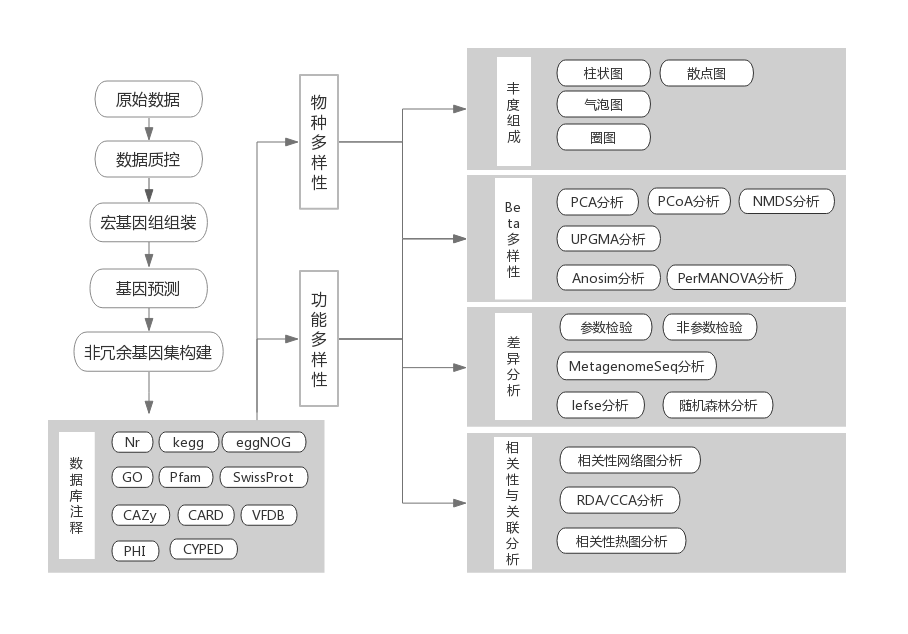
技术策略与送样要求
测序策略:NovaSeq PE150
DNA送样要求:DNA浓度≥1ng/ul(Qubit),DNA质量≥30μg(Qubit),DNA电泳条带清晰,无降解或者轻度降解。
| 环境样品 | 送样要求 | 保存及运输 |
| 普通土壤 | 除去土壤表层未分解的凋落物层、动植物残体、石砾等杂质,将大块的样品捣碎,过2mm筛后,分装至2mL或更大体积的EP管或冻存管中;每管土壤含量大概0.25~0.5g,需保证送样量在1~2g。 | 样本-80℃或液氮中长期保存,干冰运输 |
| 根际土壤 | 收集植物植株,去除根部大块土壤;摇动植株去除附着不紧密的土壤,使用无菌刷子收集根部附着紧密的土壤;随机多点取样5-10g,过2mm筛后,分装至2mL或更大体积的EP管或冻存管中;每管土壤含量大概0.25~0.5g,需保证送样量在1~2g。 | |
| 粪便 | 无菌牙签或粪便取样器截取样品中段内部(避免表层中的肠道膜脱落细胞),外部容易污染且细菌DNA由于接触空气可能有降解。将已取的粪便样品分装至2mL EP管(无菌)或冻存管(无菌)中,每管粪便量为0.5~2g,每个样品分装2~3管备份。 | |
| 肠道内容物 | 在实验对象死亡后,无菌条件下,取出整个肠道,用无菌解剖刀切取所需肠段的,用无菌手术刀挖取内容物分装至2mL EP管(无菌)或冻存管(无菌)中,每管组织量为0.5~2g,每个样品分装2~3管备份 | |
| 水体 | 过滤大量低微生物含量的清亮水样用0.22μm 的聚苯醚砜滤膜,每个样本至少1L水样。浑浊水样使用0.22μm滤膜过滤缓慢容易堵塞时,建议使用0.45μm的混合纤维素酯滤膜,每个样本0.5L-1L水样;如果水样中不可溶解的颗粒较多,需要使用2-5μm孔径的滤膜将不可溶解的颗粒杂质滤去,再使用0.22μm或0.45μm的滤膜富集菌体,每个样本0.5L-1L水样。 | |
| 空气 | 开动采样仪(浮游细菌采集器),使被测空气滤过凝胶膜(灭菌),空气中的浮游菌被截留在凝胶膜上(过滤时间越长,收集的空气中的灰尘越多,菌数量越多),收集完成后取下滤膜。 |
产品优势与项目经验
丰富的项目经验与专业的信息团队,提供全面准确的信息分析。
成功案例
物种注释
使用diamond软件将质控后的高质量测序数据比对到 nr数据库,得到样品的物种组成和相对丰度信息。
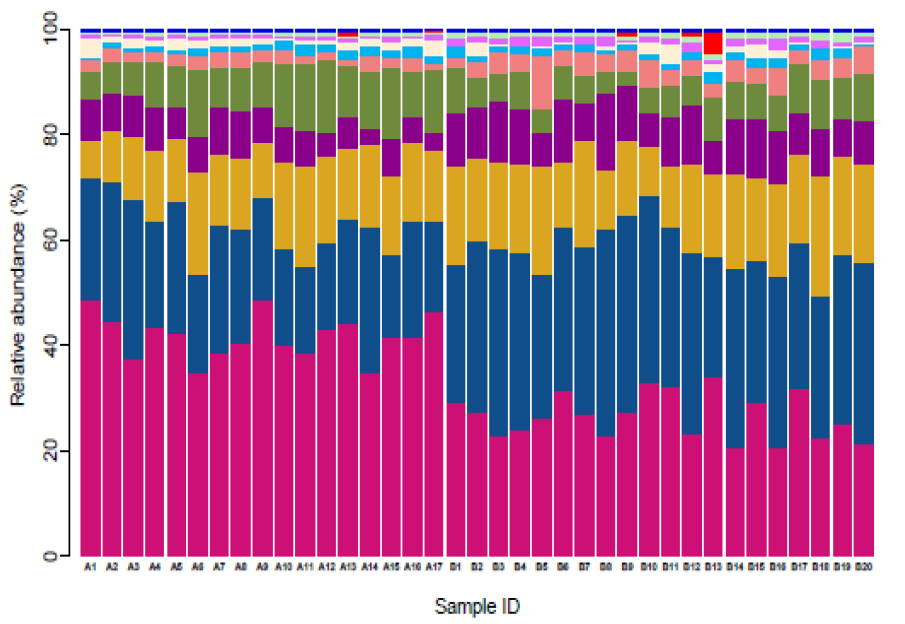
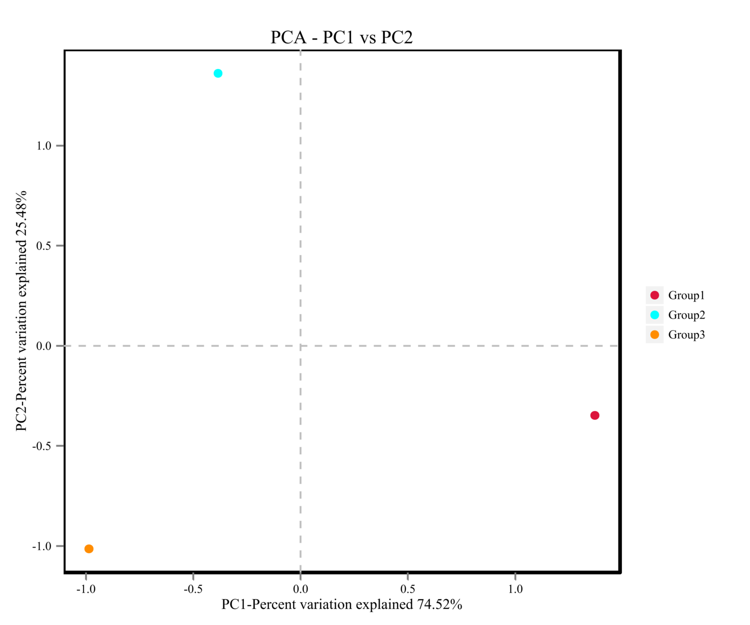
物种组成的主成分分析
主成分分析(Principal Component Analysis, PCA)是一种分析和简化数据集的技术,通过将方差进行分解,将多组数据的差异反映在二维坐标图上,坐标轴取能够最大反映方差 的两个特征值。通过分析不同样品物种组成可以反映样品间的差异和距离。PCA图上两个样品距离越近,则表示这两个样品中物种的组成越相似。使用R语言工具分别绘制不同分类水平PCA分析图
物种进化分析
基于样品物种组成信息,使用软件MetaPhlAn2绘制物种进化分枝图,可以展示样品所含物种的系统进化信息。

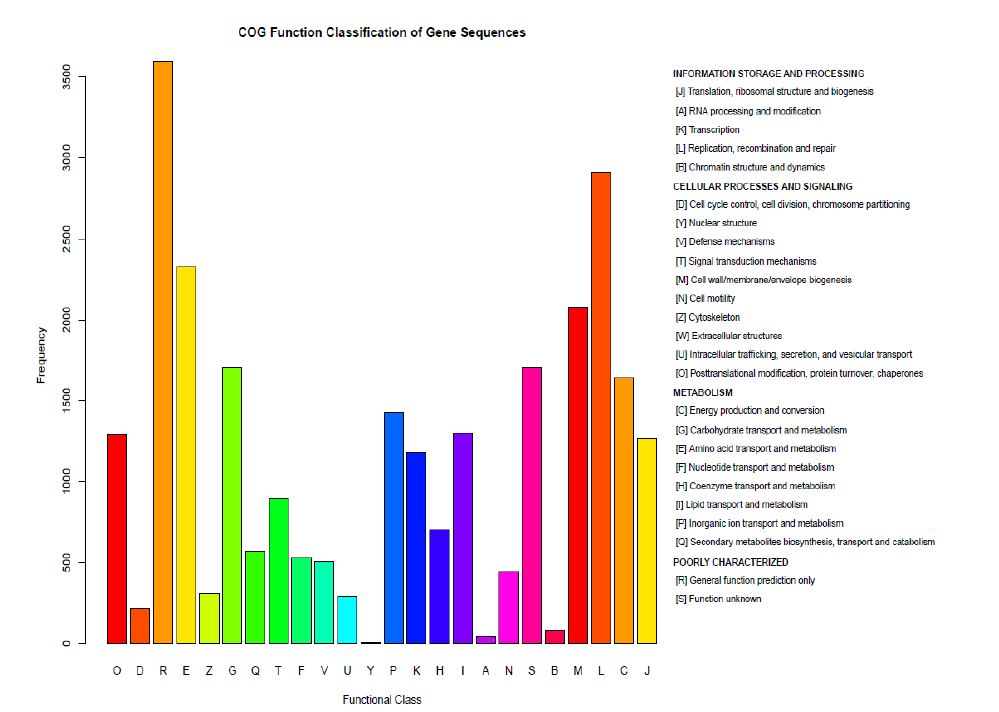
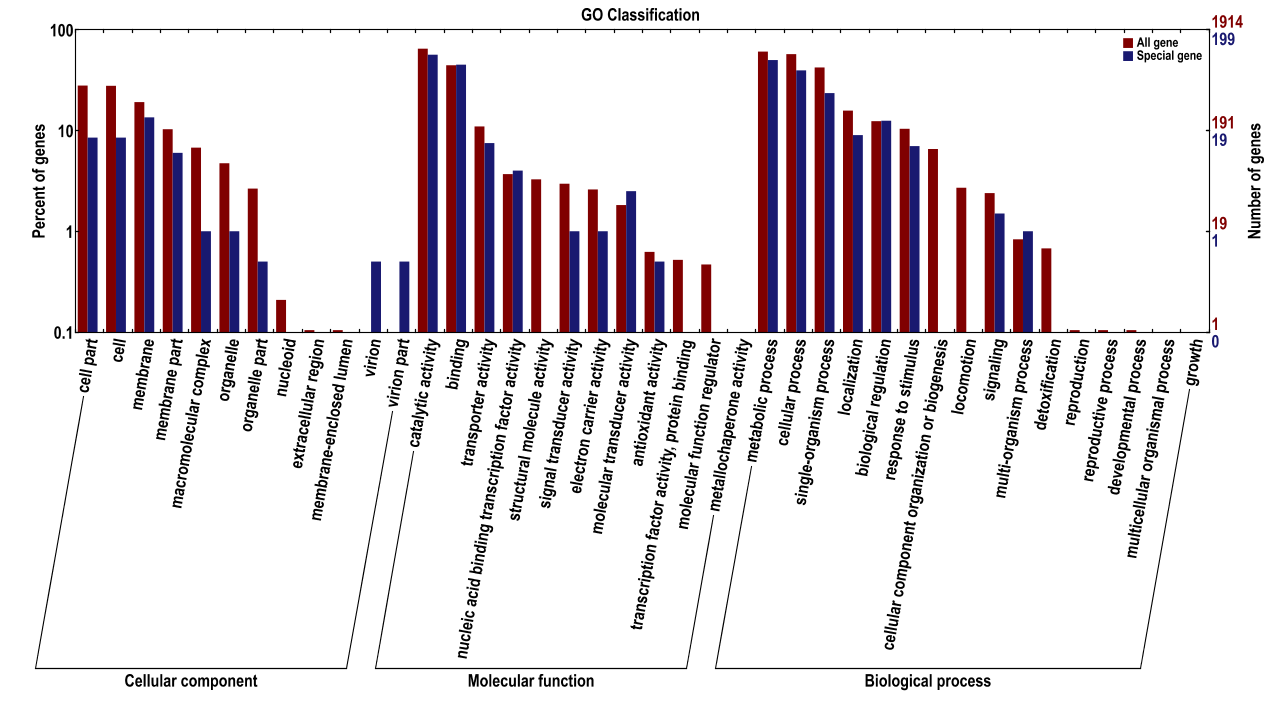
微生物基因功能注释
将预测到的非冗余基因与Nr、GO、COG和KEGG数据库比对,获得基因功能注释信息,了解环境中微生物潜在的生物学功能。
CAZy 数据库功能注释
CAZy是碳水化合物酶相关的专业数据库,内容包括能催化碳水化合物降解、修饰、以及生物合成的相关酶系家族。
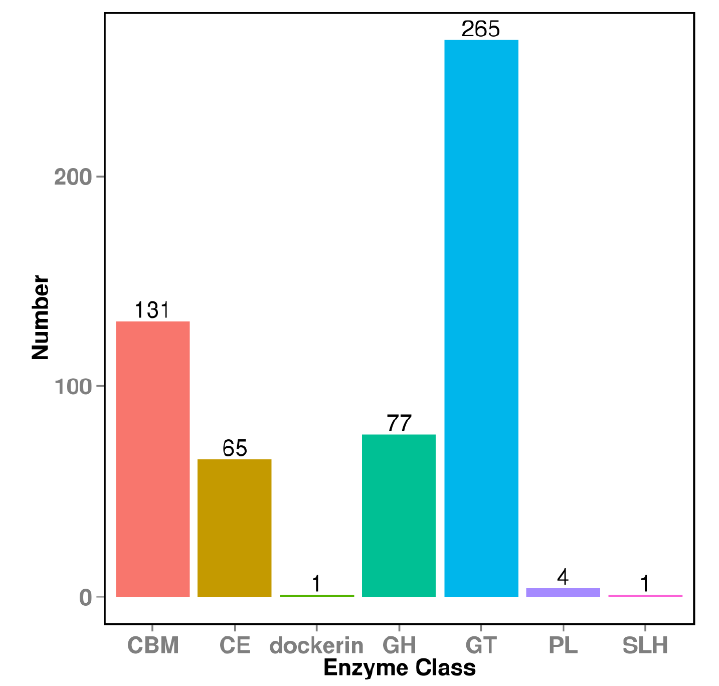
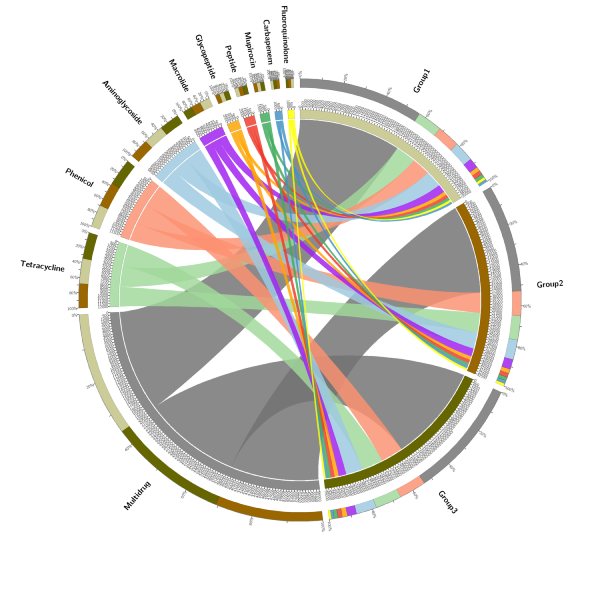
CARD功能基因组成分析
在注释时,使用CARD数据库中的工具软件rgi将非冗余基因的蛋白序列分别数据库进行比对,找出比对上的数据库中对应序列,并得到对应的抗性基因和抗性相关信息。
差异基因功能富集分析
通过比较不同环境下微生物基因的丰度,寻找不同环境下存在的差异基因并对这些差异基因做功能富集分析,研究微生物对不同环境的响应机制。
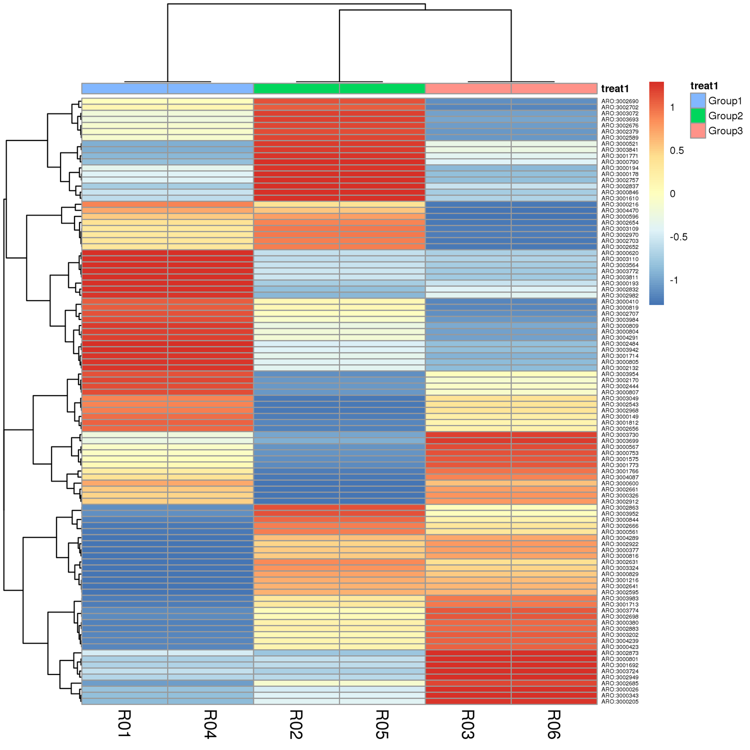
功能基因组间差异
使用Welch’s t-test进行组间差异参数检验;如果超过两组,使用ANOVA分析进行组间差异参数检验。通过参数检验获得的不同功能基因水平的丰度热图
案例一
Prevalence of antibiotic resistance genes and bacterial pathogens along the soil-mangrove root continuum
发表时间:2021
合作单位:中山大学
发表期刊: Journal of Hazardous Materials
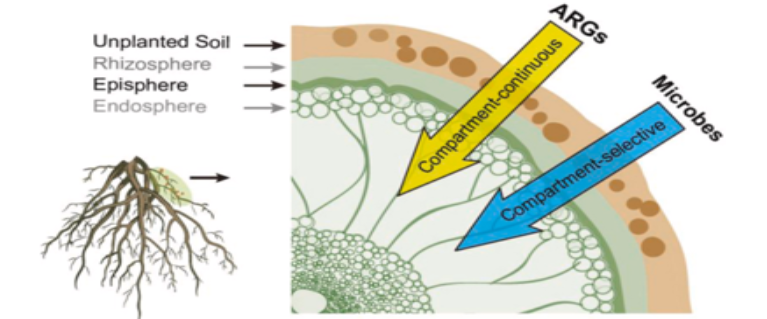
研究背景:植物的根往往被土壤细菌所侵染,这些细菌被认为是抗生素抗性基因(ARGs)的主要接纳体。ARGs可以在这些微生物和病原体之间转移,但这些ARGs和病原体在多大程度上从土壤传播到植物的机制还不清楚。本研究使用扩增子和宏基因组测序研究了红树林幼树土壤4个与根系相关的空间,揭示病原菌可能沿土壤-根系连续体传播的途径。
测序策略: 16S V3+V4 Illumina Hiseq 2500 PE250 + 宏基因组 Illumina PE150
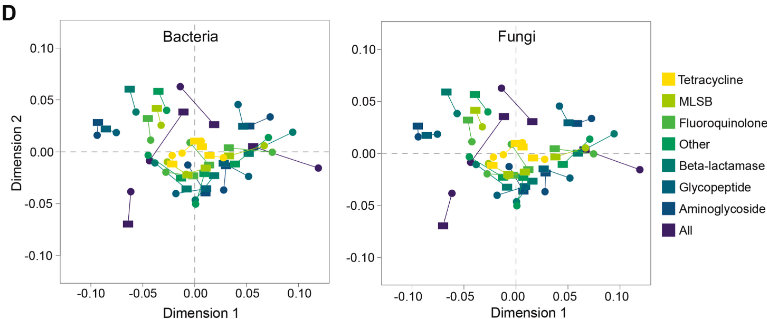
研究结论: 91.4%的ARGs在4个与根系相关的空间共享,与微生物区隔选择动力学不同,导入以一种连续的方式散布在土壤-根系连续体上。这种传播与潜在的根系相关的细菌和真菌微生物群无关,但可能是由多种可移动遗传元素促进的。创伤弧菌、致病性大肠埃希菌和肺炎克雷伯菌作为多药耐药病原菌,在4个隔室中均占主导地位,表明抗生素病原菌可能沿土壤-根系连续体传播 。
参考文献: Wang C, Hu R, Strong PJ, et al. Prevalence of antibiotic resistance genes and bacterial pathogens along the soil-mangrove root continuum. J Hazard Mater. 2021
案例二
Synergistic interactions of Desulfovibrio and Petrimonas for sulfate-reduction coupling polycyclic aromatic hydrocarbon degradation
发表时间:2021
合作单位:广东省微生物研究所
发表期刊: Journal of Hazardous Materials
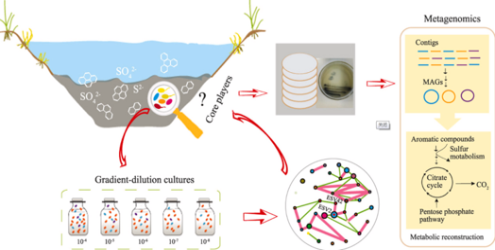
研究背景:随着工业化和城市化进程的推进,多环芳烃类污染物在水体沉积物中频繁检出,给水生态安全和人类健康造成极大威胁。本研究通过结合梯度稀释培养法、传统的分离培养技术以及宏基因组这这下学分析等多种方法手段,深入解析了黑臭河流沉积物中硫酸盐呼吸耦合多环芳烃降解的核心功能微生物组。
测序策略:宏基因组 Illumina PE150
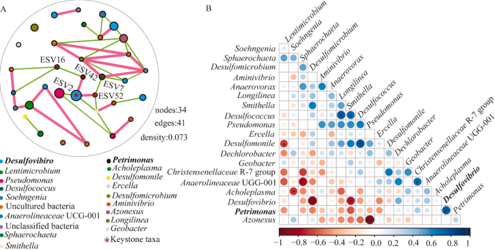
研究结论:梯度稀释培养法在显著降低沉积物中微生物群落复杂同时,并未削弱菌群的多环芳烃降解功能。Desulfovibrio和Petrimonas是硫酸盐呼吸耦合多环芳烃降解的核心功能微生物。这两种微生物通过潜在的协同作用促进多环芳烃降解转化。其中,Desulfovibrio主要负责通过羧化途径将多环芳烃降解至六氢-2-萘甲酰,而Petrimonas则利用其相对完整的三羧酸循环途径和磷酸戊糖途径实现前者多环芳烃中间代谢产物的进一步降解转化。
参考文献: Qian Y, Xu M, Deng T, et al. Synergistic interactions of Desulfovibrio and Petrimonas for sulfate-reduction coupling polycyclic aromatic hydrocarbon degradation. J Hazard Mater. 2021
宏基因组将基因组DNA随机打断成若干条小片段,然后在片段两端加通用引物进行PCR扩增测序,将得到的reads进行组装后进行基因预测得到基因序列,众多基因构成环境中微生物的基因集
微生物多样性主要针对核糖体小亚基基因序列进行测序(16s rDNA或者18s rDNA),该基因既存在高度保守的区域还包括高变区,通过特异性引物对某一段高变区(如V3区)或某几段高变区(如V3-V4区)进行扩增测序,然后与数据库比对,可特异性识别细菌种类。总的来说,微生物多样性主要告诉我们环境里有什么微生物,而宏基因组主要告诉我们环境里的微生物能做什么。
微生物多样性和宏基因组都会分析环境中物种的种类,但是物种的丰度在二者之间存在一定的差异,第一种原因是微生物多样性是基于核糖体小亚基基因序列进行物种注释和丰度统计,但是在不同物种中核糖体基因的拷贝数不一样,这会带来一些误差;第二种原因是二者在建库测序过程中会进行PCR反应,在PCR这个过程中也会存在一些误差。
- Nature Communications同款宏转录组技术详解,原来高分研究这么做!宏转录组学(Metatranscriptomics)作为一项前沿技术,用于探索不同生态系统中微生物群落在特定时 […]阅读更多
- 哥本哈根大学揭示膳食纤维其在特定病原体感染背景下的复杂性《Microbiome》2026年1月26日,哥本哈根大学Andrew R. Williams 团队在《Microbiome》期刊上发 […]阅读更多







 京公网安备 11011302003368号
京公网安备 11011302003368号