Small RNA测序
小RNA简介
Small RNA(sRNA)在细胞内具有多种重要的调节作用,主要通过miRNA与靶mRNA特异性结合调控靶mRNA的表达,从而参与许多生命过程,如细胞增殖、 细胞凋亡、脂肪代谢和细胞分化等。Small RNA(sRNA)测序是利用illumina测序平台检测已知和未知miRNA的表达水平及其表达差异,结合同一样本的转录组测序数据进行联合分析,可以同时分析miRNA及其靶基因的表达情况,为研究RNA的功能及调控机制提供有力的工具。
服务产品
miRNA鉴定
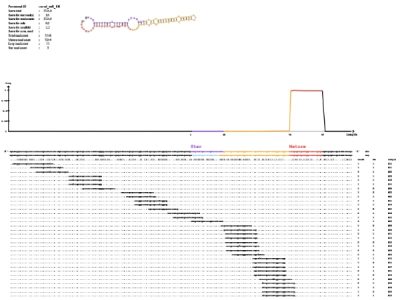
miRNA转录起始位点多位于基因间隔区、内含子以及编码序列的反向互补序列上,其前体具有标志性的发夹结构,成熟体的形成是由Dicer/DCL酶的剪切实现的。对于已知miRNA的鉴定,我们将比对上参考基因组的reads序列与已知miRNA数据库miRBase(v22)中的成熟miRNA序列进行比对。针对miRNA的生物特征,对于未鉴定到已知miRNA的序列,百迈客利用miRDeep2软件进行新miRNA的预测。
基因表达水平分析
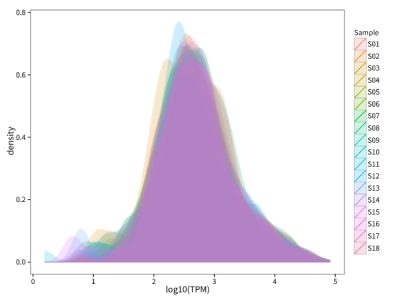
利用转录组数据检测基因表达具有较高的灵敏度。通过TPM密度图不仅可以反映单个样品miRNA的整体表达模式和离散程度,还可以直观的比较不同样品的整体基因表达水平差异。
差异表达miRNA聚类分析
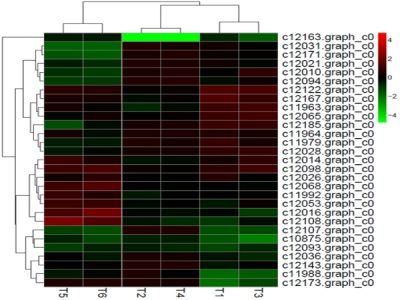
聚类分析用于判断差异基因在不同实验条件下的表达模式,可通过将表达模式相同或相近的基因聚集成类,从而识别未知基因的功能或已知基因的未知功能,同类基因可能具有相似的功能或共同参与同一代谢过程。对筛选出的差异表达miRNA做层次聚类分析,将具有相同或相似表达行为的miRNA进行聚类。
差异miRNA靶基因注释
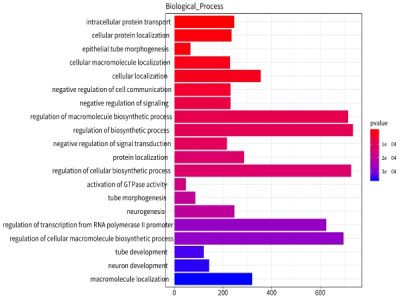
生物体内,在特定条件下某些基因对应的转录本会形成miRNA前体,失去编码功能,或者通过种子区域与目的基因进行互补结合,导致基因的表达受到抑制或降解,从而影响生物学表型。因此,针对miRNA的靶基因分别进行GO和KEGG注释及富集分析,有助于深入挖掘小RNA功能。
专业项目方案设计 深度解析数据
-
实验设计
-
RNA提取
-
建库测序
-
数据分析
-
售后答疑
成功案例
公司成立多年以来,拥有丰富的项目分析经验,累计完成1千多个样品的sRNA项目研究,物种涉及广泛,包括人鼠、动物、植物常见的组织部为及细胞样本。可根据项目需要选择方案,保障结果精准。
- 【成功案例】II型糖尿病患者肠道菌群改变与miR-122-5p表达的相关性研究2020年11月,百迈客和成都中医药大学附属医院检验科合作文章“Correlation between alt […]阅读更多
- 【项目文章】小RNA组学分析下的进化树研究揭示山茶花器官发育过程中的保守和家系特异的miRNA中文名: 小RNA组学分析下的进化树研究揭示山茶花器官发育过程中的保守和家系特异的miRNA 英文名:Phyl […]阅读更多
- 文献解读:-sRNA测序鉴定肺腺癌中具有潜在功能的microRNA–mRNA调节网络发表期刊:Front. Cell Dev. Biol. 影响因子:6.684 发表时间:2021.09 发表单 […]阅读更多
常见问题
答:sRNA测序产品可以对动植物、全血、细胞等组织或者核酸样本进行测序,分析要求需要有对应物种的参考基因组,具体的送样指导,请参考如下内容:

答:植物miRNA可与mRNA的编码区完全互补配对,并通过诱导mRNA降解而发挥抑制表达的作用。动物miRNA则可与mRNA的3’UTR区部分互补配对结合,进而抑制翻译的进行,一般的,miRNA与mRNA的配对区域位于miRNA的5’端的 2-8个碱基,称为种子区,只要种子区能与mRNA互补配对即可发挥作用,这也是一个miNRA能够调控数百条mRNA的原因。
我们根据已知miRNA和新预测的miRNA与对应物种的基因序列信息,植物用 TargetFinder软件进行靶基因预测;动物用miRanda和targetscan进行靶基因预测。
答:
RT-PCR对miRNA进行定量验证;
miRNA过表达;
荧光素酶法,过表达miRNA测定靶基因表达量;把靶基因连上一个荧光素酶,如果靶基因表达量下降,可检测到的荧光信号则减弱
降解组测序
答:
参照miRBase的命名规则:{物种名缩写}-miR-{编号};miR-前缀后面所跟着的数字,代表命名的顺序,比如,miR-124比miR-456发现得早。“miR-”代表成熟的miRNA、“mir-”代表pre-miRNA和pri-miRNA、“MIR”代表编码miRNA的基因。
miRNA几乎全是独一的编码顺序,但对于拥有一两个碱基不同的则会被标上字母以示,例如,miR-124a与miR-124b。 若成熟的miRNA相同,但pre-miRNA和pri-miRNA和编码他们的基因来自于不同的基因组,则使用数字来表示,例如,mir-194-1和mir-194-2表示两个pre-, pri-miRNA剪切后的成熟miRNA是完全相同的,但却是两个不同的来源。
前缀的三个字母代表了不同的种族来源,例如,hsa-miR-194代表miRNA来源于人类,oar-miR-124来源于绵羊。
对于形成pre-,pri-miRNA茎环的两端miRNA, 通常一端在数量上远远超过另一端。数量优势的一端往往称为guide strand,而另一端被称为passenger strand,通常被大量降解,用*号来表示,例如miR-124和miR-124*。
最新版中miRBase已经不再使用*来标记microRNA与其发夹前体互补配对位置的互补序列,而是使用“-3p”与“-5p”作为区分这两条序列的后缀替代旧的的命名法。(参考http://www.mirbase.org/help/nomenclature.shtml)
举例: aly-miR158b-3p, aly 代表这是Arabidopsis lyrata 物种的miRNA, -miR158b表示这是成熟的miRNA , 其中b表示还有一个miRNA序列与它相似,仅有一两个碱基不同。 -3p 表示它从前体的3’端剪切得到。
对新预测的miRNA 我们统一用 novel_miR_{数字编号} 来命名, 如 novel_miR_4974。
答:
在已知miRNA鉴定方面,我们将比对到参考基因组的reads与miRBase(v22)数据库中的已知miRNA的成熟序列及其上游2nt与下游5nt的范围进行比对,最多允许一个错配,这样鉴定到的reads被认为是鉴定到的已知miRNA。
miRNA转录起始位点多位于基因间隔区、内含子以及编码序列的反向互补序列上,其前体具有标志性的发夹结构,成熟体的形成是由Dicer/DCL酶的剪切实现的。针对miRNA的生物特征,对于未鉴定到已知miRNA的序列,百迈客利用miRDeep2软件进行新miRNA的预测。
我们利用miRDeep2软件包,通过reads比对到基因组上的位置信息得到可能的前体序列,基于reads在前体序列上的分布信息(基于miRNA产生特点,mature,star,loop)及前体结构能量信息(RNAfold randfold)采用贝叶斯模型经打分最终实现新miRNA的预测。miRDeep2主要用于动物miRNA的预测,通过参数的调整及打分系统的改变也可以对植物的miRNA进行预测。






 京公网安备 11011302003368号
京公网安备 11011302003368号