真核转录组学测序分析
产品介绍
二代真核转录组采用Illumina测序平台,对有参、无参真核生物特定细胞在某一功能状态下转录的所mRNA进行测序。在定量层面,有参可以对基因进行定量分析,无参只能对Unigene(优化的转录本)进行定量分析,并进行下游的差异基因分析和功能注释等;在结构层面,有参可进行可变剪切、SNP分析、基因结构优化、新基因预测。目前已广泛应用于基础研究、临床诊断、药物研发和分子育种等领域。
专业项目方案设计 深度解析数据
转录组可搭配任意其他产品进行多组学的分析,同时为了冲刺高分可选择大样本量方案进行设计。从选材到后续研究内容相关信息的挖掘,整个流程严谨进行,全程跟踪。
-
实验设计
-
RNA提取
-
建库测序
-
数据分析
-
售后答疑
百迈客云,个性化分析全面升级!
可视化操作、交互性基因深度挖掘,关注哪里“点”哪里。【基本操作】基因功能、基因名称、序列和ID的检索、关键基因功能及通路分析、WGCNA分析等基因功能挖掘。【个性化挖掘】个性化差异分析方案,个性化图表优化、共表达趋势分析、基因结构分析。
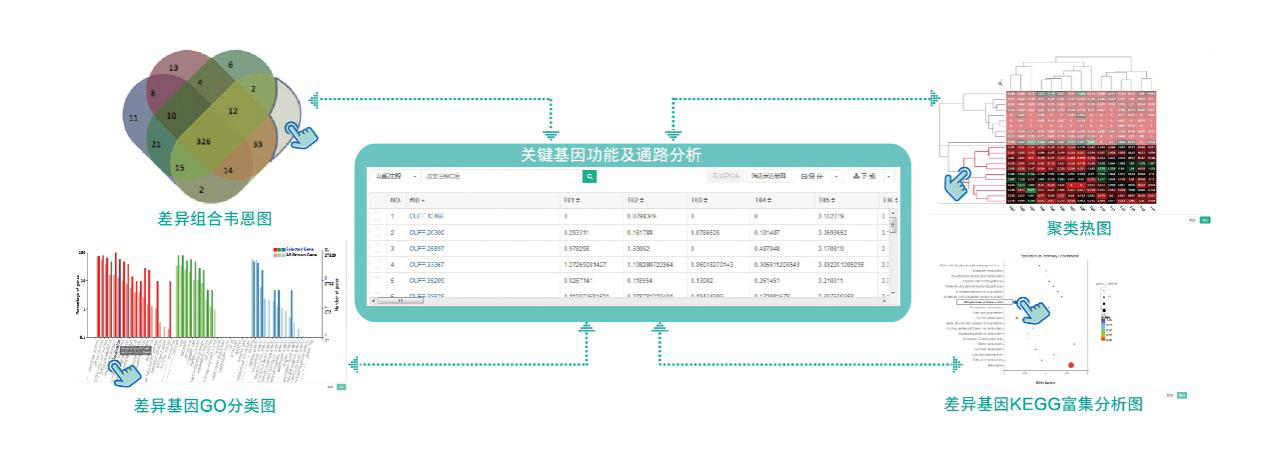
结果展示
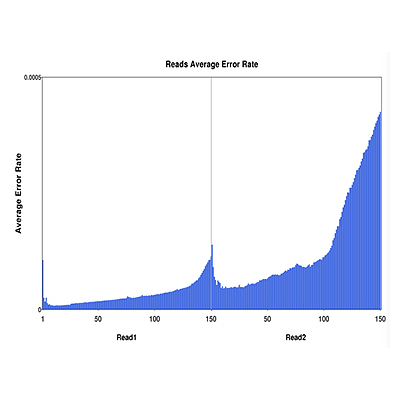
数据质控
为确保Reads有足够高的质量,将下机原始测序数据(raw reads)去掉含有带接头的、低质量的reads,得到clean reads,保证后续分析的准确性。测序因受测序仪本身、测序试剂、样品等因素影响,存在一定的错误率。碱基测序错误率分布图可以反映测序数据的质量。
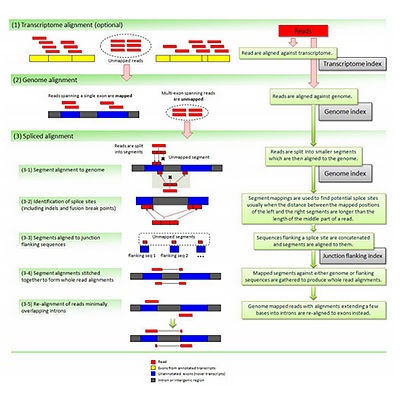
参考序列比对
将Clean Reads与参考基因组进行序列比对,获取在参考基因组或基因上的位置信息,定位区域分为Exon(外显子)、Intron(内含子)
和Intergenic(基因间区)。比对到参考基因组上的Reads称为Mapped Reads,Mapped Reads占Clean Reads的百分比,可以评估所选参考基因组组装是否能满足信息分析的需求。
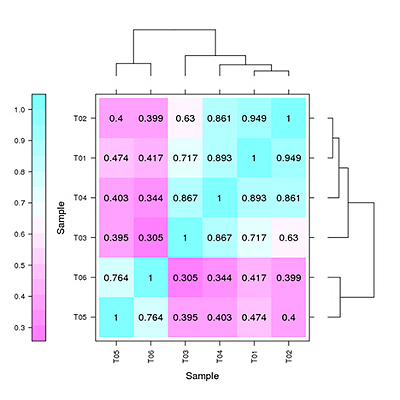
重复相关性评估
生物学重复的相关性不仅可以检验生物学实验操作的可重复性,还可以评估差异表达基因的可靠性和辅助异常样品的筛查。
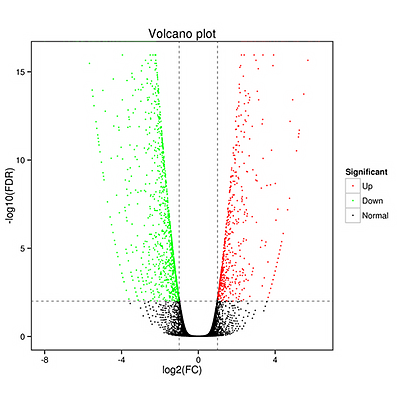
差异表达基因分析
差异表达基因以火山图、MA图、韦恩图、聚类热图、蛋白互作图等形式呈现,通过火山图(Volcano Plot)可以快速地查看基因在两个(组)样品中表达水平的差异,以及差异的统计学显著性。对于有生物学重复的样本,我们采用DEseq进行样品组间的差异表达分析,获得两个生物学条件之间的差异表达基因集;对于没有生物学重复的样本,使用EBseq进行差异分析。筛选差异基因标准一般为:Fold Change≥2,FDR<0.01。
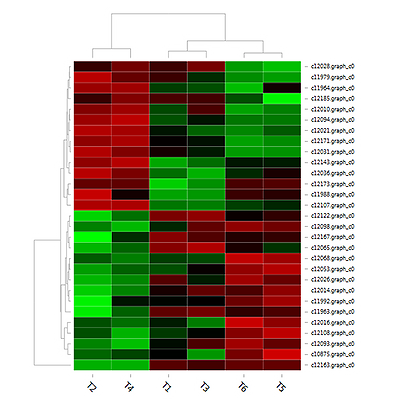
差异表达基因聚类分析
聚类分析用于判断差异基因在不同实验条件下的表达模式,可通过将表达模式相同或相近的基因聚集成类,从而识别未知基因的功能或已知基因的未知功能,同类基因可能具有相似的功能或共同参与同一代谢过程。
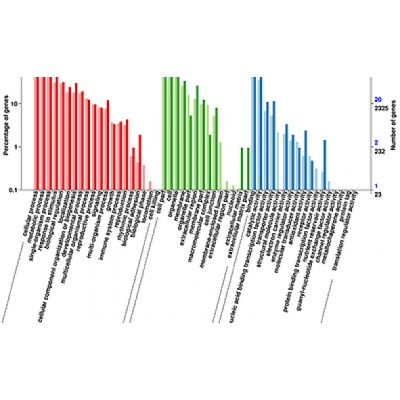
差异表达基因GO分类
差异表达基因GO注释分类统计图,直观的反映出在生物过程(biological process)、细胞组分(cellular component)
和分子功能(molecular function),所有基因和差异基因注释GO term的个数分布。可深入挖掘差异基因的功能及所在的信号通路,筛选关注差异基因注释情况。
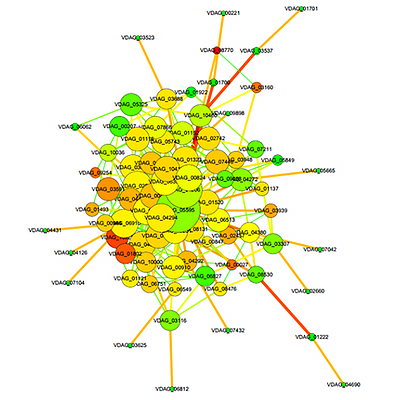
差异表达基因蛋白互作网络
STRING收录多个物种预测的和实验验证的蛋白质-蛋白质互作的数据库,包括直接的物理互作和间接的功能相关。结合差异表达分析结果和数据库收录的互作关系对,构建差异表达基因互作网络。
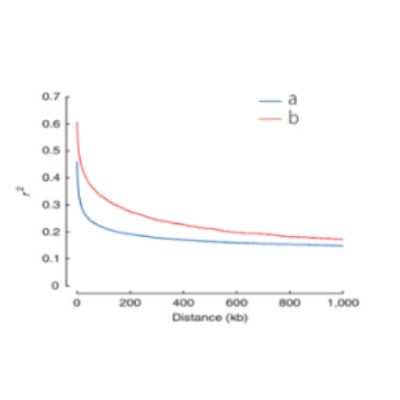
测序数据组装
对于无参转录组,过滤得到的高质量clean reads需通过Trinity软件进行组装得到转录本序列。转录本测序深度除了受测序数据量等影响,还与该转录本的表达丰度有关。为了使各样品中表达丰度较低的转录本组装得更完整,对于同物种的测序样品推荐合并组装可以间接增加测序深度,从而使转录结果更完整,同时也有利于后续的数据分析;而对于不同物种的样品,由于基因组间存在差异,推荐采用分别组装或分开分析。
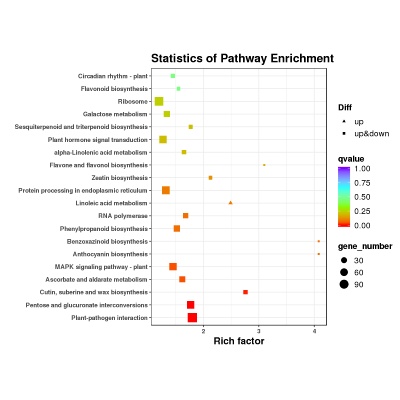
差异表达基因KEGG通路富集
差异表达基因的KEGG Pathway富集分析,系统分析基因产物在细胞中的代谢途径以及这些基因产物功能,把基因及表达信息作为一个整体的网络进行研究。利用富集因子(Enrichment Factor)分析Pathway的富集程度,并利用超几何检验方法计算富集显著性。
常见问题
答:针对每个样品,同一个基因的不同转录本会存在可变剪接,我们只是根据测序的实际数据对可变剪接进行预测,而不是进行验证;如果要判断是否存在相应的剪接方式,需要实验去验证。重复实验存在一定的差异,会导致可变剪接的不同。
答:我们分析流程中是将测序的Reads比对到参考基因组,然后进行拼接,其中一些reads比对到基因间区并且能拼接出完整的开放阅读框,拼接出来的位于基因间区的这些基因即为新基因。预测得到的新基因才会进行功能注释,所以注释结果与新基因的判断没有关系。
答:为了确保分析结果的准确性,老师通常会设置3个生物学重复,这样就可能出现生物学重复中某个样品相关性不好的情况,影响后续差异分析结果的准确性。通常可将该处理组中相关性不好的样品剔除,再进行差异分析。后期可通过RT-qPCR等试验手段弥补生物学重复的不足,不会影响文章的发表。
答:可从所有基因,差异基因及SNP三个方面进行数据挖掘。所有基因可通过功能注释信息,基因ID,基因名称,序列信息几个方面进行挖掘,同时还可以做表达基因集维恩图,WGCNA等分析。差异基因则可通过维恩图分析不同处理批次几个差异组合共同的差异基因;通常表达量变化趋势一致的基因,可能会有相似的功能,故可通过基因共表达趋势分析来进行差异基因的深入挖掘。SNP则可通过PCA分析,系统进化树,样品间差异SNP筛选及目标区域SNP查询等进行挖掘。以上这些分析均可在我公司云平台免费完成。
答: K number Count指相关的酶的数目,比如8(6)代表8个基因注释到这个通路,涉及到这个通路的6个酶,某两个基因(或多个)涉及到同一个酶。
答:Go富集我们使用的是Blast2GO R包;KEGG是我们根据fisher检验算法自己编写的程序。
KS<0.05,这个值和p-value的意义相同,是TopGO软件包中的一个检验方法。
Q-value<0.01,这个值是对p-value值的一个校正,和FDR概念相似,是fisher检验中的一个检验方法。
测序文章一般不用这两个指标,涉及到算法的文献中才有。
答:(1)用实验的方法: 针对5’端和3’端的序列来设计引物,通过PCR实验进行延长和扩增.
(2)生信办法: 将该基因与它的近源物种做同源,如果能找到同源基因,则将该区域的所有read比对到同源基因上,进而来确定中间部分的序列。
项目经验丰富,硕果累累
公司成立多年以来,拥有丰富的项目分析经验,据不完全统计,完成转录组项目10000+,完成样本数200000+;年处理样本数10000+;农学物种涉及粮食作物、果蔬、观赏植物、害虫、家禽牲畜、水产动植物等,医学物种涉及人、鼠,研究方向包括发育调控、环境适应、突变表现、遗传进化、疾病发生发展机制、耐药机制和药物的研发诊断等各种领域。可根据项目需要选择方案,保障结果精准。
成功案例
- 【成功案例】Insm1的SNAG 域调节胰腺内分泌细胞分化并抑制 β细胞转分化为δ细胞2021年5月,百迈客和暨南大学第一附属医院合作的文章“The SNAG Domain of Insm1 Re […]阅读更多







 京公网安备 11011302003368号
京公网安备 11011302003368号