ATAC-seq测序技术
获得潜在的活跃转录因子及其靶基因
产品介绍
ATAC-seq(Assay for Transposase-Accessible Chromatin with highthroughput sequencing),基于高通量测序的染色质转座酶可接近性实验。在核小体连接致密的地方,转座酶不能进入,而松散的区域,转座酶能够进入并切割下暴露的DNA区域,并同时连接上特异性的接头,有接头的DNA片段被分离出来,用于高通量测序。因此,ATAC-seq可以得是全基因度尺度上处于开放状态的染色质区域,并且通过分析染色质开放区域的motif,可以获得潜在的活跃转录因子及其靶基因。
标准服务流程
-
样品寄送
-
建库测序
-
数据分析
-
出具报告
-
售后答疑
测序实验技术流程
ATAC-seq主要实验流程首先是制备细胞悬液,然后裂解液裂解细胞获得细胞核,进一步使用Tn5转座酶酶切并纯化,最后回收DNA片段,对回收片段进行末端修复、3’端加A、连接测序接头,片段大小选择以及PCA扩增等步骤获得标准的高通量测序文库,使用Illumina测序平台进行测序。
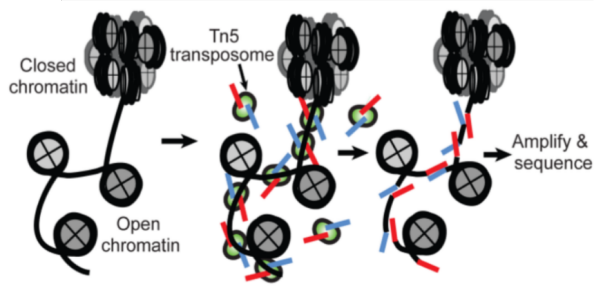
标准信息分析流程
原始测序Reads经过去接头和过滤低质量,获得Clean Reads。将Clean Reads与参考基因组序列进行比对,通过Reads在基因组上的比对位置信息,进行Peak calling,并进行motif预测、转录因子靶基因挖掘、差异Peak和Motif等分析。
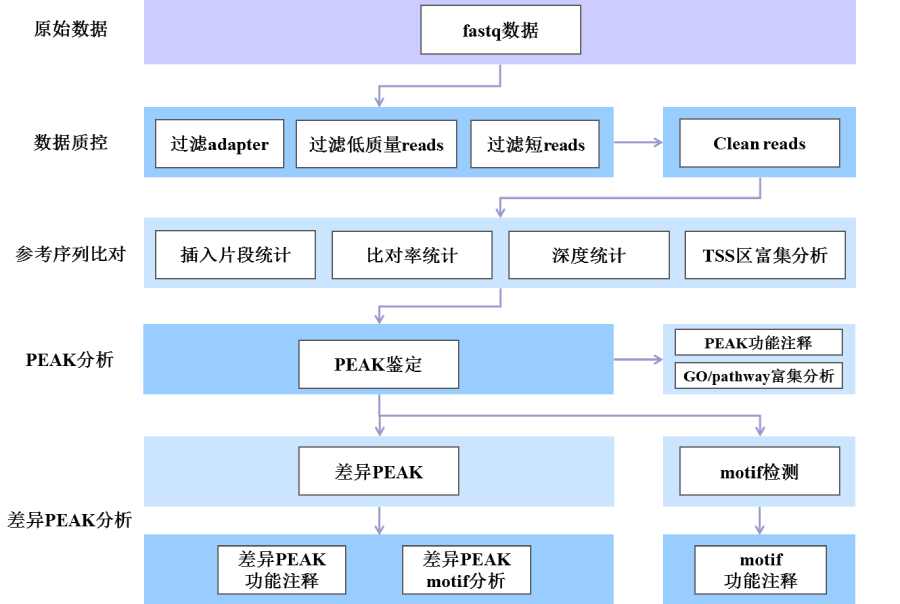
结果展示
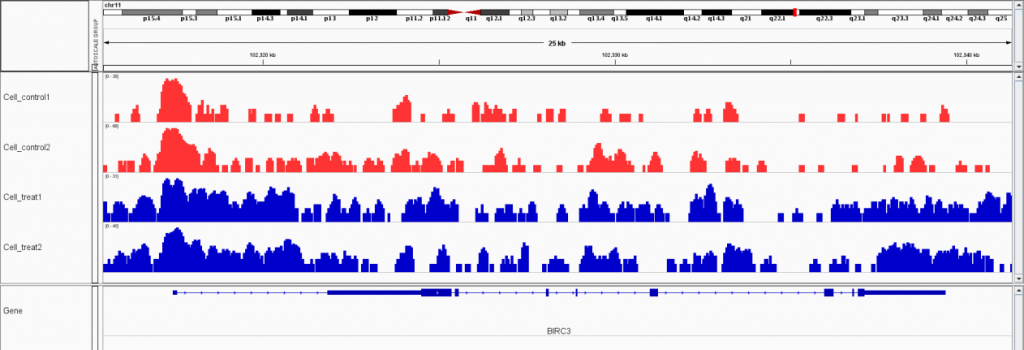
开放染色质区域可视化图
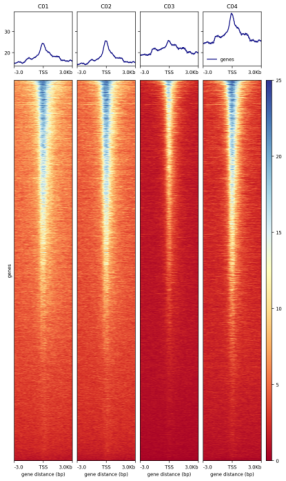
转录起始位点附近的开放染色质区域
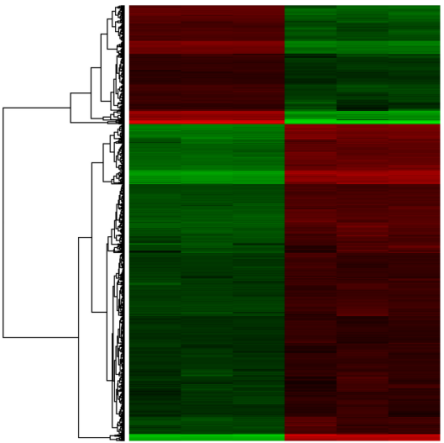
差异开放染色质区域聚类热图

开放染色质motif
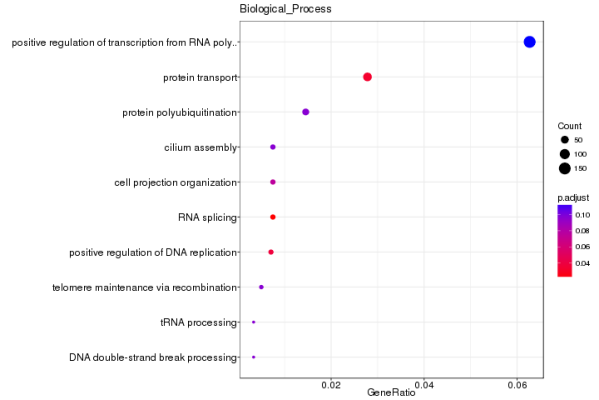
差异开放染色质区域功能基因富集分析
常见问题
ATAC-seq需要生物学重复吗?
需要的,推荐3-5个重复,目的是为了老师实验结果的准确性和可重复性。
到底motif是什么?
简单地讲, Motif 就是一段特定模式的DNA序列。可以理解为开放的染色质区域有着序列偏好性,而偏向的序列就是motif。
ATAC-seq和Chip-seq的区别?
Chip-seq是研究特定转录因子或者蛋白结合的区域,需要用抗体去免疫共沉淀相应的区域。而ATAC-seq无需抗体,可将所有转录因子或者蛋白可能结合的区域用Tn5转座酶切割下来进行测序。
相关文章
- 前沿|ATAC-seq专题-Genome Biology & Genome ResearchATAC-seq(Assay for Transposase – Accessible Chromatin w […]阅读更多
- 【文献精读】ATAC-seq+RNA-seq联手解读心肌梗死前后的心脏成纤维细胞的染色质可及性图谱ATAC-seq技术是继FAIRE-seq、MNase-seq、DNase-seq目前最火热的研究染色质开放性 […]阅读更多
- RNA-seq&ATAC-seq揭示TRIM28参与HIV-1潜伏感染机制【成功案例】2019年新年伊始,百迈客与中山大学中山医学院合作发表了RNA-seq & ATAC-seq多组学文章 […]阅读更多
- 【文献精读】HiC&ATAC-seq多组学应用又来了!揭示CTCF耗竭重新连接全基因组的染色质可及性Hi-C (High-through chromosome conformation capture) 是以整 […]阅读更多
- Hi-C+ATAC+Chip+RNA-seq多组学揭示胰腺癌转移过程中3D表观图谱胰腺癌是消化道常见恶性肿瘤之一,晚期极易出现转移,其中胰腺癌肝转移很常见。据柳叶刀杂志记载,胰腺癌确诊后的五年 […]阅读更多







 京公网安备 11011302003368号
京公网安备 11011302003368号