宏基因组(ONT)
产品介绍
ONT测序是新一代基于纳米孔的单分子实时电信号测序技术,其各平台的测序原理相同。DNA/RNA链在马达蛋白的带领下与镶嵌在生物膜上的纳米孔蛋白结合并解螺旋,在生物膜两侧电压差的作用下,DNA/RNA链以一定的速率通过纳米孔通道蛋白,由于DNA/RNA链上不同碱基化学性质存在差异,所以当单个碱基或DNA/RNA分子通过纳米孔通道时,会引起不同电学信号的变化。通过对这些信号进行检测及对应,即可计算获得相应碱基的类型,完成序列的实时测定。
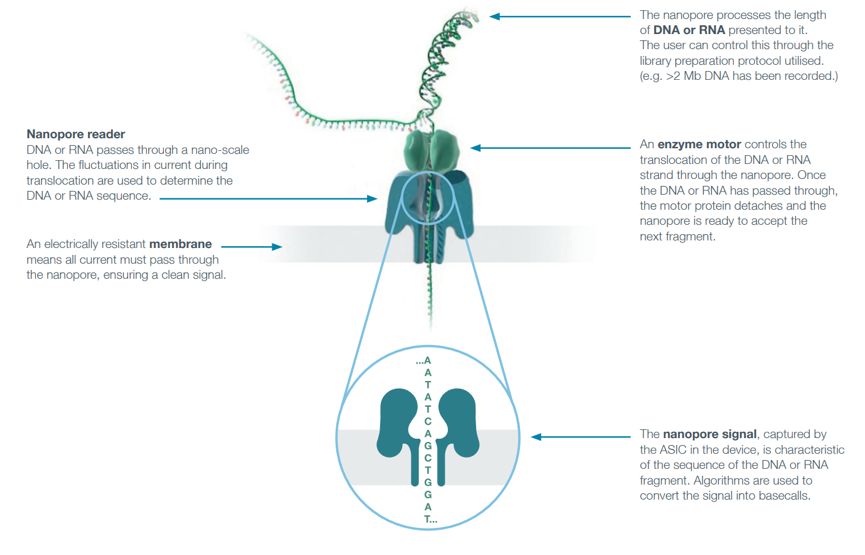
Nanopore的碱基判读是依据其电流信号而产生,因此其判定过程比较复杂。目前Nanopore根据电流的大小及电流大小的变化情况,通过“递归神经网络(Recurrent Neural Network)”的复杂算法对碱基进行判读。
宏基因组是对环境样本中(土壤、粪便、肠道、水体等)微生物总DNA进行测序。关注环境微生物的种群结构、进化分析、功能基因;解析微生物功能与环境之间的关系;挖掘和研究特定功能基因及代谢通路。纳米孔测序技术能够对环境样本进行√确的微生物组分析。并且已经在宏基因组领域广泛应用。
基于Nanopore测序平台的宏基因组测序,通过生物信息分析获得环境中的基因功能信息和物种组成信息。通过基因功能的角度分析环境样品所具有的功能多样性;通过物种的角度分析分析环境样品中含有的物种的多样性。
产品优势
宏基因组binning
含义:分箱、聚类,指从微生物群体序列中将不同个体的序列(reads或contigs等)分离开来的过程。
contig binning在常规宏基因组基础上,进一步将拼接的contig进行cluster聚类,可实现单菌的组装以及宏基因组关联分析(MWAS)分析。
基于宏基因组数据的contig binning分析,可基于宏基因组组装结果,将组成相似以及丰度分布模式一致的contig划分到同一物种,并进一步进行单菌的草图组装。从而可在基于单菌组装结果的基础上进行菌株水平的基因和功能注释、比较基因组分析、进化分析等。

实验流程
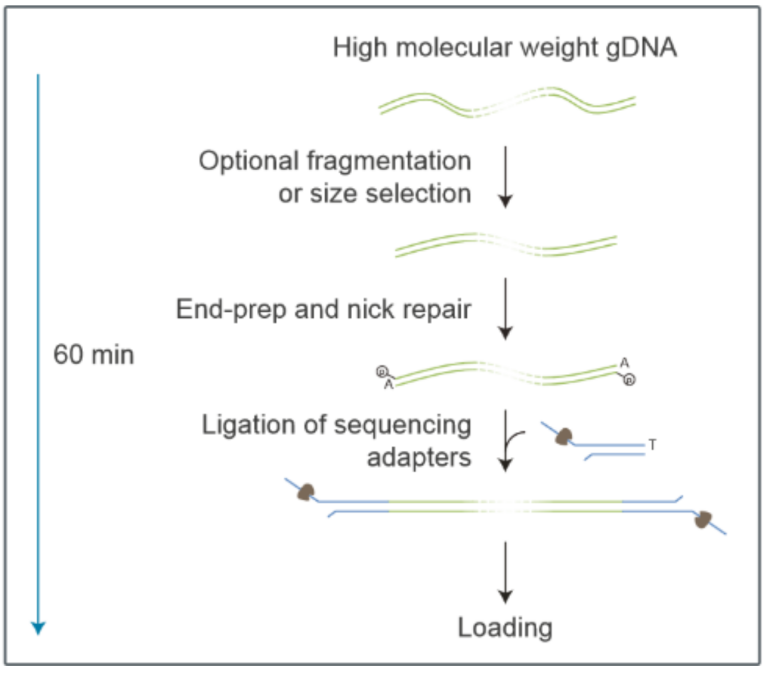
按照Oxford Nanopore Technologies(ONT)公司提供的标准protocol执行,包括样品质量检测、文库构建、文库质量检测和文库测序等流程,主要包括如下步骤:
1、提取高质量基因组DNA,利用Nanodrop、Qubit和0.35%琼脂糖凝胶电泳进行纯度、浓度和完整性质检;
2、BluePippin全自动核酸回收系统回收大片段DNA;
3、文库构建(SQK-LSK109连接试剂盒);
1) DNA损伤修复和末端修复,磁珠纯化;
2) 接头连接,磁珠纯化;
3) Qubit文库定量。
4、上机测序。
对测序得到的原始reads进行质量控制并过滤得到Clean reads,用于后续生物信息学的分析。对Clean reads进行拼接组装、预测编码基因,并对编码基因进行通用数据库和专用数据库的功能注释;同时,对Clean reads进行分类学分析,统计样品物种组成和丰度信息。
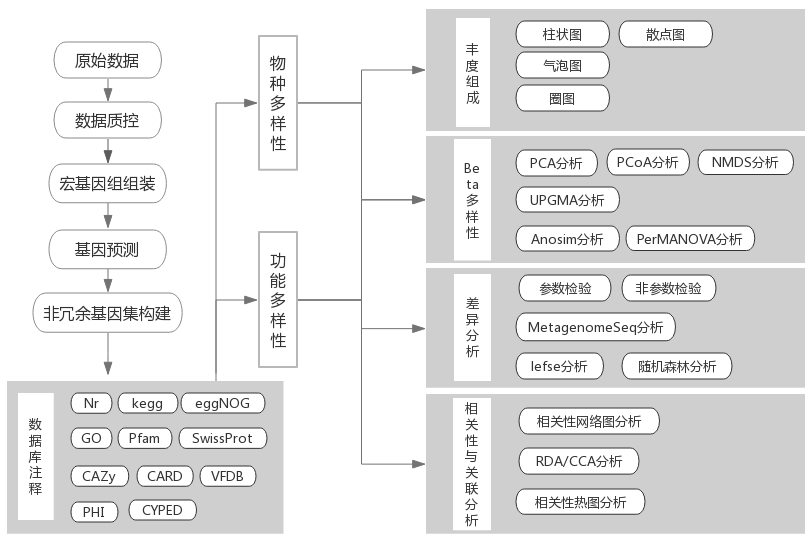
生信分析流程
结果展示
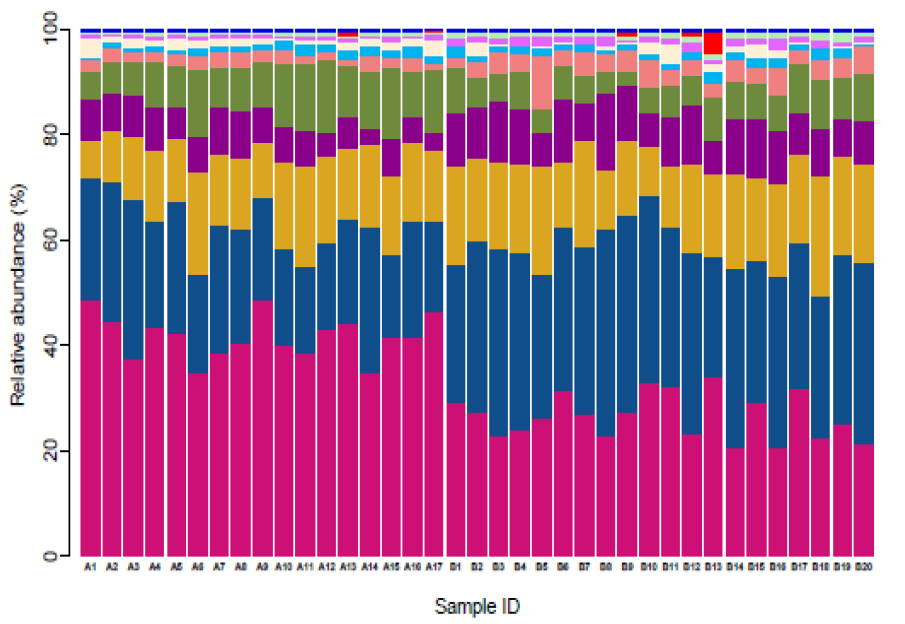
物种注释
使用diamond软件将质控后的高质量测序数据比对到 nr数据库,得到样品的物种组成和相对丰度信息。
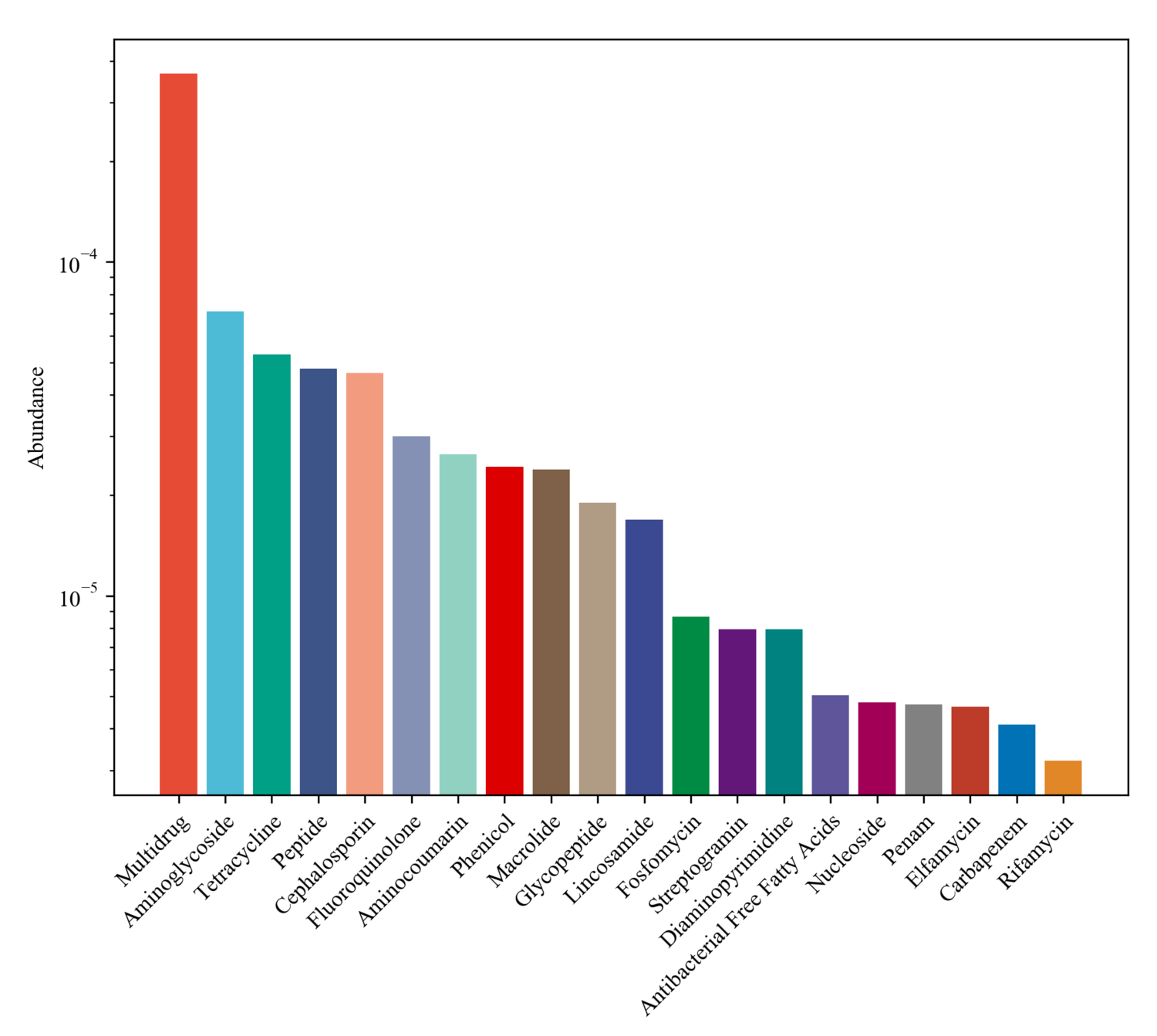
CARD功能基因组成分析
CARD是一个持续更新的抗生素抗性相关信息的数据库。 使用CARD数据库中的工具软件rgi将非冗余基因的蛋白序列分别数据库进行比对,找出比对上的数据库中对应序列,并得到对应的抗性基因和抗性相关信息。
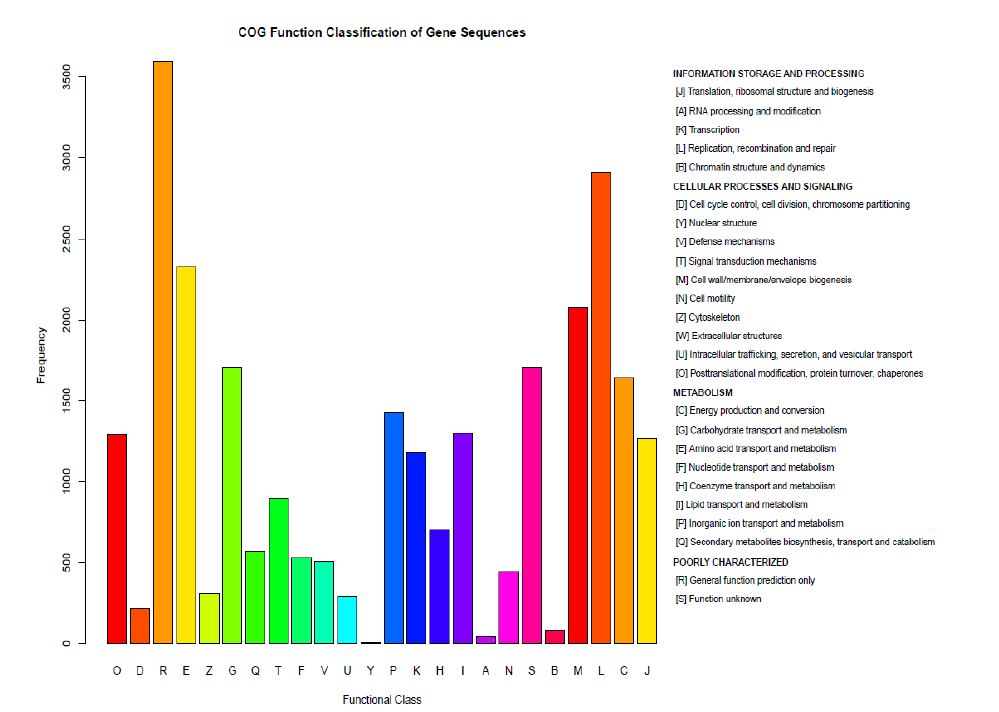
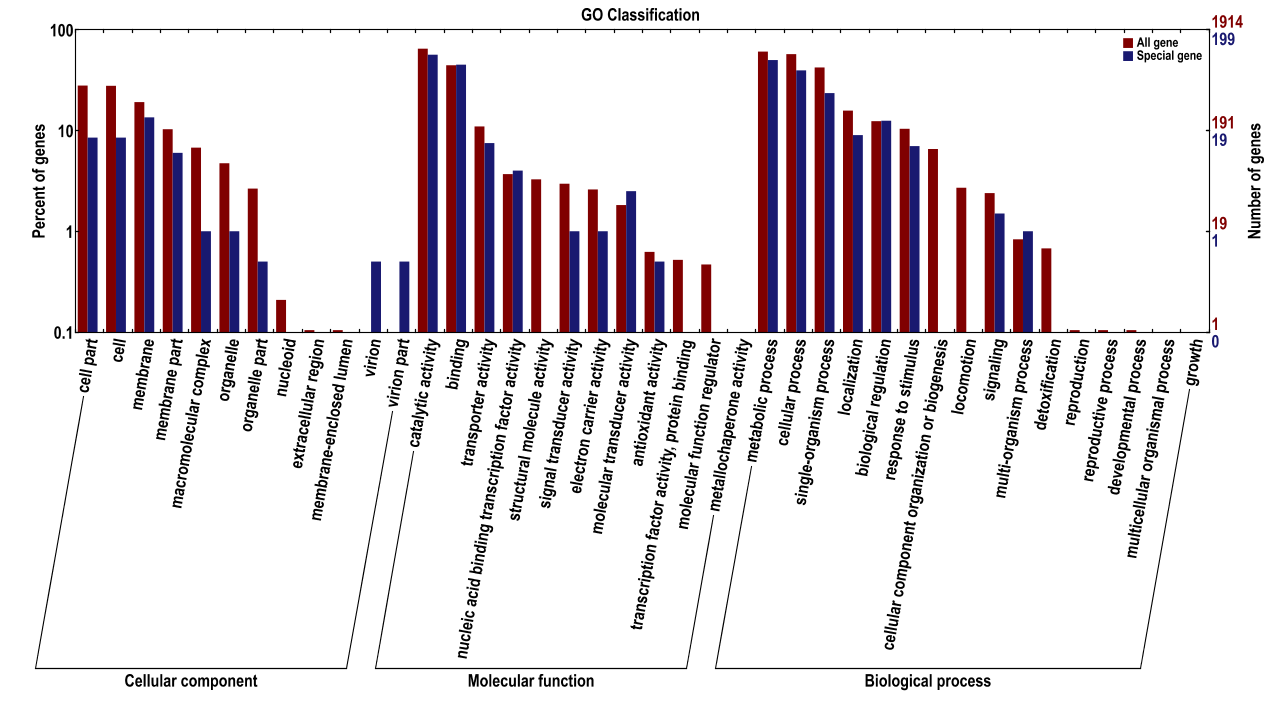
微生物基因功能注释
将预测到的非冗余基因与Nr、GO、COG和KEGG数据库比对,获得基因功能注释信息,了解环境中微生物潜在的生物学功能。
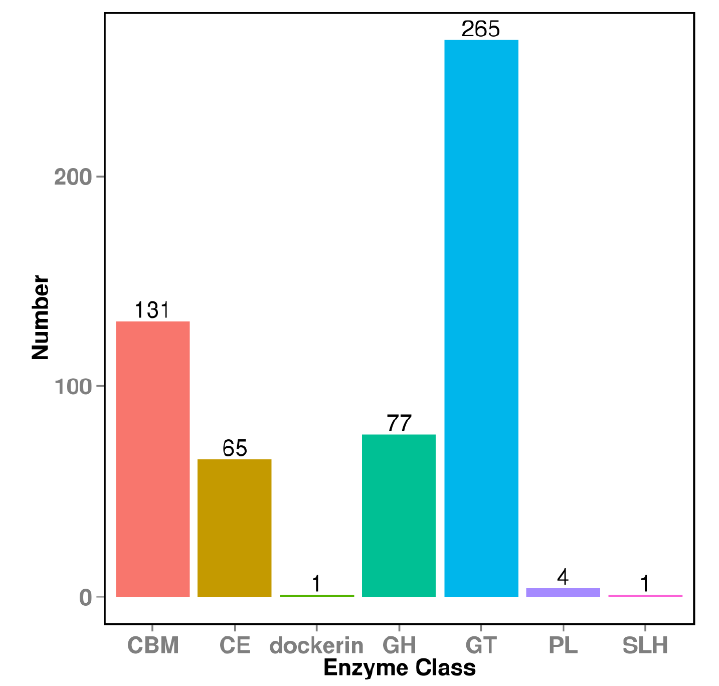
CAZy 数据库功能注释
CAZy是碳水化合物酶相关的专业数据库,内容包括能催化碳水化合物降解、修饰、以及生物合成的相关酶系家族。
差异基因功能富集分析
通过比较不同环境下微生物基因的丰度,寻找不同环境下存在的差异基因并对这些差异基因做功能富集分析,研究微生物对不同环境的响应机制。
常见问题
ONT宏基因组和二代宏基因组相比有何优势?
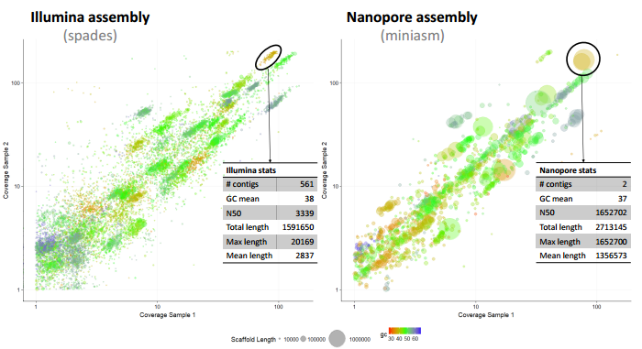
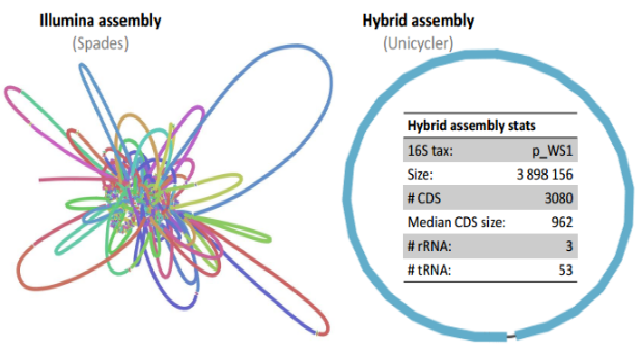
平均读长>10Kbp,有的长可达2M左右;直接跨越重复序列、高度多态性区域等特殊区域;无需PCR扩增;避免PCR扩增引入的错误;携带甲基化信息;Contig N50平均可达100Kbp,较二代平均提升100倍;可以绕过培养,通过Binning等方法,获得部分中高丰度的细菌、古菌、病毒等的基因组完整图或近完整图
宏基因组binning是什么意思,有何作用?
binning含义是分箱、聚类,指从微生物群体序列中将 不同个体的序列(reads或contigs等)分离开来的过 程。简单来说就是把宏基因组数据中来自同一菌株的 序列聚到一起,得到一个菌株的基因组。可获得环境样本中,不可分离培养的单菌基因 组草图,结合三代测序,甚至可获得单菌完成图,基 于基因结构和功能注释,可进一步进行数据挖掘






 京公网安备 11011302003368号
京公网安备 11011302003368号