Hi-C辅助基因组组装
每个基因组都需要一个Hi-C
产品介绍
Hi-C辅助基因组组装是指在已有二代或三代或光学图谱辅助组装的的Draft genome序列和已知染色体数目的前提下,
利用Hi-C测序数据将Draft genome序列进行染色体群组的划分,
并确定各序列在染色体上的顺序和方向,使基因组组装组装水平提升到染色体水平的技术。

不借助遗传图将基因组挂载到染色体水平。每一个基因组都需要一个Hi-C
HI-C特点
分析内容

服务流程
-
样品寄送
-
建库测序
-
数据分析
-
出具报告
-
售后答疑
产品优势
百迈客公司成立于2009年,深耕基因组测序领域8年之久,
是目前国内为数不多的拥有最强基因组测序平台的公司,
其拥有二代测序仪,三代测序仪(RS II 和sequel),光学图谱Irys,Hi-C组装技术等。
拥有自主研发的领先的基因组测序和分析技术,目前已经获得30多项发明专利,超过150多项核心软件著作权。
什么是Hi-C技术?
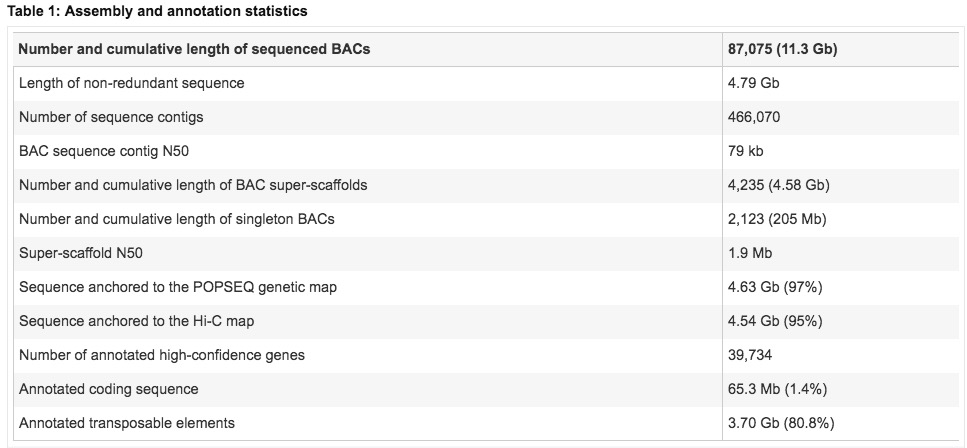
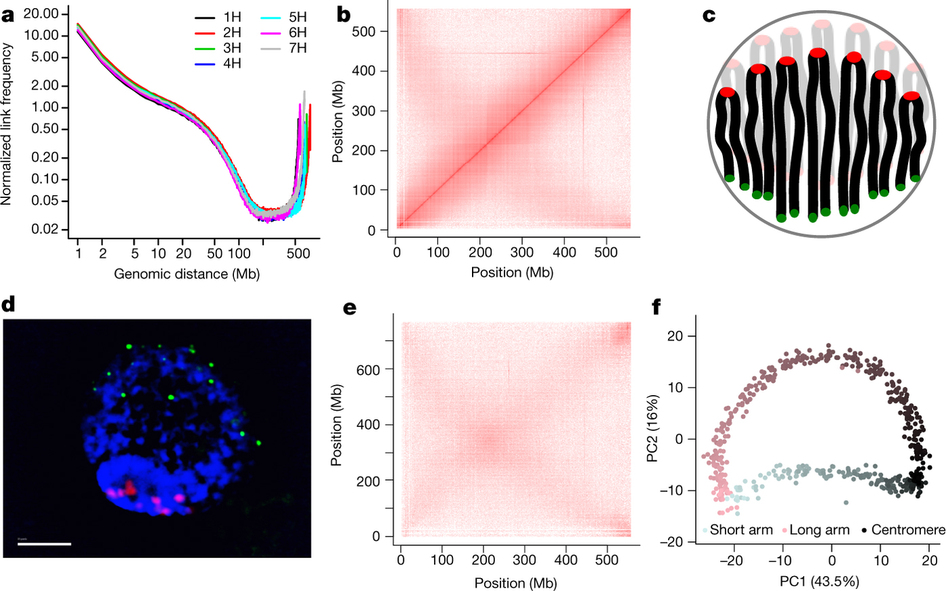
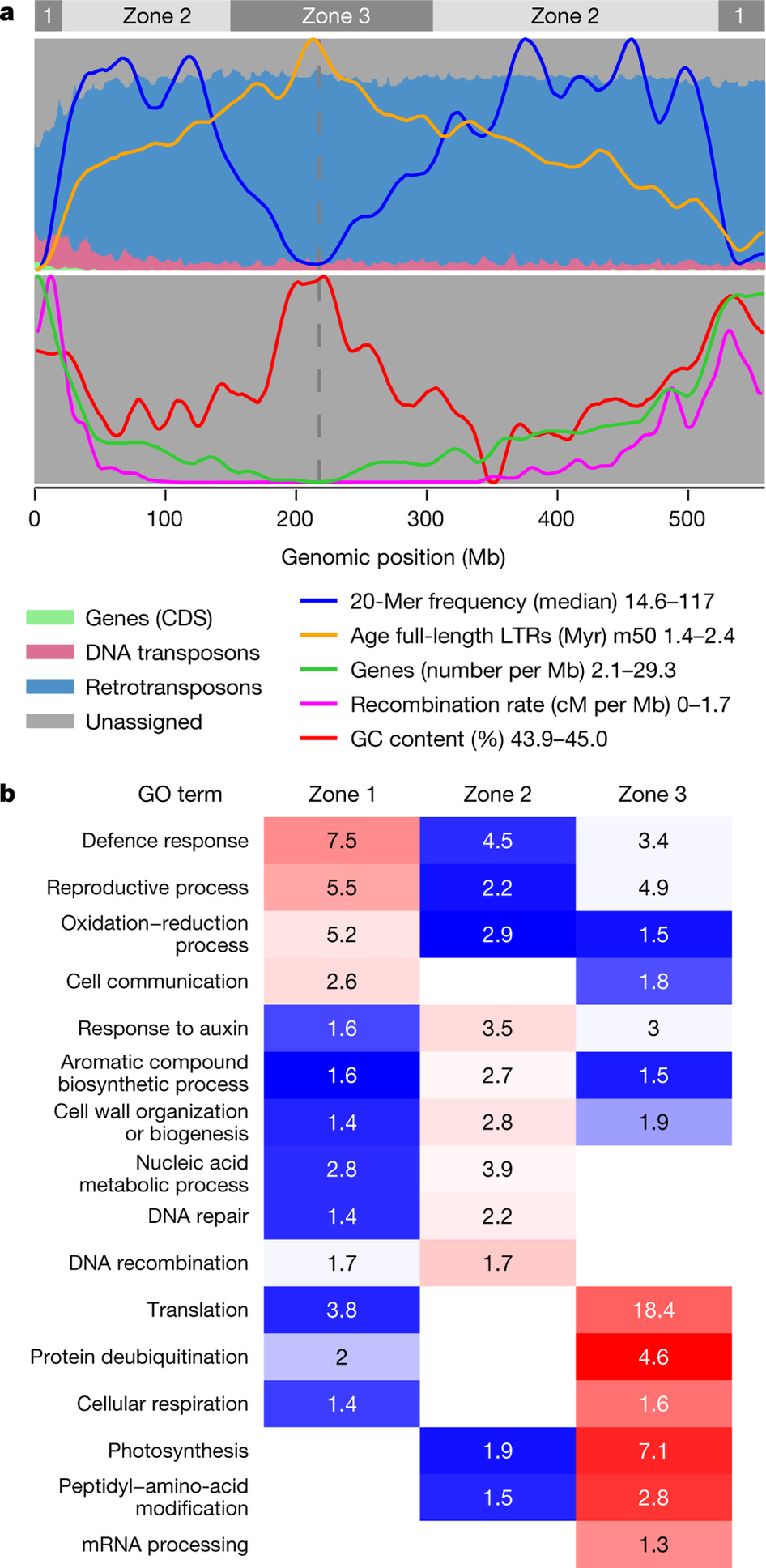
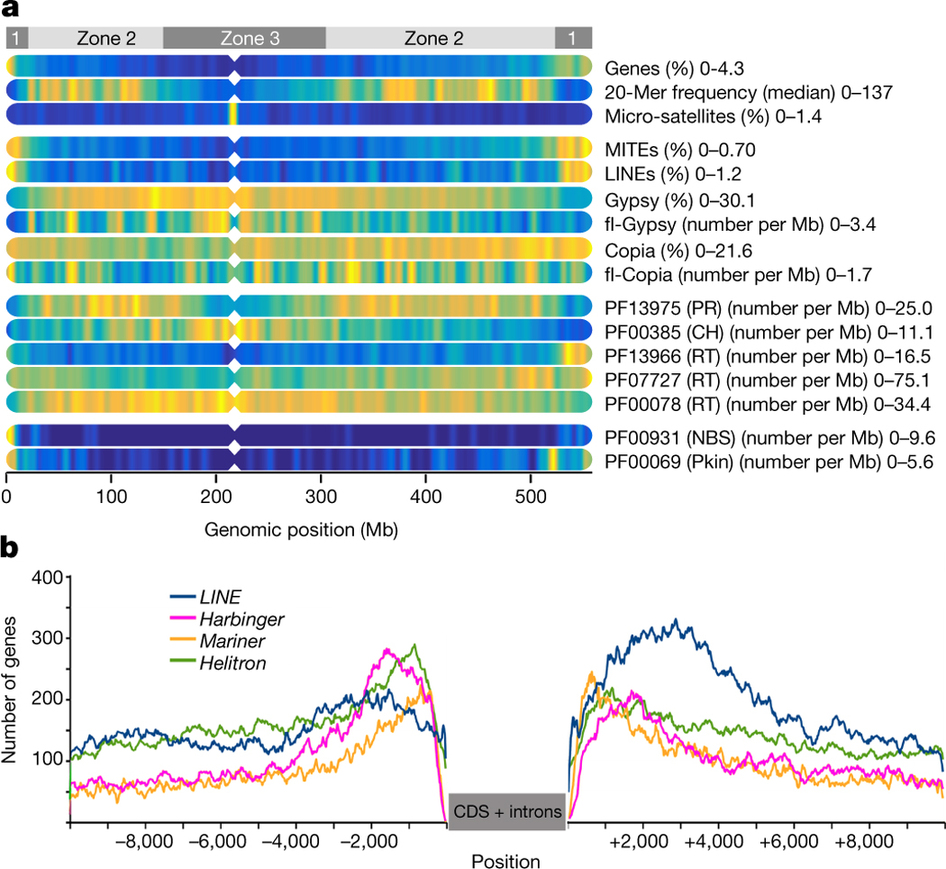
Hi-C技术是染色质构象捕获技术( Chromosome conformation capture )与高通量测序( High-throughput sequencing )结合衍生的一种技术。主要是利用全基因组范围内整个染色质DNA在空间位置上的关系,对染色质内全部DNA相互作用模式进行捕获,结合生物信息学方法,来获得染色体水平的基因组序列并得到染色质三维结构信息。此外还可以并与Chip-seq、转录组数据联合分析,从基因调控网络和表观遗传网络来阐述生物体性状形成的相关机制。
Hi-C在辅助基因组组装时有什么作用?
- Hi-C最主要的作用是将零散的基因组序列锚定到染色体上(这一点类似遗传图谱);
- 还可以对组装的基因组进行纠错处理;
- 在某种程度上进一步提升Contig N50.
Hi-C技术与遗传图谱的差异?
- Hi-C应用单个个体就可以完成染色体构建;
- Hi-C挂载染色体效率高达90%以上;
- 但Hi-C技术不能进行QTL定位。
Hi-C技术的样品要求?
- 植物样品要选择活体幼苗;
- 动物样品先用全血;
- 其他样品请咨询百迈客。






 京公网安备 11011302003368号
京公网安备 11011302003368号