背景介绍
DNA在染色体上是高度折叠的,DNA与DNA片段之间不可避免的形成了高强度的交互作用。2002年提出的3C技术,用于测定染色体特定位点与位点之间的交互作用,随后发展出4C、5C 技术,分别用于测定染色体上一点到多点和多点与多点之间的交互作用。Job Dekker在2009年开发出Hi-C 技术,实现了全基因组范围内染色体片段间相互作用的捕获。Hi-C 除了用于辅助基因组组装、对已组装的基因组进行纠错,还可以揭示基因组的三维结构特征,包括从隔室(A/B Compartments)到拓扑相关结构域(TAD),最后到染色体环(loop),目前,已在人、果蝇、酵母、拟南芥、水稻和棉花等物种成功构建基因组三维结构,并完成了对不同样本基因组三维结构的比较分析。
1、Hi-C三维构象研究内容
1)绘制全基因组的Hi-C map、Cis/Trans和IDEs等分析;
2)Compartment A/B 鉴定与分析,包括与ChIP-seq、WGBS和RNAseq等联合分析;
3)TAD鉴定与分析,包括与ChIP-seq、WGBS和RNAseq等联合分析;
4)绘制全基因组LOOP模型图,需DNase-seq或ATAC-seq,并与ChIP-seq和RNAseq联合分析;
5)多样本三维结构差异分析,包括Compartment A/B、TAD和Loop差异鉴定与分析;
6)基因/重复序列互作分析,包括与RNAseq联合分析。
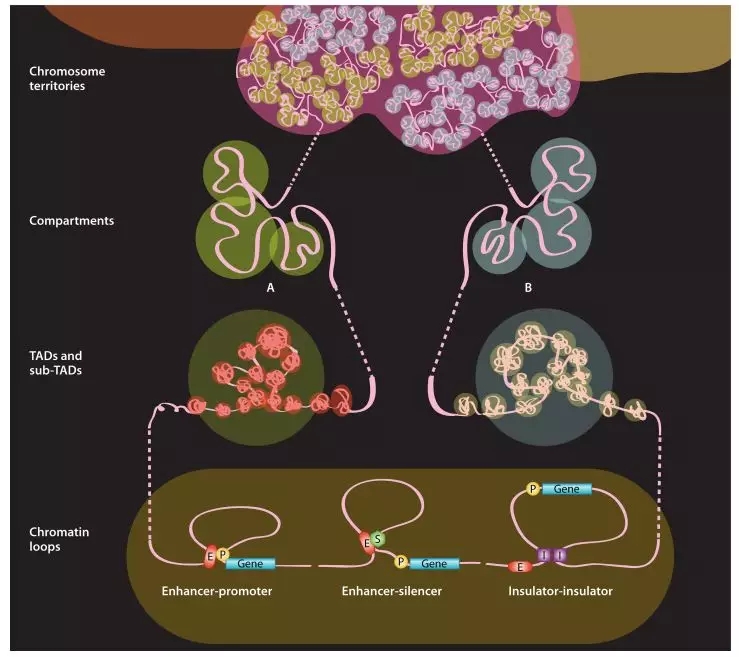
图1 染色体不同层级三维构象图
(Fraser J, et al, 2015)
2、染色体三维结构重点研究内容
A/B compartments
高等动植物基因组可以人为划分为两个compartments,称作A/B compartments,通过计算PCA,第一主成分中正值表示A compartment,负值表示B compartment。A/B compartments在基因组中一般呈间隔分布,A compartments呈松散染色质状态,基因密度高,属于转录活跃区域,而B compartments则呈压缩染色质状态,基因密度低,属于转录抑制区域。通过对多个样本A/B compartments 的分布进行对比,在全基因组水平找出A/B compartments保守的区域以及发生A/B compartments转换的区域,结合RNA-Seq对发生A/B compartments转换区域内的基因的表达量进行统计,有助于解释不同样本间的差异与染色质活性状态之间的关系。如图2:
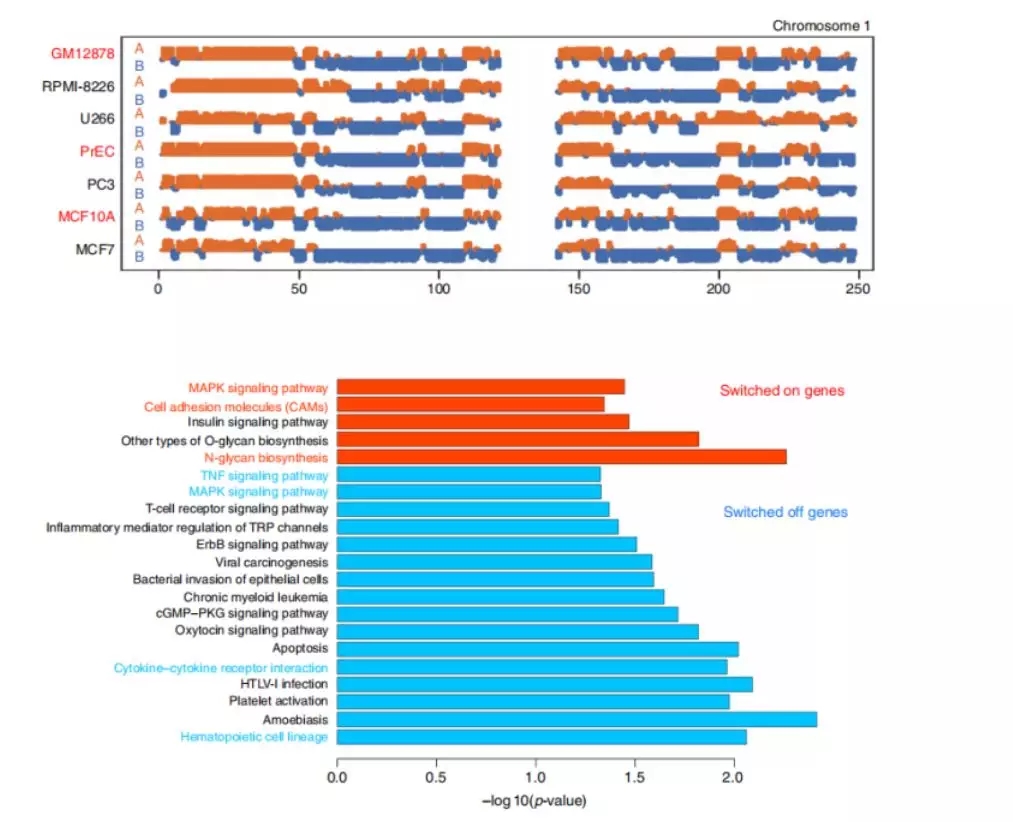
图2 人正常细胞和癌症细胞A/B compartments
(Wu P, et al, 2017)
例如在人的多发性骨髓瘤文章中,作者利用正常B细胞(GM12878)和两种亚型的骨髓瘤细胞(近二倍体U266和近三倍体RPMI-8226)进行Hi-C、WGS和RNA-seq测序,结果发现,相比于GM12878,U266和RPMI-8226癌症细胞中6%的区域均存在B转换到A,1%的区域均存在A转换到B,结合RNA-seq数据,发现B转换到A区域内的基因变成转录活跃状态,而A转换到B区域内的基因变成转录抑制状态,KEGG富集通路发现这些基因与多发性骨髓瘤相关,主要涉及MAPK信号转导途径、TNF信号转导途径、细胞因子和细胞因子受体相互作用途径等,进一步在染色体2q11.2-q12.142处发现一个细胞因子基因簇,几种白细胞介素IL1R1,IL1R2,IL18R1和细胞因子MAP4K4下调表达,这些基因在后续研究中重点关注。
TAD
TAD(拓扑关联结构域)是一个高度自关联的连续区域,通过明显的边界与相邻区域分离开来,形成一个独立的调控单元,内部的基因拥有共同的调控元件,存在协同表达特征。TAD边界通常具有大量的绝缘子蛋白和黏连蛋白(植物中TAD边界一般缺少绝缘蛋白,边界不明显),对于维持TAD结构及稳定性具有重要作用,不但可以指导染色体折叠成高级结构,还可以正确指导远距离转录调控,该边界发生变化会导致基因调控变得紊乱。TAD边界还与组蛋白修饰、甲基化修饰等密切相关,通常与转录活性相关的表观遗传标记富集在TAD边界,而与转录失活相关的表观遗传标记远离TAD边界。通过对多个样本TAD进行对比,在全基因组水平找出发生变化的TAD(重点关注TAD边界),结合ChIP-seq、WGBS等分析TAD的变化是否与表观遗传修饰相关,进一步利用RNA-Seq对相关基因的表达量进行统计,有助于解释不同样本间空间结构的差异与表观遗传修饰及转录调控之间的关系。如图3:
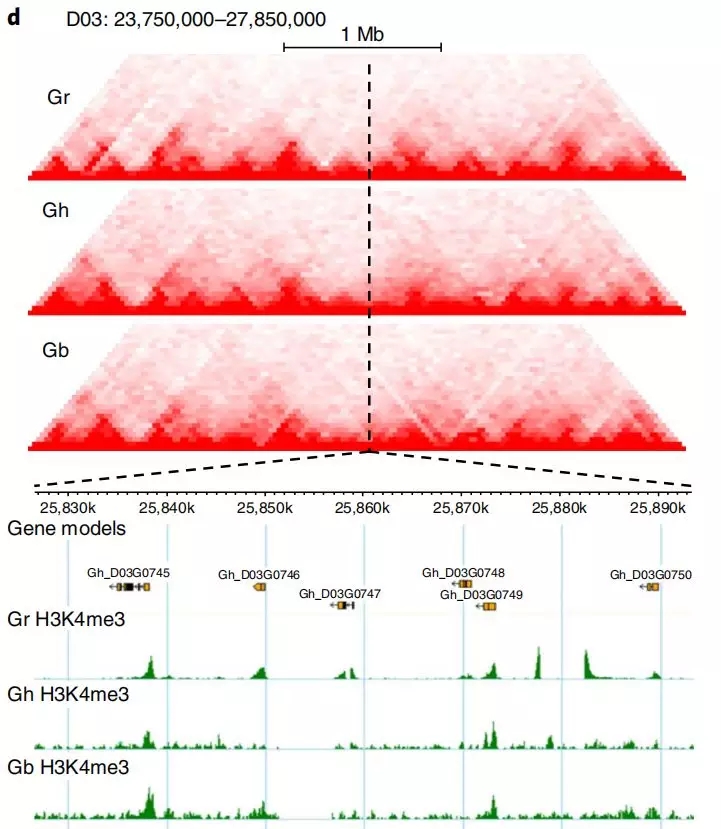
图3 不同种棉花TAD边界及表观遗传修饰
(Wang M, et al, 2018)
例如,在棉花的三维基因组进化文章中,作者对二倍体雷蒙德氏棉、二倍体亚洲棉、四倍体陆地棉和四倍体海岛棉构建了三维基因组图谱,与雷蒙德氏棉D03染色体相比,陆地棉和海岛棉对应的D亚基因组中发生了染色质重排,虚线对应的TAD结构发生了变化,雷蒙德氏棉中该TAD结构完整,存在明显的边界,陆地棉中该边界发生了左移,而海岛棉中该TAD趋向于消失。因而通过鉴定TAD发生变化区域的基因,可以研究多倍化过程中基因表达调控的改变(研究多倍体的老师们,这可是一个很好的套路文章哦)。同时结合ChIP-seq数据对该TAD边界进行分析发现,雷蒙德氏棉富含丰富的H3K4me3(活性染色质标记),而陆地棉和海岛棉中H3K4me3显著减少,说明在棉花的多倍化与表观遗传修饰存在密切关联。
Loop
Loop(染色质环)将线性距离很远的位点拉至空间距离很近,在Hi-C图谱中表现为峰值位点(peak loci),该peak位点通常一端为启动子,另一端为增强子,Loop将启动子和增强子拉至空间很近的距离,从而调控基因的表达,存在这种loop(启动子-增强子环)相关基因的表达量将几倍甚至几十倍的增加。loop通常与DNAase-seq或者ATAC-seq结合使用(特异性识别启动子),从而鉴定启动子-增强子环。通过对多个样本Loop进行对比,在全基因组水平找出发生变化的Loop,结RNA-Seq对相关基因的表达量进行统计,有助于解释不同样本间loop及基因转录调控之间的关系。如图4:
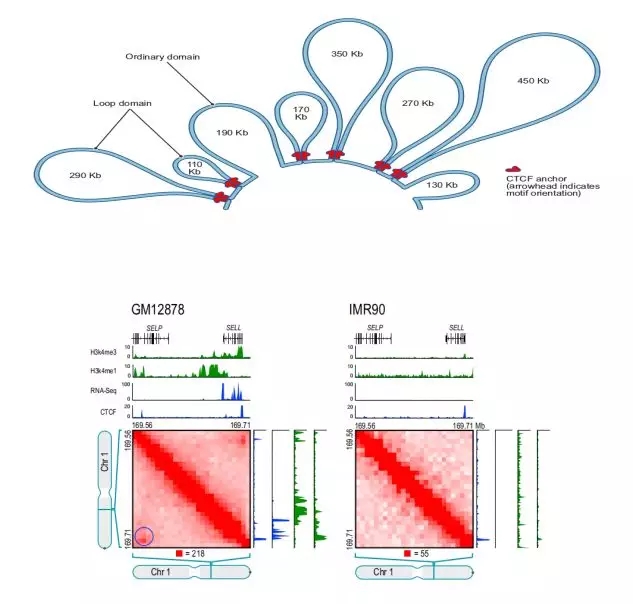
图4 人不同细胞loop(Rao S S.P. , et al, 2014)
例如,在人的loop文章中,作者在GM12878细胞系发现9448个loop,其中2854个loop与已知的启动子-增强子作用相关,基因的启动子存在loop比不存在loop时的表达量显著增加。GM12878细胞系中存在一个Loop,这个Loop连接了SELL启动子和一个远端增强子SELP,基因开启转录,表达量增加,而IMR90细胞系中没有这个Loop,基因不表达。
3、百迈客Hi-C研究优势
百迈客自2016年初以来,利用Hi-C技术进行染色体水平的基因组组装及染色体三维构象的研究,成功开发出六碱基、四碱基酶切方案,组装、互作轻松拿下。在植物Hi-C领域,更是迈进了一大步,在同行还只能处理植物活体样本的时候,我们已经可以轻松“驾驭”离体枝条,感觉一大波离体枝条在向小编招手~
迄今为止,保持着近100%的建库成功率,积累了大量植物、动物(哺乳动物、昆虫、水生动物等)、微生物等的项目经验。植物一次建库成功率在97%以上,很多物种对应文库的Valid Interaction Pairs(%)值在80%以上,最高达92.61%;动物样品文库的Valid Interaction Pairs(%)平均值在60%,最高达86.68%;真菌样品文库的Valid Interaction Pairs(%)平均值在72%,最高达84.6%。2018,百迈客Hi-C服务平台将继续为您的科学研究保驾护航!如果您Hi-C测序技术感兴趣,欢迎点击下方按钮联系我们,我们将免费为您设计文章思路方案。
参考文献:
1.Fraser J, Williamson L, Bickmore W A. et al. An Overview of Genome Organization and How We Got There: from FISH to Hi-C. Microbiology and Molecular Biology Reviews, 2015.
2.Wu P, Li T, Li R, et al.3D genome of multiple myeloma reveals spatial genome disorganization associated with copy number variations. Nature communication, 2017.
3.Wang M, Wang P, Lin M, et al.Evolutionary dynamics of 3D genome architecture following polyploidization in cotton. Nature plants, 2018.
4.Rao S S.P. , Huntley M H., Durand N C., et al.A 3D Map of the Human Genome at Kilobase Resolution Reveals Principles of Chromatin Looping. Cell, 2014.5









 京公网安备 11011302003368号
京公网安备 11011302003368号