作物数据库作为农业科研领域的核心基础设施,通过系统化整合遗传、基因组、表型等多维数据形成资源库,为农业科研、遗传资源保护、分子育种加速、表型 – 基因型关联分析等研究提供了关键支撑。本文聚焦水稻、小麦、玉米、大豆四大作物,梳理其核心遗传与基因组数据库,旨在为农业科研工作者提供全面的数据资源参考。
水稻数据库
☆ 国家水稻数据中心:提供水稻优异品种、突变体、分子标记、基因、QTL、品种、系谱等信息,以及水稻期刊、软件服务等资源。

☆ 水稻基因组注释项目数据库RGAP与水稻注释计划RAP:旨在提供水稻日本晴基因组的序列和注释数据。该网站提供基因组序列和12条水稻染色体的注释,基因的分布,或者根据基因名称查找基因的功能和序列、下载数据或使用blast搜索同源基因。

☆ 水稻基因组变异及其功能注释的综合数据库RiceVarMap v2.0:提供了来自4,726个水稻种质的测序数据中的17,397,026个基因组变异(包括14,541,446个SNP和2,855,580个小 INDEL)的精选信息,高质量和完整的基因型数据,变异的全面注释,表型数据和 GWAS 结果,对功能基因定位,标记开发的客户非常友好。
☆ 水稻功能基因组育种数据库RFGB(v2.0):该数据库整合了3024份水稻种质资源的18M SNP、2.3M InDel、IRGSP所有基因单倍型和12种表型数据,具有基因检索、基因组变异可视化浏览、BLAST等功能,将表型数据和单倍型数据联合分析,为挖掘基因有利单倍型提供数据支持。
☆ Oryzabase是一个综合性水稻科学数据库,包含大量水稻品系库存信息,突变体信息,染色体图谱,基因字典,水稻科学基础知识。OryzaGenome是野生Oryza物种的基因组数据库,为水稻研究提供了比较和进化的组学方法。
☆ 水稻基因索引数据库RGI:在这个数据库里,亚洲稻的每一个基因都能轻松找到同源或相近的基因,以及追踪其演变历史。
☆水稻功能基因组表达数据库RiceGE
☆ 水稻基因表达库RED:是一个基因表达谱存储库,对跨越整个水稻生长阶段的组织进行 RNA-Seq 数据分析,涵盖各种生物和非生物处理。
☆ 水稻表达谱数据库RiceXPro:针对水稻植物在自然田间条件下的整个生长过程、用各种植物激素处理的水稻幼苗以及通过激光显微切割(LMD) 分离的特定细胞类型/组织,使用微阵列技术进行水稻转录组分析,旨在表征水稻中所有预测基因的表达谱。
☆ 水稻多元表观基因组数据的数据库RiceENCODE:该数据库收集了包括ChIP-seq,ATAC-seq, MNase-seq, FAIRE-seq, BS-seq, RNA-seq, ncRNA-seq, Hi-C和ChIA-PET 等水稻高通量数据,可查询不同品种、不同组织、不同染色体区段交互信息,两两基因之间交互网络等。
小麦数据库
☆ 小麦基因组信息数据库:由国际小麦基因组测序联盟维护(IWGSC),可查找参考基因组序列、基因,BLAST,批量注释等多个功能,可帮助科研工作者加速开发改良品种,为小麦基础应用科学提供支持。
☆ 小麦组学数据在线平台WheatOmic:实现了对多套麦族物种基因组、转录组、变异组、突变体库外显子组、表观修饰组等大数据的可视化,并具有拟南芥/水稻-小麦同源基因鉴定、基因共表达网络构建、蛋白互作、转录因子结合位点分析等多个分析工具。

☆ 小麦族同源基因数据库TriticeaeGeneTribe:利用小麦族物种中已发表的基因组,对不同参考基因组间同源基因鉴定,构建了小麦族同源基因数据库。同时,数据库具有基因功能查询,GO富集分析,同源基因查询,基因组区间共线性分析等功能。

☆ 小麦蛋白质组数据库
☆ 小麦及其祖先的基因组变异数据库:适用于多套小麦及祖先种变异数据集的查询,可对大规模样本的VCF文件实现在线的快速检索和分析可视化。
☆ 小麦基因组变异与选择信号数据库WGVD:汇总了从968个面包小麦及其祖先中收集到的SNPs、indels,以及基于93个小麦全基因组重测数据的SNPs评估的小麦驯化和改良过程中的选择特征。数据库主要提供变异、基因组选择信号搜索、基因组浏览和比对功能。
☆ 小麦WheatCNVb数据库:基于小麦CNVb新型分子标记系统,支持查询小麦种质资源的CNVb标记,可视化CNVb指纹,并进行基于CNVb指纹的品种比较。
☆ 利用整合基因调控网络探索小麦功能基因的交互式平台wGRN:提供了基因调控查询、功能基因预测和QTG挖掘等十余个功能模块,建立了小麦整合基因调控网络,实现了可持续整合大规模调控数据集的网络化研究框架。
☆ 异源六倍体小麦及其祖先的比较共表达网络分析数据库WheatCENet:集成了功能注释,蛋白质-蛋白质相互作用、miRNAs,GO注释、GSEA等工具能够搜索和比较特定的功能共表达网络,识别其中聚集的基因的相关功能。
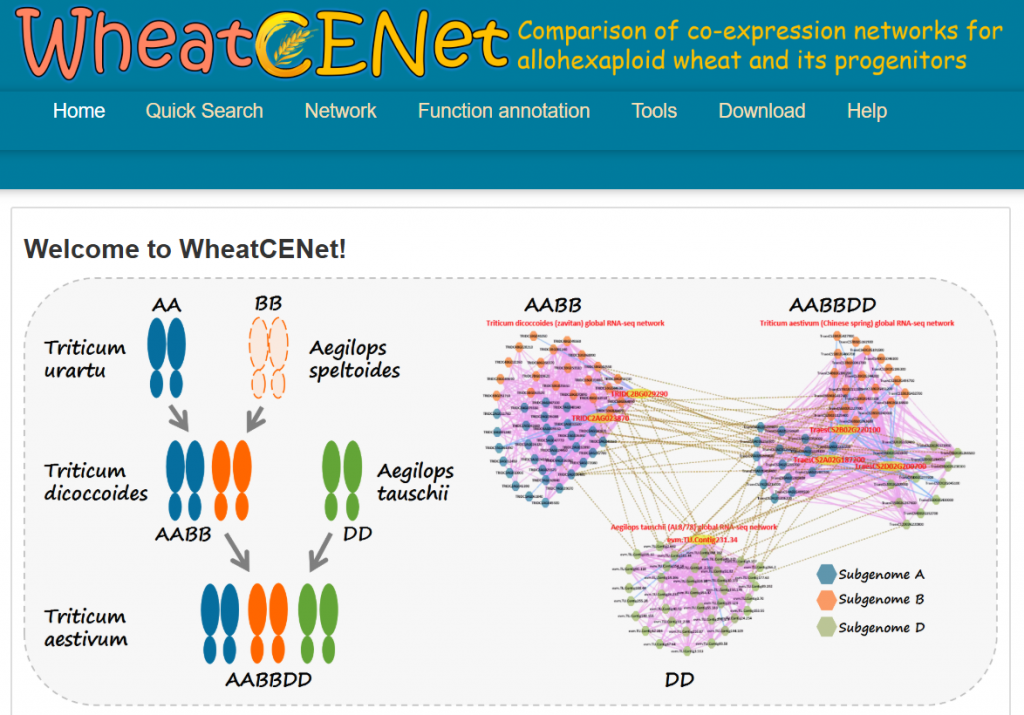
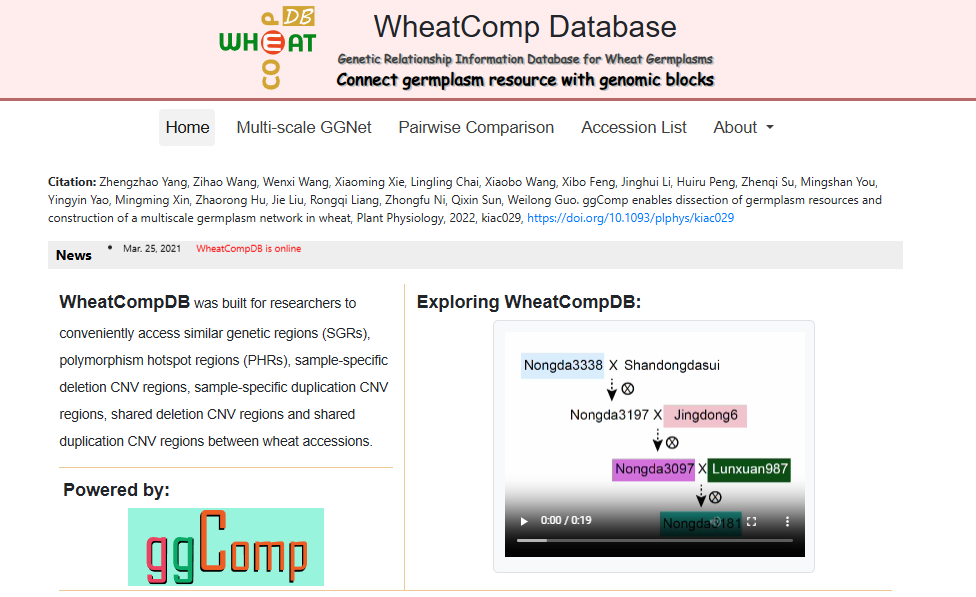
☆ 小麦种质资源血缘区间比较数据库WheatCompDB:主要针对数据集中任意两个样本间的种质资源血缘区间比较分析,支持基因组水平、染色体水平和局部区间水平下种质资源网络的构建和挖掘。
玉米数据库
☆ 玉米基因组和遗传分析平台:数据库包含了玉米的基因组序列、基因注释、遗传图谱、突变体信息、表达数据、相关的文献和研究工具。旨在支持玉米遗传学和基因组学的研究,为科研工作者提供访问和分析玉米的遗传和基因组数据的平台。
☆ 玉米及其野生近缘种的基因组工程资源库:包括玉米及其野生亲属禾本科的复杂性状,稀有遗传变异如何影响整体植物功能。
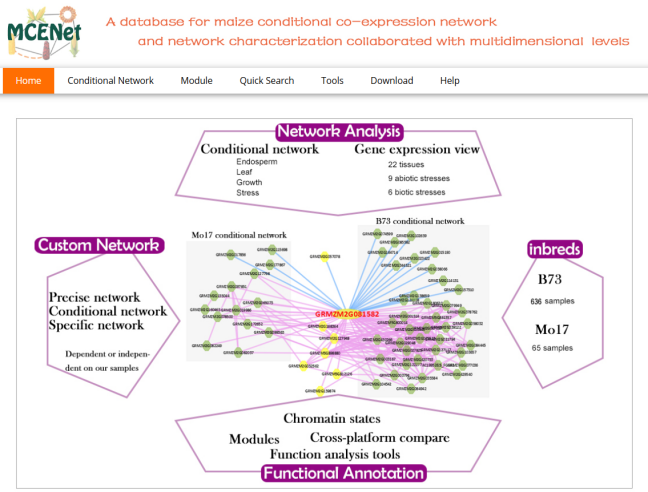
☆ 玉米多组学基因网络分析平台:整合了701个转录组学数据和108个表观基因组数据,并研究了具有多维组学水平的不同条件网络。MCENet 还提供 5 个网络访问分析工具包(即 Network Search、Network Remodel、Module Finder、Network Comparison 和 Dynamic Expression View)和多个网络功能支持工具包。
☆ 适应玉米多组学时代的综合数据库:整合了来自于同一玉米群体的基因组、转录组、表型组、代谢组、表观基因组、遗传变异以及遗传定位结果等多组学数据,同时收录了多个玉米基因组,可以进行比较基因组、共线性分析、表达聚类、遗传变异基因分型、连锁图谱、定位结果、组蛋白修饰以及甲基化等多组学数据进行检索和分析,实现在不同组学数据之间进行跳转。

☆ 拟南芥和玉米蛋白质组数据库:为通过实验鉴定拟南芥和玉米中的蛋白质提供了集成资源。内部BLAST比对链接玉米和拟南芥信息,实验鉴定基于细胞类型特异性蛋白质组或特定亚细胞蛋白质组(例如叶绿体,类囊体,核苷)和总叶蛋白质组样品的内部质谱(MS)。

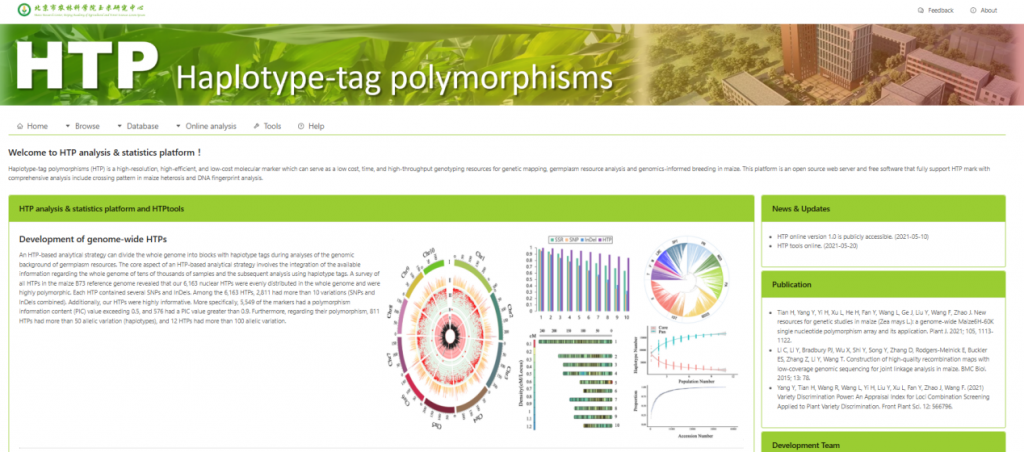
☆ 利用玉米单倍型标签多态性分析工具:HTP数据库(HTPdb):该数据库覆盖了育种中常用的3,587个重要玉米自交系,其中包括172,921个非冗余的HTP等位基因变异,提供了丰富的单倍型标签资源。同时,基于HTP标记的特点,研究人员在HTPTools中集成了智能区块数据填充算法,当仅需获取少量HTP标记的基因型时,可以通过该功能快速地获取对应区块的完整数据。
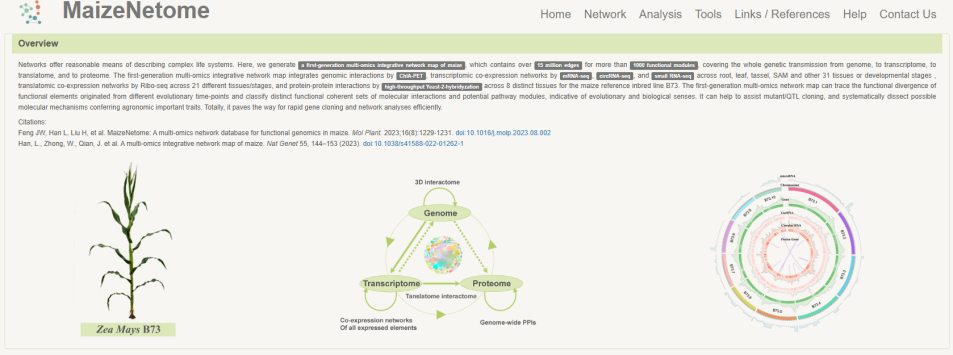
☆ 玉米网络数据库MaizeNetome:其中包含从基因组到转录组、翻译组和蛋白质组的整个遗传传递。多组学整合网络图谱通过ChIA-PET整合基因组相互作用,包括跨根、叶、穗、 SAM和其它31个组织或发育阶段mRNA-seq、circRNA-seq 的转录组共表达网络,跨 21 个不同组织/阶段Ribo-seq 数据, B73 材料8 个不同组织的酵母杂交蛋数据。数据库可以帮助辅助QTL定位,并系统剖析农艺性状的可能分子机制,为快速基因克隆和高效的网络分析铺平道路。
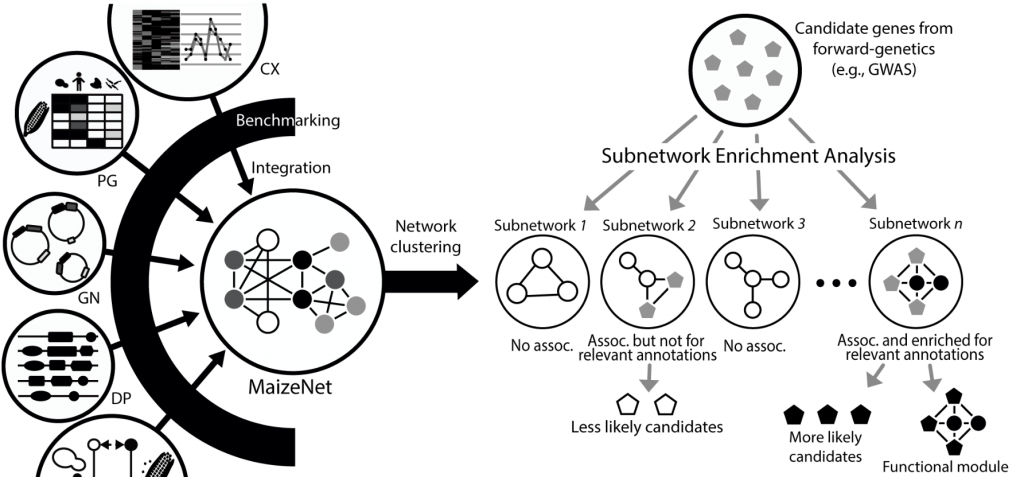
☆ 全基因组范围的玉米基因辅助功能网络数据库MaizeNet:是通过集成 21 个组件网络构建的,其中每个网络都是从不同的组学数据中推断出来的。 通过整合异构数据,MaizeNet 提供了玉米遗传结构的全面视图,为特定生物过程确定候选基因的优先级。可以利用 MaizeNet 中实现的优先级服务来生成新的功能假设,缩小候选基因的范围,并确定该功能的新基因。
☆ 中国玉米品种系谱数据库:收录自交系及品种信息10000余条,涉及自交系1218个,杂交种7823个。该数据库实现了品种信息检索、系谱追溯、子代查询、定制化查询、用户上传及纠错系谱信息等功能,便于中国玉米育种家及科研工作者快捷查询玉米自交系及品种的信息及系谱关系。

大豆数据库
☆ 水稻、小麦和大豆的分子育种资源库:这是一个集成数据库,收集了群体测序、种质资源、表型和各种基因组数据。
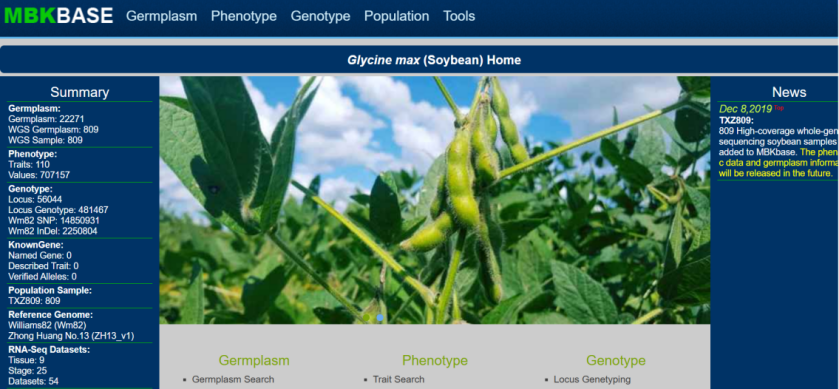
☆ 大豆基因组学和分子生物学数据库:为大豆研究者提供了大量的数据和工具,以支持大豆的遗传学、基因组学和育种研究。用户可以通过SoyBase查找大豆的基因组信息、遗传资源、表型数据等,并利用其中的分析工具进行数据挖掘和可视化。
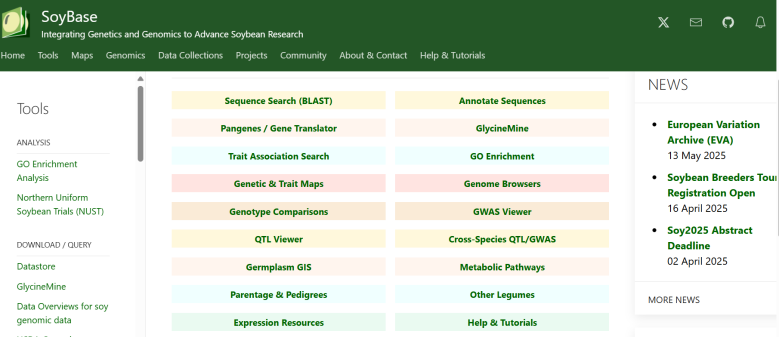
☆ 大豆种质资源组学数据库:用户可提供离散值的表型数据来帮助用户识别用于育种或遗传研究的“有用”种质资源,实现了2K-SG的33个数量性状与9个质量性状的共享;用户可以利用SoyFGB或未公开表型数据来实现表型和单倍型变异的相关性在不同基因组分辨率下的在线解析;获得基因组作图定位与表型性状相关区域,使用 “搜索”和“浏览”模块,用户可以获取2K-SG 的基因组变异,用于实验验证。
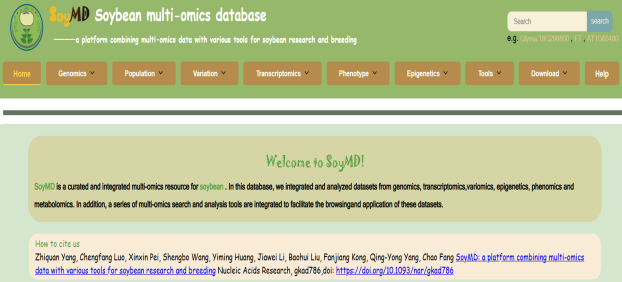
☆ 大豆多组学数据库SoyMD:共提供了8个组学模块以方便用户浏览和分析多组学数据,包括基因组、转录组、表观基因组、表型组、变异组、工具、下载和帮助模块。
☆ 大豆多组学数据库SoyOmics:从基因组、变异组、转录组、表型组等不同层面整合了大豆相关数据集,实现了不同层次组学数据的交互查询和联合比较分析。数据库目前收录了多个大豆品系的基因组组装,注释数据,以高质量的ZH13作为参考基因组,对2898份材料的全基因组测序数据进行了全基因组序列变异检测,鉴定到约3800万条SNP/INDEL变异数据,提供了来自大豆泛基因组分析的约55万条结构变异数据以及基于结构变异构建的图泛基因组。收录了多个品系不同组织时期的表达数据。数据库针对115个表型多年多点测定的约2.7万条表型记录进行了本体注释和归类,并将表型数据与变异数据进行关联。除以上组学数据外,数据库同时提供了甲基化测序数据,Soy40K大豆芯片数据。
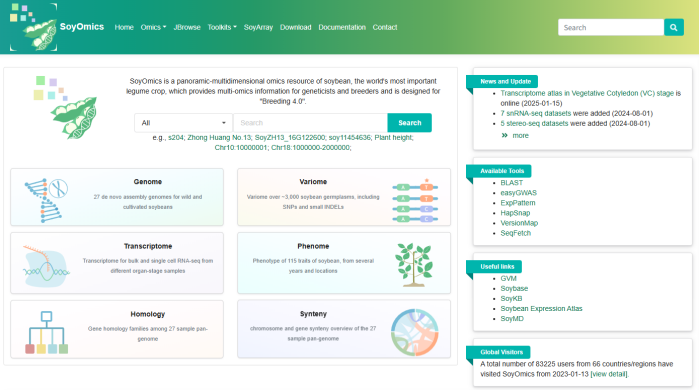
☆ Soybean Expression Atlas的升级版本其中包含 5481 个公开可用的 RNA-seq 样本的转录本和基因级转录本丰度矩阵。
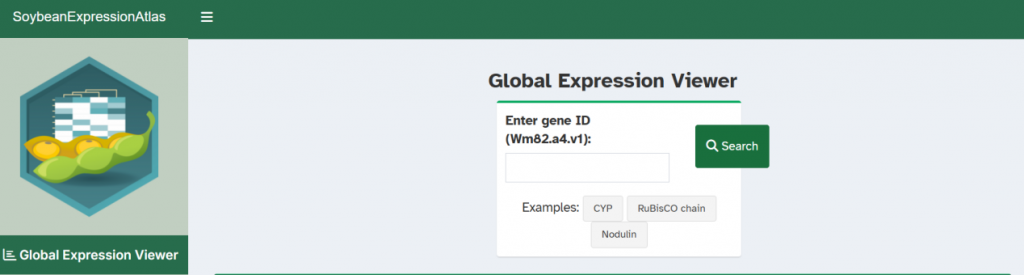
☆ iSoybean:收集了EMS诱变的大豆突变种群,并利用全基因组测序技术对1,044个突变系中的变异进行了详细表征。该网站致力于提供关于大豆突变体的详尽信息,并通过种子库分发这些突变体,促进其在功能基因组学研究领域的快速应用。

☆ SoyDNGP:构建了一个基于预测模型SoyDNGP的大豆表型预测平台,为基于基因变异的作物表型预测研究提供了创新的工具和方法。
百迈客生物深耕物种多组学数据库建设,已为众多科研机构与高校提供从数据存储到分析挖掘的全流程解决方案,已成功应用,取得了显著的研究成果。无论是物种保护、生态监测还是遗传育种,我们的数据库都为其提供了强有力的数据支持,助力科研人员攻克一个又一个科学难题。

 部分合作案例
部分合作案例
百迈客生物致力于为您提供最准确、最全面的作物数据,将持续更新作物育种动态、种植技术及市场资讯,欢迎科研工作者提出数据需求与合作建议,共同推动农业科技创新。








 京公网安备 11011302003368号
京公网安备 11011302003368号