导读
大量数据表明,肠道菌群在人类健康和疾病的几乎所有方面都发挥极其重要且深远的作用。然而,地球上的大量微生物都是“未经培养的”,这对微生物组研究来说是一个重大挑战。宏基因组分箱(Metagenomic binning)是一种将宏基因组测序得到的混合了不同生物的序列或contigs按物种起源和类别分开归类的生物信息学分析方法,提供了一种不依赖培养的方法来克服各种自然生态位中“未培养的大多数”缺乏参考基因组的问题。
2023年1月5日,内蒙古农学大学张和平教授和孙志宏研究员团队在微生物领域国际期刊《Nature Microbiology》(IF:30.964)上发表了题为“A high-quality genome compendium of the human gut microbiome of Inner Mongolians(“内蒙古人肠道菌群的高质量基因组简编”)的研究论文。该研究基于一项益生菌(Probio-M8)发酵乳人群干预实验,采用混合长读(ONT)和短读HiSeq测序来表征60名内蒙古人的粪便菌群,基于分箱的宏基因组分析策略和方法,构建了内蒙古人肠道高质量基因组数据库(IMGG)。百迈客为该研究提供了ONT宏基因组测序服务。
 主要结果
主要结果1、超深度宏基因组混合测序与组装
研究者利用了之前进行的一项基于生物标记物的纵向人体试验,该试验调查了每天摄入益生菌酸奶(包含Bifidobacterium lactis ProBio-M8,ProBio-M8)与普通酸奶相比的益处。随机挑选了60名参与者在三个时间点(开始食用酸奶后0、7和28d)采集了180份粪便样本,在PromethION 和HiSeq平台上进行了超深度测序,生成了3.7Tbps的三代数据和20.1Tbps的二代数据(即每个样本20.5±4.5 Gbps和111.8±8.65 Gbps)。该混合超深宏基因组测序实现了平均N50长度为278Kbps宏基因组组装,平均总组装长度为314Mbps。*长contig为6.77 Mbps,6688个contig大于1 Mbps。三代数据的质量远远超过了大多数现有的人类肠道三代数据库,目前的三代数据库中平均N50长度为8Kbps;平均reads长度为6 Kbps;平均reads质量为9.5。
为了评估组装流程的准确性,研究者基于三种测序和组装策略,直接从摄入益生菌酸奶的个体粪便样本中组装Probio-M8基因组:即仅三代序列组装、二三代混合组装、仅二代序列组装。对于每种组装方法,分别重建了50个Probio-M8基因组,并与Probio-M8参考基因组进行比较。和预期一致,与单独使用二代或三代测序构建的基因组相比,二三代混合组装的基因组在组装连续性、碱基对和基因预测准确性、基因组完整性、全基因组相似性度量以及功能注释的精准度等多个基因组质量参数方面表现出了极大的提高 。此外,二三代混合组装的基因组组装错误率与二代数据覆盖率呈负相关,但与测序深度无关。因此,二三代混合组装是一种强有力的策略,可以直接从复杂的人类肠道宏基因组数据库组装环状和更加准确的基因组。
2、高效组装种水平的CMAG
基于二三代测序混合组装共获得了802个CMAG,在准备本手稿时,只有225个CMAG保存在公共基因组数据库中。其中大多数(n=160)是从环境元基因组中重建的。该研究组装的CMAG较大,从1.0 Mbps到5.5 Mbps(平均为2.4 Mbps),这个范围涵盖了大多数已知肠道细菌的基因组大小。为了评估目前组装的CMAG的代表性,按照平均核苷酸同源性(ANI)95%阈值进行物种水平基因组去冗余,然后将该数据集与统一人类胃肠道基因组(UHGG)中去复制的基因组进行比较。总共保留了134个种水平的CMAG,涵盖11个门、14个纲、27个目、45个科、94个属(图1A和补充表5)。其中有131个物种水平的CMAG是之前未报道的环状的、完整、人类特有的各自物种的代表性基因组,65个未培养物种的参考基因组的质量得到了改善。这些结果表明了当前工作流程在从复杂的宏基因组数据集和组装挑战的区域组装完整基因组方面的能力和有效性。
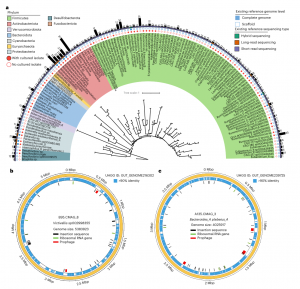
图1.有效组装大量物种水平的完整(环状,无间隙)宏基因组组装基因组
3、内蒙古人高质量肠道基因组数据库
最初的bins是通过两种装箱方法(MetaBAT23和Vamb11)产生的,然后通过DASTool和内部脚本进行整合和改进。连同上述CMAG,该研究总共还原了12,391个符合MIMAG标准中的中位质量标准的MAG(>50%完整性和<5%污染)。其中的6,729个MAG也满足MIMAG标准高质量定义(完整性>90%和污染率<5%,具有5S、16S和23S rRNA基因以及至少18个tRNA(TRNA)基因;称为IMGG数据集)。
接下来,研究者对IMGG数据集中的6,729个MAG和从UHGG收集检索到的147,835个高质量基因组进行聚类分析(图2A)。聚类过程产生了485个宏基因组种(MGS),其中至少包含一个IMGG。从分类上划分为11个门、14个纲、30个目、40个科,横跨220个属(图2B和补充表8)。
工作流程显著提升了MAG的连续性(图2C),这从IMGG数据集中的contig数量显著少于UHGG数据集中的N50长度或代表性基因组(重叠群数量;N50长度。此外,尽管UHGG数据集包含相当多的MAG(n=111,744),但只有5,053个MAG符合MIMAG标准中规定的高质量标准,而且其中大多数(n=4,058)是培养的分离物,这表明基因组质量需要进一步完善。另一方面,该研究为288个物种提供了具有代表性的高质量MIMAG参考基因组,可用高质量MIMAG的数量在154个物种中扩大了50%以上,其中包括一些在UHGG目录中具有较高代表性的物种,如Agathobacter rectalis、Alistipes putredinis、Bacteroides_B dorei和Lachnospira eligens_B(图2D和补充表8)。

图2.内蒙古人肠道基因组IMGG数据库是一个扩展的基因组资源
4、提高肠道复杂基因组区域的分辨率
为了揭开复杂的肠道基因组区域(包括rrn、MGC、前噬菌体和IS),该研究将从IMGG数据库组装的485个MGS与UHGG数据库中的对应片段进行了比较。正如预期一样,与UHGG相比,IMGG有着更多的rrn拷贝(包括16S、23S和5S rRNA)(图3a);并且IMGG和NCBI基因组数据库中对应的完整分离基因组的rrn拷贝数基本一致,这表明UHGG数据库中MGS严重低估了的rrn基因拷贝数。此外,通过对全面的基因拷贝数数据库(rrnDB)进行分类搜索,其中85%(413/485)在当前组装的MGS是找不到的,包括一些16S基因拷贝数高的未培养MGS。这些结果表明,由于基因组质量不足,目前可用的基因拷贝数参考数据库在很大程度上仍然不完整。
研究者进一步使用MGC预测工具gutSMASH,比较了IMGG和UHGG中MGC的完整度。结果表明,在UHGG中发现的mGCs有很大比例(56.5%)位于contig边缘,可能是不完整的,而在IMGG中只有4.5%的比例较低(图3B)。此外,在IMGG和UHGG数据集中分别发现了每个基因组具有6个和3个完整的MGC,这表明在IMGG中识别全长MGC方面有了实质性的改进(图3B)。包含MGC编码序列的基因组比例在IMGG中显著高于UHGG(图3C),特别是在几个MGC类别中,包括非蛋白源氨基酸、芳香族和其它短链脂肪酸(图3D)。值得注意的是,MGC区的基因组比例只与短读基因组的组装N50长度有关,而与IMGGs无关,这表明MGC识别和分配的有效性和分辨率受到短读数据组装连续性不足的限制。
与UHGG数据库相比,IMGG数据库中前噬菌体基因组的分辨率有了很大提升。该方法在95%的IMGG中都能检测到前噬菌体序列(与UHGG(55%)相比)。IMGG和UHGG每个基因组编码前噬菌体的平均数量分别为4个和1个。IMGG中的前噬菌体序列的连续性也较高,N50长度为37974 bp(UHGG中为31064 bp;图3f);IMGG中前噬菌体编码区的基因组比例是UHGG的4.2倍(图3g)。与UHGG相比,IMGG中的IS区域更加jing q,表现在在每个物种的IS区域总拷贝数(增加了三倍;图3h)和IS编码区域的基因组比例(增加了近四倍;图3i)。IS类别中改善程度*高的是IS1380(与UHGG相比,改善了5.6倍),其次是IS5、IS1634和ISAS1(分别是5.5倍、5.5倍和5.3倍;图3j)。
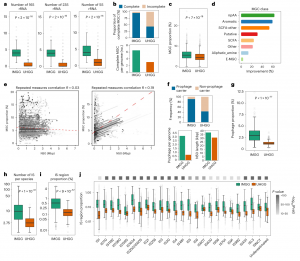
图3.内蒙古人肠道基因组增强遗传元件的基因组分辨率
5、人类肠道微生物中高度多样化和分化的 MGC
进一步深入探索了可用的高质量基因组中编码的代谢潜力。分析了 UHGG 和 IMGG 数据集中的高连续性和高质量基因组。共从15512个基因组中还原了97428个MGC区域,其中78675个是完整的,并被纳入进一步分析。这些完整的MGC属于所有八个MGC类别,分布在58个MGC类型中。*常见的类别是SCFA(40.0%),其次是Putative(28.9%)和E-MGC(17.2%)。然后,门水平分布和聚类分析揭示了MGC类别的总体分布在门水平的显著差异(图4a),以及在十个主要的门之间,它们在不同门中的数量各不同(图4a)。此外,基于涵盖九个主要门的功能性MGC分类分布的特异性聚类表明,MGC概述和组成在门水平间存在显著差异,这些未培养物种中的一些最近发现了以前未报告的Christensenellales和Oscillospirales分类群,突出了这些代表性不足的分类群在促进宿主健康方面的潜力(图4c)。
约12%的MGC具有多个核心功能域。混合MGC显著大于单一功能域MGC(P<2×10−16;扩展数据图4A)。普遍的混合MGC组合是丁二酸酯和红细菌固氮(RNF)复合体。大的混合MGC大小为117Kbps。它含有5个MGC功能结构域,其中两个属于E-MGC和SCFA-其他类(分别属于RNF复合体和乙醇胺利用途径),占MGC总长度的80%以上。这些数据表明,肠道微生物组MGC编码了广泛的代谢潜力,不同关键的人类肠道分类群的代谢潜力差异很大。这项研究为系统揭示人类肠道代谢潜能提供了起点。
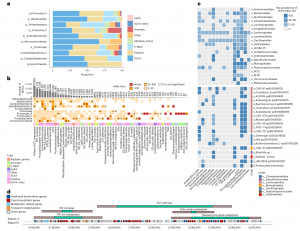
图4 人肠道菌群中代谢基因簇库概述
6、2834个肠道前噬菌体的分类和功能注释
噬菌体是另一组被低估的肠道微生物群。因此,研究者进一步挖掘了 IMGG 数据集以寻找未知的肠道噬菌体。从 IMGG 数据集中还原了总共 21,217 个前噬菌体基因组,通过应用未培养病毒基因组 (MIUViG) *低信息标准,进一步聚类为 13,437 个物种水平病毒操作分类单元(vOTUs)。这些 vOTU 代表 39,839 Kbps N50 长度。
通过与宏基因组肠道病毒(MGV)数据库进行比较,对vOTU进行分类。值得注意的是,95.5%(n=12834)的vOTU与MGV数据库在物种水平不具有同源性。除了那些可分类到物种水平(4.5%)的病毒外,90.9%的vOTU与MGV水平具有科水平的同源性,但只有46.7%的病毒被分类到已知的病毒科(图5a)。进一步分析表明,常见的细菌宿主是Firmicutes_A(71.4%),这也是先前未报告前噬菌体的主要宿主。Siphoviridae和Myoviridate都是人类肠道中的优势前噬菌体科,其宿主范围广泛,涵盖多个门(图5c);然而,Siphoviridae(Firmicutes_A [68.8%],Bacteroidota [15.58%],Actinobacteriota [5.3%])和Myoviridae [Firmicuters_A[83.0%])的主要细菌宿主在门水平的分布却有很大差异。如预期那样,crAssphage的宿主范围小且具有特异性,仅由拟杆菌属组成。
接下来,对IMGG数据库中的前噬菌体基因组进行深入分析,以探究其功能能力。从12834个物种水平的vOTU代表性基因组中鉴定出596193个蛋白质编码基因,并将这些假定基因与几个常见的功能和/或病毒注释数据库进行了比较。结果表明,仅有55.4%的前噬菌体基因在功能上能够被分类,而44.6%的基因与任何交叉比较的数据库都不具有同源性,被分配给了未知的功能(图5d)。这些结果表明,尽管携带ARG的噬菌体很少存在于肠道微生物中,但这些噬菌体可能编码ARG(例如,A163.CMAG_1_1_7_59800编码假定的β-内酰胺酶基因;图5h),可能成为ARG转移的关键载体,并对人类健康造成严重威胁。这些结果表明,肠道前噬菌体在分类和功能上比先前我们所了解的更加多样化。
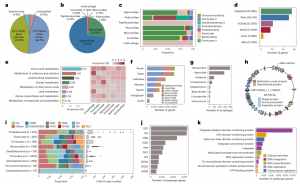
图5 肠道中前噬菌体和插入序列的概述
总 结
在该项研究中,使用混合长读长 PromethION 和短读长 HiSeq 测序对 60 名内蒙古人(三个时间点, 180 个样本)的粪便微生物群进行了表征。构建了 IMGG 数据库,其中包含 802 个环状基因组和 5,927 个符合 MIMAG 标准中规定的高质量基因组标准的高质量基因组,极大地扩展了当前人类肠道高质量 MIMAG 数据库(n = 7,492)。此外,该 CMAG 基因组数据集,是目前可用 CMAG 的三倍多。因此该研究构建的IMGG 数据库是一个有价值的基因组资源。
内蒙古农学大学张和平教授和孙志宏研究员为该论文通讯作者,第一作者为食品科学与工程学院2018级博士研究生靳昊,2022级博士研究生全柯谕和何秋雯助理研究员为共同第一作者。
参考文献
Jin H, Quan K, He Q, et al. A high-quality genome compendium of the human gut microbiome of Inner Mongolians. Nat Microbiol. 2023;8(1):150-161. doi:10.1038/s41564-022-01270-1








 京公网安备 11011302003368号
京公网安备 11011302003368号