小编掐指一算,好久没有和大家分享SLAF遗传图谱的文章了,今天,小编带来百迈客与客户合作发表的两篇遗传图谱的成功案例,给大家尝尝鲜图片
案例一:苹果F1群体高密度遗传图谱的构建与相关性状的QTL分析

合作单位:山西农业大学
发表时间:2021.09
材料与方法
群体构建:‘Y-2’(Malusdomestica)בDanxia’(Malus baccata)杂交构建的F1群体
性状调查:株高 (PH)、树干直径 (TD)、总花数 (TF)、分支花数 (BF)
测序技术:SLAF-seq
图谱构建:HighMap
QTL定位:mapQTL
研究结果
4个表型性状评估
在2015-2017年,对F1群体的4个表型性状进行了评估,其中,PH是通过用尺子测量从地面到树冠顶部的方式来调查;TD是记录移接处上方10cm的直径;TF和BF是计算总花数和分支花数。此外,相关性分析显示,PH和TD呈显著正相关(r>0.74, p<0.01)。
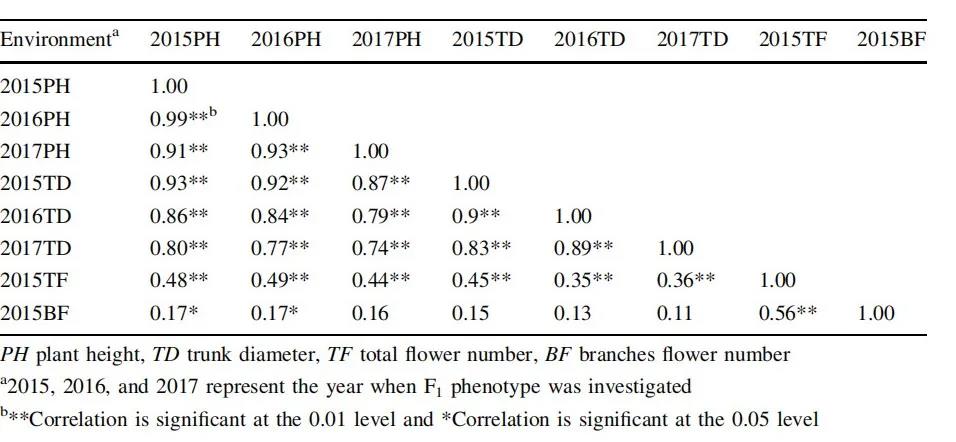
SLAF测序与基因分型
对双亲和150个F1子代进行SLAF建库测序,双亲共获得2.28GB的数据量,每个子代获得~554Mb的数据量,数据质控显示~93.42%的reads质量值达到或超过Q30;亲本及子代的平均测序深度分别为15.57×(母本)、16.9×(父本)和6.28×(子代),共开发出337,249个SLAF标记,其中,132,722是多态性标记,在这些多态性标记中,36,829个标记被成功分成8种基因型((ab × cd, aa × bb, ab × cc, cc×ab, ef × eg, hk × hk, lm × ll 和 nn × np) ,由于双亲均为杂合种,进而利用除aa × bb基因型外的7种标记(16,126)用于后续的图谱构建。
苹果17条染色体的SLAF多态性标记分布
遗传图谱构建
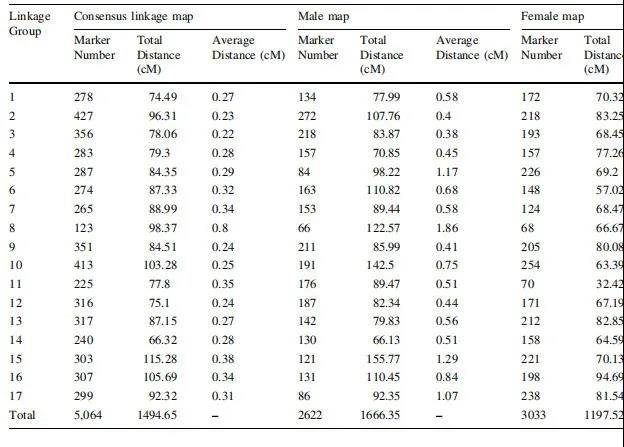
最后,利用HighMap将5113个高质量SLAF标记用于遗传图谱构建,其中,雌性图谱包含2622个SLAF标记,总图距1666.35cM。雄性图谱包含3033个SLAF标记,总图距为1197.52cM。中性图谱包含5064个SLAF标记,形成17条连锁群,总图距为1494.65cM,平均图距为0.36cM。在该图谱中,有189个SLAF标记在11个连锁群中存在偏分离,包含112个SLAF标记的LG7标记偏分离率*高。在LG1、LG2、LG3、LG6、LG12和LG16中均未出现偏分离现象。并对以上图谱进行了单体来源评估、热图评估和共线性评估,结果表明该图谱是高质量的评估遗传图谱。
 QTL定位
QTL定位
为了检测F1群体中与4个性状相关的位点,对其进行QTL分析。对于PH,结合3年的表型,在11号染色体上共鉴定到5个QTLs(qPH15-1, qPH16-1, qPH15-2, qPH16-2和qPH17),这些QTLs对表型变异的解释范围为17.20% ~ 22.40%。对于TD,共定位到6个QTLs(qTD15-1, qTD16-1, qTD17-1, qTD15-2, qTD16-2和qTD17-2),均位于11号染色体上,对表型变异解释范围为14.00~20.80%,PH与TD定位结果相重叠,均位于两个QTL簇,该结果进一步证实了PH和TD在表型上呈显著正相关。对TF,定位到2个QTLs(qTF15-1和qTF15-2),分别位于2和4号染色体上,解释表型变异率分别为8.80%和9.20%;另外,还定位到1个BF相关的QTL qBF15,可以解释34.80%的表型变异。qTF15-2和qBF15在4号染色体被定位到同一个染色体区间内。
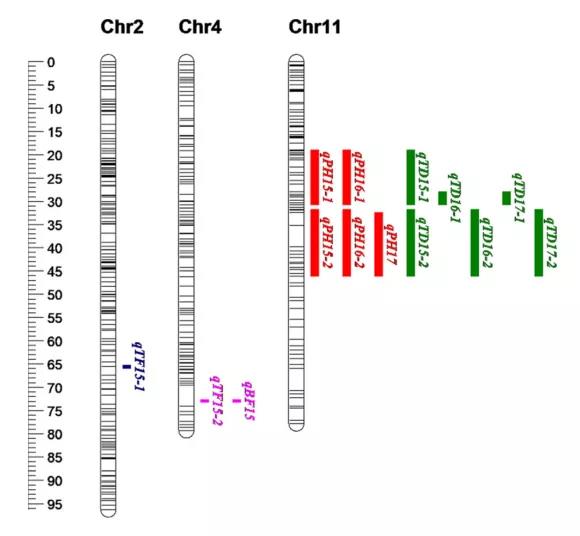
‘Y-2’בDanxia’ F1群体中四种表型的QTL分析
案例二:利用R1R1 × R6R6群体进行苹果红肉位点的连锁定位和QTL定位

合作单位:西北农林科技大学
发表时间:2021.09
材料与方法
群体构建:‘Fuji’(非红肉,携带纯合的R1R1等位基因)和‘Red3’(深红色的果肉,携带纯合的R6R6等位基因)构建的F1群体,包含168个单株(携带杂合的R1R6等位基因);
性状调查:采集成熟果肉进行矢车菊素半乳糖苷(cyanidin-3-galactoside)浓度测量,每株3个重复,每个重复5个果实;
测序技术:SLAF-seq
图谱构建:Joinmap
QTL定位:MapQTL
研究结果
F1群体表型评估
矢车菊素半乳糖苷是红果肉苹果中的主要红色素,作者首先对F1群体的矢车菊素半乳糖苷浓度进行测量,发现它们的分布倾向于低浓度的矢车菊素半乳糖苷,共有139个分离个体显示出低水平的矢车菊素半乳糖苷浓度,并且个体之间表现出显著的差异。
分离群体的SNP基因分型
SLAF测序共获得108.49Gb的数据,Q30为94.07%,GC含量为40.32%。其中,母本的测序数据量为9.69Gb(12.92×),父本的测序数据量为9.35Gb(12.47×);F1群体的SLAF标签数量为152,603个,多态性SLAF标签为120,974个;共鉴定出7,997,517个SNPs,其中,成功将8599个有效SNPs分成4种基因型(ef × eg, hk × hk, lm × ll, 和nn × np)。
苹果遗传图谱构建
经过人工筛选后,有969个SNPs无法定位到连锁群或者存在偏分离,最终有7630个SNPs用于构建遗传图谱。母本和父本的上图标记分别有3903个和3925个。对于母本‘Fuji’遗传图谱,每个连锁群的SNP数量范围是为108 ~ 366个,平均为231个,每个连锁群遗传距离为70.17 ~ 168.23 cM,平均遗传距离为109.14 cM;对于父本‘Red3’遗传图谱,各连锁群SNP位点数为58 ~ 434个,平均为230个,各连锁群遗传距离为72.11 ~ 227.10 cM,平均遗传距离为140.26 cM;‘Fuji’和‘Red3’的总图距分别为1855和2384 cM,平均图距分别为0.47和0.61 cM。中性图谱共17个连锁群,总图距为2270.21cM,平均图谱为0.3cM;并且该图谱与基因组物理图谱具有很高的共线性(各连锁群的相关系数范围为0.81 ~ 0.99,平均值为0.94)。
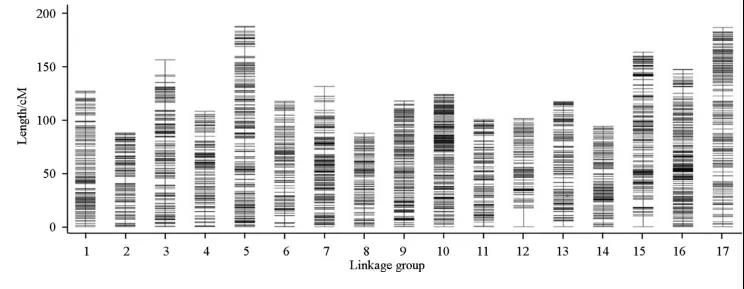
苹果SLAF遗传图谱
成熟果肉中矢车菊素半乳糖苷浓度QTL鉴定
采用Kruskal-Wallis检验对矢车菊素半乳糖苷浓度进行QTL分析,采用区间作图(IM)和多QTL模型(MQM)检测潜在的QTL,阈值LOD为3.0。结果显示,只在‘Red3’的LG16位点的顶端检测到一个LOD值为4.49的QTL,该QTL的区间范围为0 ~ 40.79 cM,解释表型变异率为14.4%。此外,该QTL两侧有两个标记,分别是marker2187260 ~ marker2173766,作者进一步调查了不同株型之间的表型差异,结果表明,最接近QTL峰的标记之一mark2175442将分离群体分为纯合子ll型和杂合子lm型两类。纯合子ll型表现出明显高于杂合子lm 型的矢车菊素半乳糖苷浓度。结合前人的研究,该研究结果表明,LG16顶部的QTL热点区域可能在控制苹果红肉着色中发挥潜在的作用。
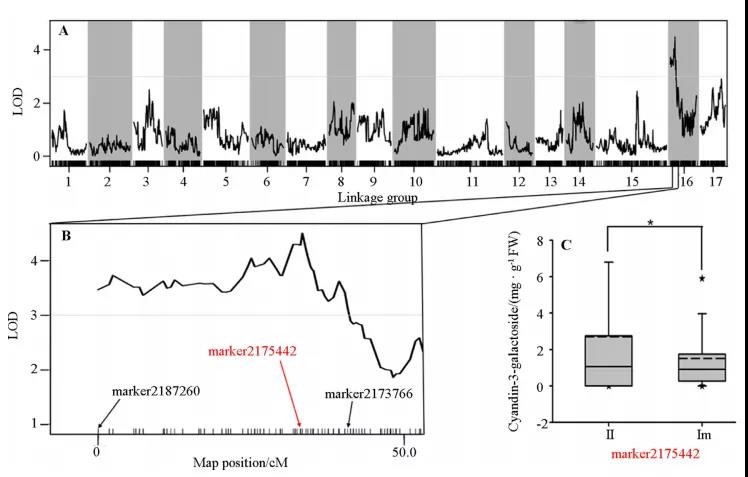
矢车菊素半乳糖苷浓度QTL分析
总结
遗传图谱作为经典的功能基因定位方法,可以用来探索不同物种的复杂性状;上述研究基于苹果SLAF高密度遗传图谱对苹果的株高、树干直径、果实红肉等性状为苹果的遗传研究提供了基础,这些结果丰富了苹果的接穗矮化、苹果果肉着色的育种资源。
SLAF-seq技术是百迈客研发的一种简化基因组技术,此技术酶切方案灵活、避开重复序列、不受参考基因组的限制。SLAF-seq技术已成功应用于群体进化、图谱构建、QTL定位和大规模群体的全基因组关联分析等研究中,帮助科研者进行功能基因定位和分子育种。目前,此技术已在多个物种中成功实施,涵盖作物、林木、花卉、果树、家畜家禽、水产、昆虫等逾百个物种,多项研究成果发表在国际杂志上。








 京公网安备 11011302003368号
京公网安备 11011302003368号