在动植物遗传育种中,通过连锁分析对数量性状位点(Quantitative trait locus,QTL)和质量性状基因进行定位,对于加速育种改良的进程具有重要的意义。
连锁分析是基于家系群体利用连锁的原理研究相关基因与遗传标记的关系的一种研究方法。根据基因在染色体上呈直线排列,不同基因相互连锁成连锁群的原理,即应用被定位的基因与同一染色体上另一基因或遗传标记相连锁的特点进行定位。
对于动植物群体,通常利用性状具有显著差异的两个亲本进行杂交,再通过自交、回交或者其他方式来构建家系群体。家系群体按照遗传稳定性划分可分为暂时性分离群体和永久性分离群体;如F1、F2、F3、BC等,这类群体中分离单位是个体,一经自交或近交,其遗传组成就会发生变化,无法永久使用,所以属于暂时性分离群体;如RIL、DH等,这类群体中分离单位是株系(或者叫株行),不同株系间存在基因型的差异,而株系内个体间的基因型是相同且纯合的,自交不分离。这类群体通过自交或近交繁殖后,遗传组成不会改变,可永久使用,所以属于永久性分离群体。
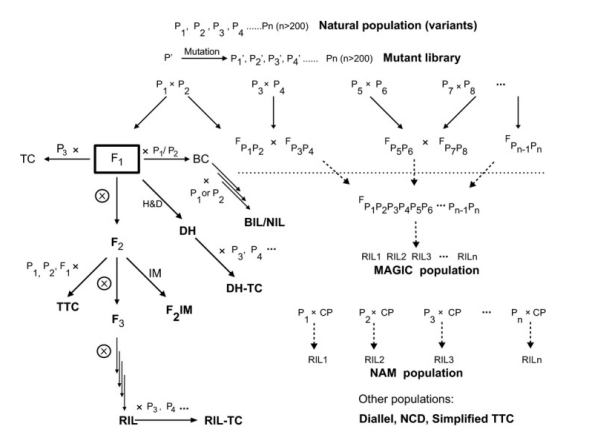
植物家系群体类型[1]
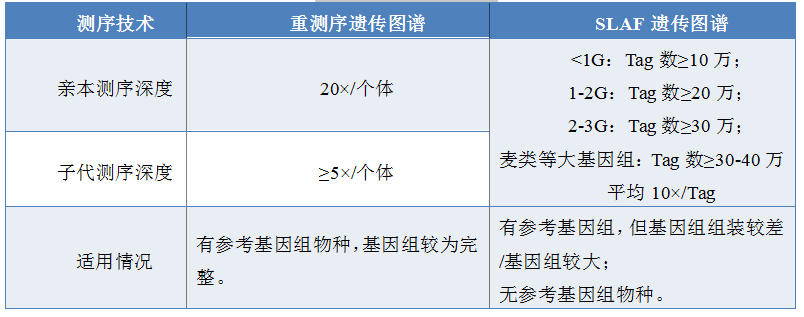
构建遗传图谱方案
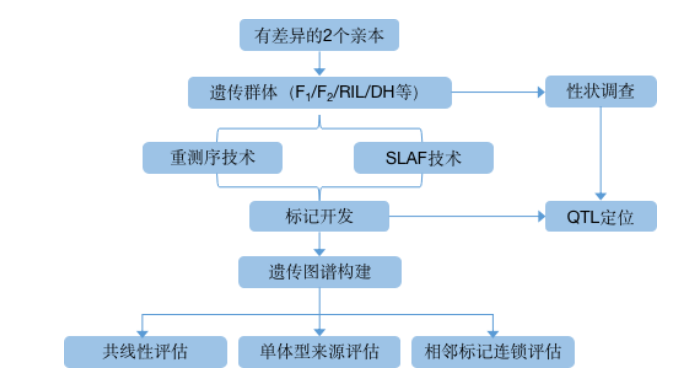
遗传图谱QTL定位分析流程
各群体遗传图谱构建与QTL定位
基于SNP的高密度遗传图谱构建及桃果实相关QTL和候选基因的鉴定[2]
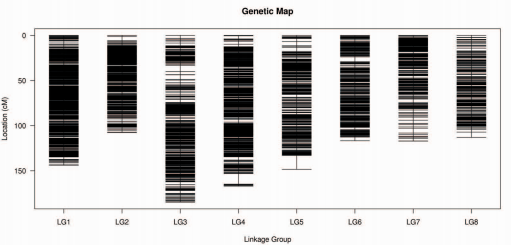
图1 桃遗传图谱
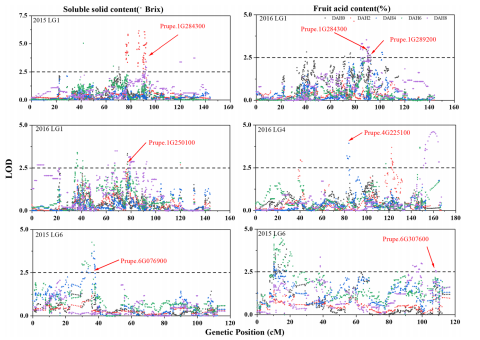
图2 桃可溶性固形物含量(SSC)和果酸含量(FA)QTL定位
2.F2群体遗传图谱
基于高密度遗传图谱和BSA共定位油菜株高相关的候选基因 [3]
本研究在ZS11-HP(高)×sdw-e(矮,自然变异)构建的包含200个子代的F2群体中,分别挑选20株极高和极矮的材料进行混池构建,进行重测序,亲本测序深度为69.96×和69.62×,子代测序深度为32.82×和33.47×,进行BSA分析,通过SNP-index的方法定位到A10染色体上0 Mb-5.5 Mb, 6.0 Mb-12.0 Mb(11.5 Mb大小)的显著区间。同样利用该群体构建遗传图谱,对子代测序7.67×,依据基因型开发出4323个bin标记,利用Highmap构建了油菜的19条连锁群,总遗传距离为2026.52cM;又利用R/qtl的区间作图法(IM)和复合区间作图法(CIM)定位油菜株高性状的QTL位点,定位到3个主效QTL,其中qPHA10与BSA定位结果高度一致,显示出定位的可靠性。
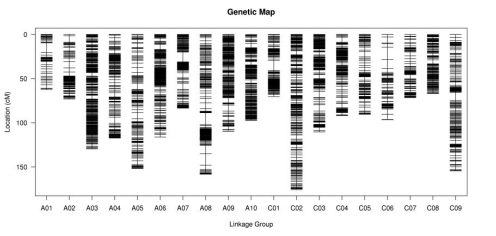
图1 油菜高密度遗传图谱
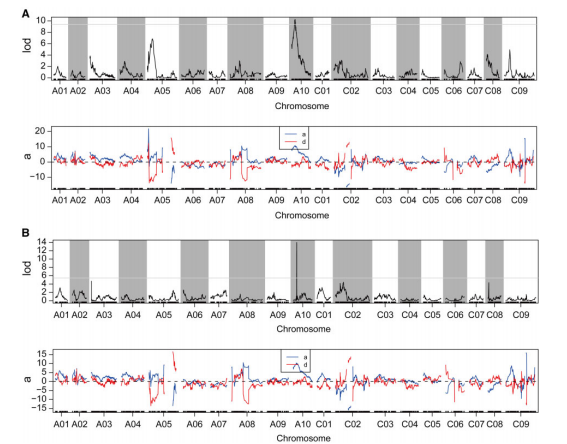
图2 基于 IM和CIM的QTL定位结果
3.BC群体遗传图谱
QTL定位和转录组分析鉴定甜玉米果皮厚度的候选基因 [4]
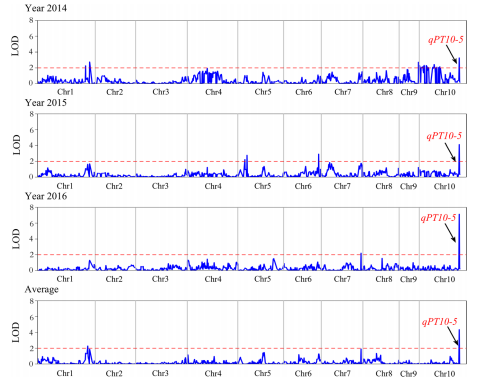
图1 甜玉米果皮厚度QTL定位
4.DH群体遗传图谱
花椰菜高密度遗传图谱构建及紫色萼片性状位点的鉴定 [5]
发表期刊:BMC Plant Biology
影响因子:4.215
合作单位:浙江省农业科学院
分析内容:SLAF遗传图谱
一些西兰花种质有紫色萼片,寒冷的天气会使紫色加深,而其他西兰花品系的萼片即使在寒冷的冬天也始终是绿色的。本研究中,对包含127个子代的DH群体进行SLAF测序并构建高密度遗传图谱。其中上图标记有6694个SLAF标记,平均测序深度为母本81.37×,父本84×,子代15.76×。在所有记录的表型数据中,确定了三个QTL,它们都分布在连锁群1(LG1)上。其中,位于36.393 cM LG1的主效QTL qPH.C01-2是在所有分析中都可以检测到。除了该位点外,根据2018年春季的表型数据,在qPH.C01-2附近还发现了另外两个次要基因座qPH.C01-4 和qPH.C01-5。紫色萼片性状可由一个主要的单基因座和两个次要的基因座控制。西兰花紫色萼片性状的遗传图谱和定位为西兰花其他复合性状的定位和与紫色萼片性状相关基因的鉴定提供了重要基础。
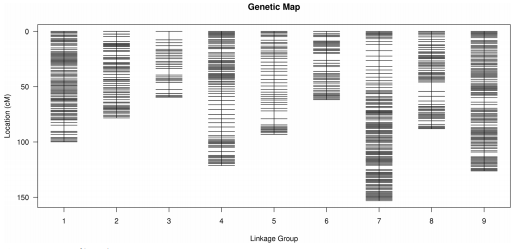
图1 西兰花遗传图谱
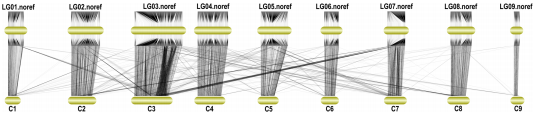
图2 遗传图谱与物理图谱共线性分析
5.RIL群体遗传图谱
基于重测序构建绿豆高密度遗传图谱定位叶形相关性状 [6]

图1 绿豆高密度遗传图
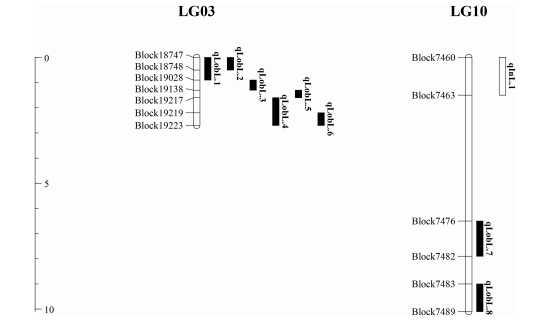
图2 缺刻叶和皱缩叶相关QTL分布
参考文献
[1] Zou C, Wang P, Xu Y. Bulked sample analysis in genetics, genomics and crop improvement. Plant Biotechnol J. 2016;14(10):1941-1955.
[2] Shi P, Xu Z, Zhang S, et al. Construction of a high-density SNP-based genetic map and identification of fruit-related QTLs and candidate genes in peach [Prunus persica (L.) Batsch]. BMC Plant Biol. 2020;20(1):438.
[3] Dong Z, Alam MK, Xie M, et al. Mapping of a major QTL controlling plant height using a high-density genetic map and QTL-seq methods based on whole-genome resequencing in Brassica napus. G3 (Bethesda). 2021;jkab118.
[4] Wu X, Wang B, Xie F, et al. QTL mapping and transcriptome analysis identify candidate genes regulating pericarp thickness in sweet corn. BMC Plant Biol. 2020;20(1):117.
[5] Yu H, Wang J, Sheng X, et al. Construction of a high-density genetic map and identification of loci controlling purple sepal trait of flower head in Brassica oleracea L. italica. BMC Plant Biol. 2019;19(1):228.
[6] Wang J, Li J, Liu Z, et al. Construction of a High-Density Genetic Map and Its Application for QTL Mapping of Leaflet Shapes in Mung Bean (Vigna radiata L.). Front Genet. 2020;11:1032.







 京公网安备 11011302003368号
京公网安备 11011302003368号