2019年7月22日,Nanopore平台全新测序芯片R10到达百迈客实验室,百迈客将开启ONT平台测序新征程!
近年来,由于三代测序相较于二代测序,拥有单分子无需扩增,长度长等优势,因此在各组学研究中大放异彩。而作为三代测序的代表技术之一,Nanopore平台因能获得更长读长,在基因组学研究中更是备受青睐。但相较于二代平台,三代平台测序数据的准确性一直是比较大的劣势。本次Oxford Nanopore Technologies公司即将推出的R10芯片,则剑指准确性。相较于当前广泛使用的R9.4.1芯片,新型R10芯片可提供相当的读长,产量及更准确的数据。


R10芯片测序原理
新升级R10芯片大的更新是纳米孔采用了双读取器(Reader)的设计,能够对同一碱基进行两次信号识别。理论上,同一碱基的识别次数为R9.4.1版本芯片2倍,由此来增加碱基的判断率,减小随机错误,并提高一致准确性。R10与R9.4.1测序方式比较如图1
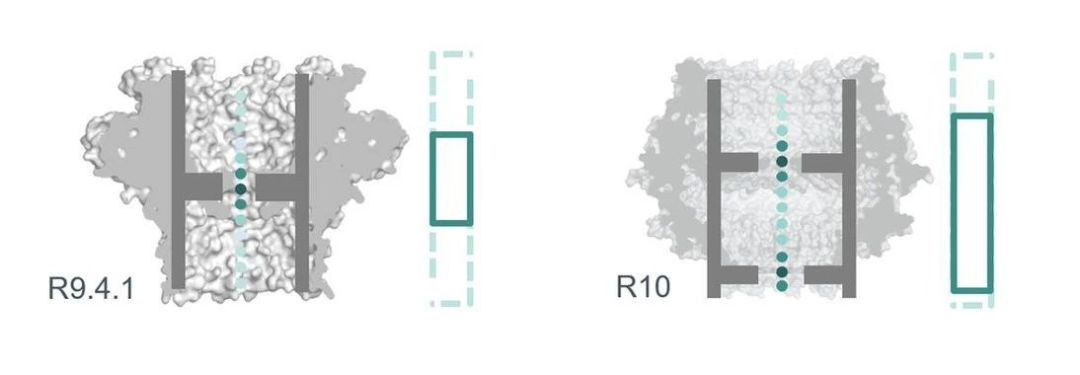
图1 测芯片更新前后测序方式比较
01、单reads测序准确性:
目前主流的R9.4.1芯片的表现能力如何?我们随机抽取了200个已经完成常规ONT DNA测序的样品(包含各种动植物)进行统计,总共约22.4T数据(足够有代表性了),结果如下:
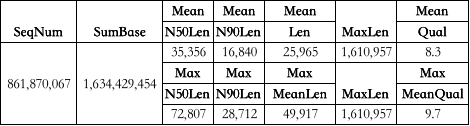
可以看到,R9.4.1芯片数据的读长是非常理想的:Reads N50高达到72 Kb,平均达到35 Kb;单分子读长高达到了1.6 Mb;而关于质量值平均为8.3,对应准确性为:85.21%;单个样品平均质量值高达到9.7,对应准确性为:89.28%;
R10的质量表现如何呢?我们统计了拿到的四个样品的数据,具体统计结果见下图:
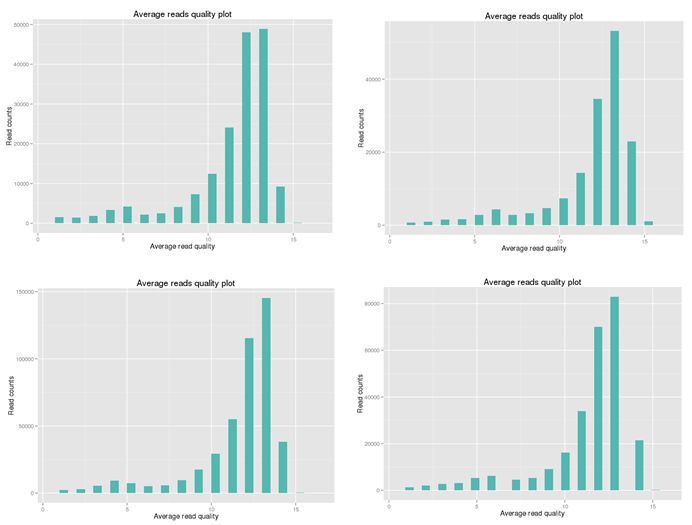
图2 R10数据质量值分布
绝大部分R10 reads的质量值达到了10以上(对应准确性为90.0%),而平均质量值为12.1(对应准确性为93.8%),而reads质量值已经达到了15以上(对应准确性为96.84%)。由此可见,对于单条reads来说,提升还是非常明显的,平均reads准确性从85%提升到了93.8%。
02、Consensus序列准确:
看完单条reads准确性后,那么组装时用到的高深度consensus序列准确性怎么样呢?官方比较了同一样本不同芯片测结果的准确性,见图3。从结果可知,相同测序深度的情况下,consensus后序列的准确性Q值,R10比R9.4.1提升了5~10之间,对应的准确性有的提升了1个百分点之多。
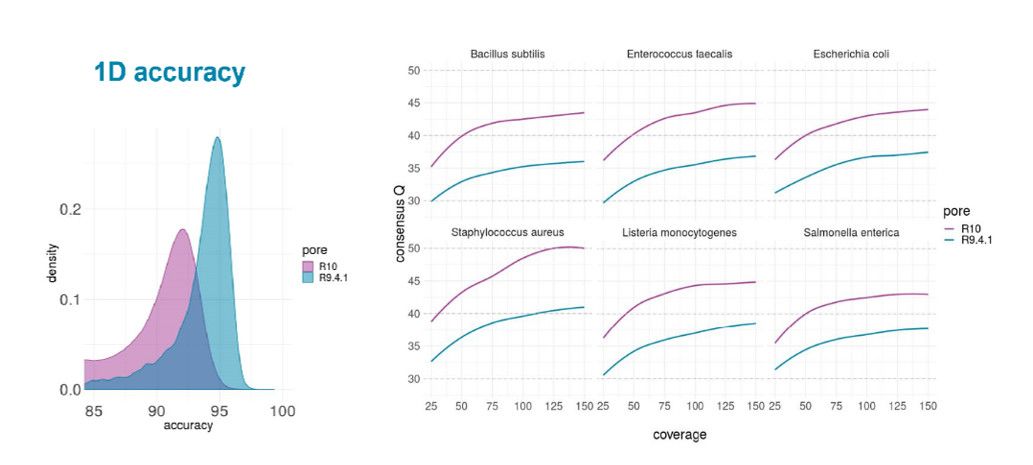
图3 不同物种中R10和R9.4.1 reads准确度
从官方的数据显示,当数据深度为50X时,R10测序数据的consensus序列的准确性已经达到了Q44,即99.996%以上(图4)。且随着测序深度的增加,R10芯片一致准确性逐渐增加。官方对多物种测试发现,在100X的测序深度下,consensus准确性可以保持在Q45以上,在有些样本上甚至已达到Q50(99.999%)水平(图4),当然加大测序深度也可以想要达到更高水平,测试结果中,最高已经达到了Q52(99.9994%)。需要注意的是,这都是未经过二代测序数据矫正的结果!
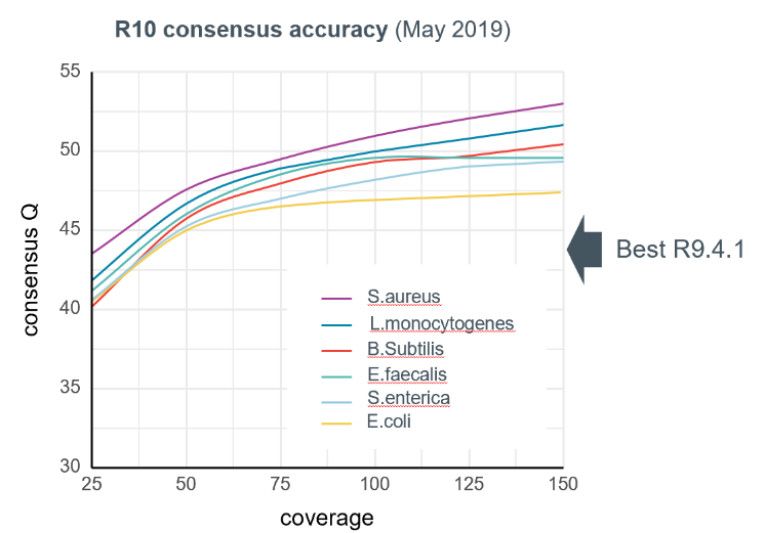
图4 不同测序深度下R10 consensus序列准确性比较
03、同聚物识别准确性:
同聚物的识别准确性是大家一直都比较关心的问题,从ONT官方释放的测试结果显示,相比于R9.4.1,R10版本芯片能够显著提高其在同聚物区域的碱基识别准确度,并且几乎不会引入缺失错误(图5)。
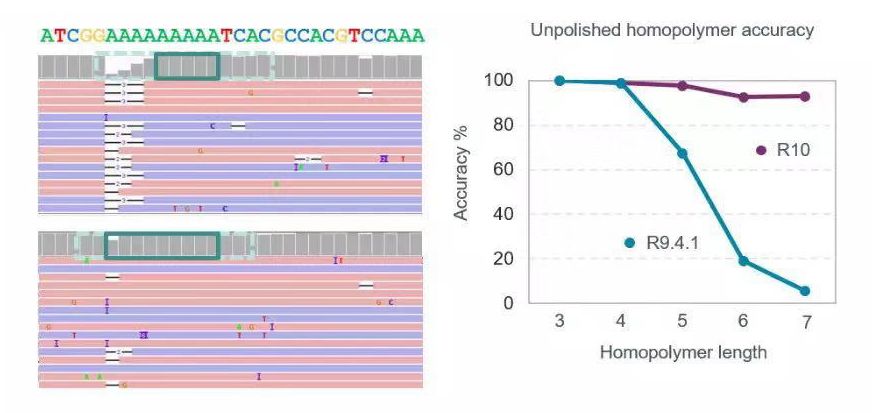
图5 R10、R9.4.1芯片在同聚物区域的测序准确度
04、数据错误分布:
官方通过检测同一样本R10和R9.4.1中的错误在基因组上的情况,发现R9.4.1与R10的错误在基因组上分布并不是完全重叠的,通过两种版本芯片的数据进行混合分析,可极大地提高序列的准确性(图6)。这表明了,在不使用二代数据的前提下,后续也可考虑通过R10和R9.4.1数据组合的方式来提高结果的准确度!
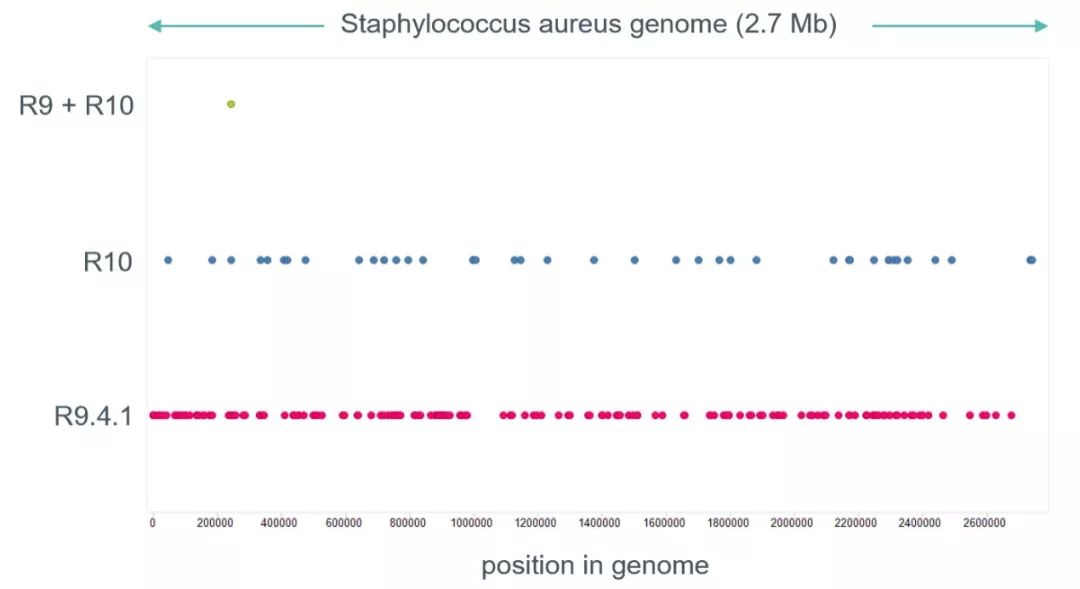
图6 R10和R9.4.1错误分布情况
小结







 京公网安备 11011302003368号
京公网安备 11011302003368号