原文:
Jan Bettgenhaeuser, Simon G. Krattinger.Rapid gene cloning in cereals, Theoretical and Applied Genetics, 2018.
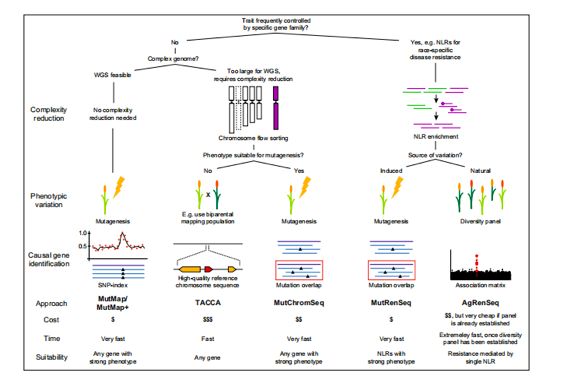
Positional cloning 2.0 怎样避免在基因克隆上花费10年?
就像Xa21的克隆,图位克隆一直是基因定位的有效方法,它的关键在于产生高分辨率的作图群体应用于表型鉴定和基因分型,这对亲本和创建合适的群体要求较高。今天,随着测序技术、芯片技术的发展,开发出“海量”的SNP标记,当分子标记密度不再是克隆基因中的绊脚石,我们得关注其它问题并进行解决。以下总结3点:
❖ 加速基因克隆的方法1-缩短群体构建时长
针对这个问题,作者总结出以下几点:
第一:构建DH系,由单倍体加倍获得的双单倍体,可直接用于育种,极大地缩短育种周期。
第二:Speed breeding,它是一种缩短作物生长周期方法,采用了22:2小时的光:暗循环实现了大麦、小麦、豌豆等一年6代的生长繁殖,对油菜每年可增长至4代。近日,Nature Protocol发表了题为”Speed breeding in growth chambers andglasshouses for cropbreeding and model plant research”的研究论文,作者从光强、湿度、温度及光周期等条件阐述了温室中加速育种的方法,包括小麦、大麦、硬粒小麦、燕麦、芸苔属植物、豌豆、草豌豆、鹰嘴豆、二穗短柄草和藜麦等的快速生长的条件。
将DH系和Speed breeding相结合可大大缩减构建群体的周期。
❖ 加速基因克隆的方法2-候选区段基因组装
一旦将基因定位至一段遗传区间内,此时会涉及到遗传距离向物理距离,CM向bp的转化,这也是图位克隆的限速步骤,在大基因材料中表现的更明显,此前它涉及了多轮BAC文库筛选,例如,小麦中数百个基因抗病基因被定位,而只有几十个被克隆。每个BAC克隆含有100-200 kb的DNA,这样,覆盖小麦基因组需要500,000个BAC克隆,且三个高相似度的基因组为对我们的筛选进行干扰,高质量的参考基因组可部分克服这种困难,仍存在由于BAC克隆所含片段大小的限制,图位克隆已100-200 kb的速度步移着。如何解决这个问题?
2017年Thind提出了‘Targeted chromosome-based cloning via long-range assembly’(TACCA)的方法,利用Illumina测序得到的短片段序列,对其组装,比较片段差异。利用此方法,成功在小麦中克隆了小麦抗叶锈病基因Lr22a。
而Illumina读长短,百迈客引入ONT 测序(OxfordNanopore Technologies,简称ONT)平台,部分项目的结果中,平均reads长度几乎均在20Kb以上(不同样品DNA抽提难易程度不同,会造成一定的影响),最长readsN50高达48.3 Kb,最长Reads更是高达1.29 Mb!这为后期不用组装,直接检测定位区间内的变异提供可能。

❖ 加速基因克隆的方法3-突变体创造
目前,研究单个基因和单个基因效应常用方法有诱导突变,DNA诱变剂包括物理诱变与化学诱变。跳跃基因,T-DNA的发现也为创造突变体提供新的方法。而人工诱变相较于自然突变有如下优点:1、变异丰富,原则上可在基因的任何部位发生突变;2、单基因隐性突变体可直接研究表型与基因型的关系,从而排除背景基因对其影响;3、绝大多数诱变植物与未处理植物之间的表型变化由诱变基因控制。
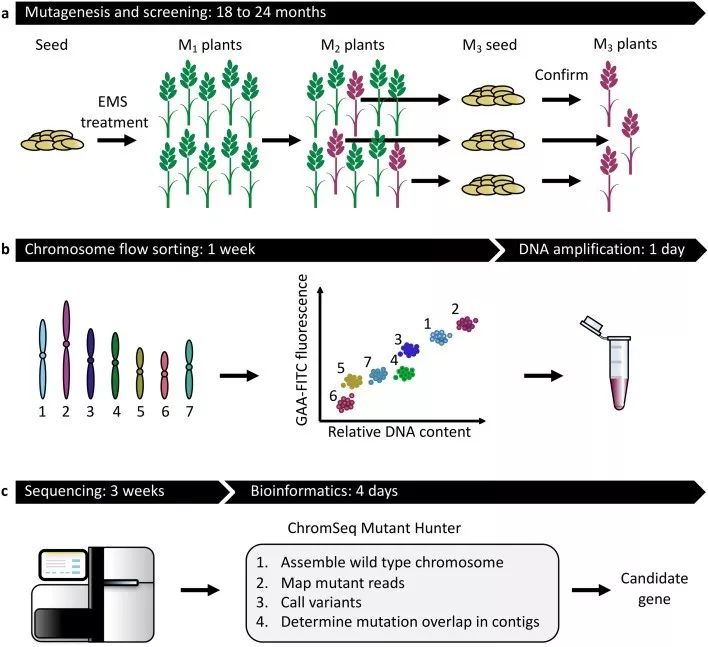
TILLING,即Targeting Induced Local Lesions IN Genomes(定向诱导基因组局部突变技术),是由Steven Henikoff领导的研究小组发展起来的,TILLING技术借助高通量的检测手段,快速有效地从由化学诱变剂(EMS)诱变过的突变群体中鉴定出点突变。目前此技术广泛应用于作物研究中,另外还有基因全基因组重测序的MutMap和MutMap +方法,实现功能基因的快速定位。MutRenSeq和MutChromSeq也应用于突变基因定位,2016年John Innes Centre组织利用EMS诱变产生的小麦突变体,使用液相捕获技术,通过特异性的RNA捕获探针对目标区域进行富集,二代测序后,通过比对野生型和抗性突变体的序列,找到了目标抗性基因。MutChromSeq在此基础上进行改进,利用流式细胞仪技术将含目标基因的单条染色体分离出来,后期结合高通量测序,通过比较突变体及野生型的差异,快速克隆基因,此方法大大降低重测序的难度,减少了其它染色体序列对目标序列的影响。
相关阅读:







 京公网安备 11011302003368号
京公网安备 11011302003368号