BSA(Bulked Segregant Analysis),又称集群分离分析法或混合分组分析法,针对目标性状,选择表型极端差异的亲本构建家系,同时对两亲本和该家系目标性状表型极端的子代分别混合得到的两个混池进行全基因组重测序,检测到的两混池间DNA差异片段即为候选区域,并注释该区域基因,可进一步定位到目标性状相关的基因或标记。利用该方法,可以快速的对单一性状在基因组上进行定位。
随着高通量测序技术的兴起,基于全基因组测序的BSA方法广泛地应用在作物、蔬菜、花卉等物种中,并且成功定位出了许多农艺性状相关的基因。BSA因其具有“快速、准确、性价比高”的优点,受到了广大育种家的青睐。
下面小编为大家分享几篇BSA文章案例,一起来看下BSA分析的应用思路~
案例分享
基于BSA和转录组水稻孕穗期耐寒性候选基因鉴定
研究背景
水稻是全世界最重要的粮食作物之一,同时也是冷敏感作物。孕穗期冷害是影响水稻穗部形态建成和制约水稻高产稳产的主要因素之一。黑龙江省位于中国最北端,是中国的水稻主产区。低温胁迫,特别是孕穗期,严重影响了黑龙江省水稻总产量。因此,水稻抗寒育种是黑龙江省水稻增产的重要策略。由于水稻孕穗期耐冷性遗传机制的复杂性和传统QTL定位方法的局限性。从而导致耐冷遗传研究较为缓慢。因此,发掘孕穗期耐冷主效QTL是挖掘耐冷基因的基础,同时,对分子标记辅助选择在耐冷品种的选育上具有重要指导作用。
研究思路
研究结果
BSA分析
本研究在Longjiang25(耐冷)和Longjiang11(不耐冷)杂交构建的F2群体中(245份材料),挑选50个耐冷和50个不耐冷极端材料分别进行混池测序,亲本各10×,子代分别是67×和80×,进行BSA分析;利用SNP index 和indel index方法进行性状与标记的关联,最终将候选区域定位到6号染色体和9号染色体上的0.82Mb区域内,分析发现这些区域共包含98个注释基因。
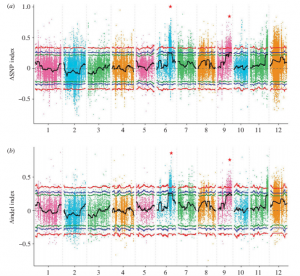
图1 BSA定位结果
转录组分析
作者对低温胁迫(12℃)条件下,对第0、2、4天水稻花序上3.5-4.5mm的幼穗进行取样, 做 RNA-seq分析 ,分析两亲本在低温胁迫条件下,差异基因表达情况。结合BSA关联分析的定位区间,发现候选区域内有50个差异表达基因。通过对差异表达基因进行GO和KEGG富集功能分析结合功能富集分析,确定4个(Os09g0514200、Os09g0516500、Os09g0516600和Os09g0511600)潜在候选基因与耐寒性相关;进一步通过qRT-PCR验证这4个基因的表达情况,结果显示与转录组表达一致。
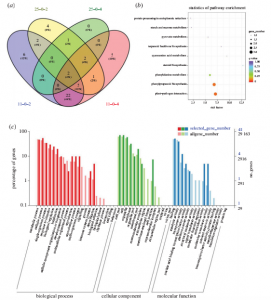
图2 转录组结果
总结
本研究中对水稻耐冷材料和不耐冷材料构建 F2 群体,选择两个极端混池进行全基因组混池测序,同时该研究结合 RNA-seq和基因功能富集分析,确定4个与耐寒性相关的潜在候选基因。
基于高密度遗传图谱和BSA共定位油菜株高相关的候选基因
研究背景
油菜是世界上最重要的油料作物之一,株高是油菜株型的主要决定因素之一,不但与油菜收获指数以及产量密切相关,而且也是影响抗倒伏能力和机械化收获特性的一个重要因素。因此,解析油菜株高遗传结构,挖掘矮秆种质资源和矮秆基因对植物株型的改良和培育油菜理想株型品种具有重要的理论与现实意义。
研究思路

研究结果
BSA分析
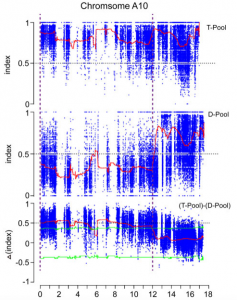
本研究在ZS11-HP(高)×sdw-e(矮,自然变异)构建的F2群体中,分别挑选20株极高和极矮的材料进行混池构建,进行重测序,亲本测序深度为69.96×和69.62×,子代测序深度为32.82×和33.47×,进行BSA分析,通过SNP-index的方法定位到A10染色体上0 Mb-5.5 Mb, 6.0 Mb-12.0 Mb(11.5Mb大小)的显著区间。
图1 BSA分析结果
高密度遗传图谱构建与QTL分析
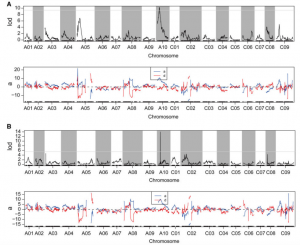
用于高密度遗传图谱构建的群体是同一个亲本构建的包含200份单株的F2群体,对子代测序7.67×,依据基因型开发出4323个bin标记,利用Highmap构建了油菜的19条连锁群,总遗传距离为2026.52cM;又利用R/qtl的区间作图法(IM)和复合区间作图法(CIM)定位油菜株高性状的QTL位点,定位到3个主效QTL,其中qPHA10与BSA定位结果高度一致,显示出定位的可靠性。

图2 油菜高密度遗传图谱
图3 基于 IM和CIM的QTL定位结果
转录组分析
为了进一步挖掘候选基因,本研究进行了ZS11-HP和sdw-e中SAM组织的转录组分析。根据定位的QTL区间,在稳定的QTL的区间内共鉴定出11个上调和14个下调的候选基因。进一步基于两个亲本的变异信息和RNA-seq,确定BnaA10g08290D、BnaA10g09290D和BnaA10g08230D可能是本研究的候选基因。后续又结合qRT-PCR分析,验证了差异基因表达。
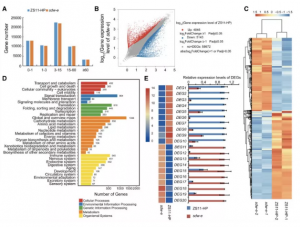
图4 RNA-seq分析和差异表达基因KEGG富集分析
总结
本研究中基于两个F2群体分别进行BSA分析和高密度遗传图谱构建,采用BSA和遗传图谱共定位的方法对油菜株高相关的QTL进行定位,在两个F2群体中定位到油菜株高相关的主效QTL qPHA10。
基于BSA和GWAS分析揭示大豆花叶病毒G2和G3株的抗性机制
研究背景
大豆是重要油料作物,生长发育过程受各种不利环境及病虫害影响。大豆花叶病毒病是多数由大豆花叶病毒(Soybean mosaic virus,SMV)侵染引起的病毒性病害,全球范围内分布较广、具有破坏性的病毒性疾病之一,其对大豆植株危害严重,传播范围广。挖掘抗病基因、培育抗病品种是抵抗病害和保证大豆产量的有效途径。
研究思路

研究结果
GWAS分析大豆对SMV菌株G2的抗性
本研究对352份栽培大豆种质材料进行SLAF测序,共开发出26,143 SNPs标记,对其进行全基因组关联分析,在13号染色体上鉴定到与SMV菌株G2抗性相关的显著SNP位点,其中SNP rs29384570解释了16.58%的表型变异;作者将与rs29384570(Chr. 13: 29384570 bp)相关的区域确定为SMV抗性的主要基因座,命名为Rsvg2。
连锁分析
为了进一步验证Rsvg2的准确性,作者基于130份RIL群体材料构建极端混池,采用SLAF-seq与BSA分析结合的策略,进行BSA分析,结果发现与SMV抗性相关基因组簇分布在13号染色体上的3个相邻基因区域(28Mb-32Mb);第2个区域与GWAS关联的区域重叠,表明Rsvg2是一个稳定的位点,可以潜在控制大豆对SMV G2和G3的抗性,并且抗性基因更可能在第2个热点区域。利用基于130个RIL群体材料中的40个多态SSR和SNP标记的连锁图,进一步将Rsvg2基因座定位在SNP 9和SNP 11之间的区间中,该区间包含30个候选基因。
接着,作者又利用一个超大F2分离群体,在Rsvg2的间区和侧翼区,利用55个具有多态性的SSR标记与F2中抗性植株的表型数据进行关联分析,将Rsvg2定位至2.83 kb区间内,该区间包含一个编码SOT(磺基转移酶)的GmST1基因。作者将GmST1确定为Rsvg2位点的候选基因。
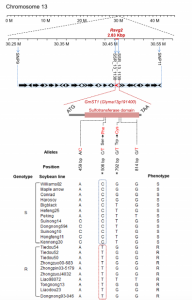
图1 Rsvg2的精细定位
转基因验证
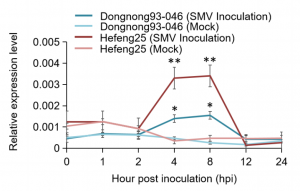
作者进一步通过表达分析、遗传转化分析等方法验证GmST1基因对大豆花叶病毒的抗性,通过qRT-PCR和RNA-seq对SMV抗性株系Dongnong93-046和SMV-感性株系Hefeng25的不同组织进行分析,分析结果表明,GmST1基因受SMV感染调控,且该基因在抗SMV和易感SMV大豆品种之间存在差异表达。通过转基因过表达遗传转化实验发现,GmST1的RSVG2-R等位基因提升了大豆对SMV G2和G3菌株的抗性。最终发现编码磺基转移酶的GmST1基因, 赋予大豆对大豆花叶病毒G2和G3株的抗性。
图2 GmST1基因在不同抗性材料中的相对表达量

图3 转基因植株表型
总结
本研究结合GWAS,BSA,QTLs等群体基因定位研究方法,对大豆植株对大豆花叶病毒G2和G3株的抗性基因座进行精细定位,进一步通过转基因过表达,转录组等方法进行验证,最终发现GmST1基因, 其编码磺基转移酶, 赋予大豆对大豆花叶病毒G2和G3株的抗性。
尾声
通过以上文献我们可以看出BSA在遗传群体基因定位中发挥重要作用,可通过BSA分析对极端性状进化基因定位,进一步与遗传定位结果进行互相验证。然而,为了能让广大科研工作者能够自主便捷的通过BSA进行相关材料的群体遗传学研究,百迈客云自上线以来,百迈客群体研发团队进一步全情打造群体云分析平台,耗时一年半时间重磅推出的BSA云分析平台,可实现极速标准分析、个性化分析、实时查看的优势,另外还可大大降低数据分析成本,让科研更加高效!
在此年末促销之际,百迈客特推出做BSA项目,送BSA云平台分析权限,欢迎垂询!
参考文献
[1] Guo Z, Cai L, Chen Z, et al. Identification of candidate genes controlling chilling tolerance of rice in the cold region at the booting stage by BSA-Seq and RNA-Seq. R Soc Open Sci. 2020;7(11):201081.
[2] Dong Z, Alam MK, Xie M, et al. Mapping of a major QTL controlling plant height using a high-density genetic map and QTL-seq methods based on whole-genome resequencing in Brassica napus. G3 (Bethesda). 2021;jkab118.
[3] Zhao X, Jing Y, Luo Z, et al. GmST1, which encodes a sulfotransferase, confers resistance to soybean mosaic virus strains G2 and G3. Plant Cell Environ. 2021;10.1111/pce.14066.









 京公网安备 11011302003368号
京公网安备 11011302003368号