研究背景
单个基因不同转录本isoform产生的蛋白质具有不同的生物特性,包括稳定性、细胞内定位、酶活性和翻译后修饰。Isoform是可选择性转录起始位点(transcription start sites,TSS)、转录终点(transcription end sites,TES)和可变剪接事件等的产物。据预测,大部分人类基因存在可变剪接。可变剪接突变与人类遗传病和肿瘤均密切相关。故而,不仅需要在基因水平鉴定转录组多样性,也需要在转录本表达水平分析细胞真正的转录多样性。二代转录组测序弊端:目前基于二代平台的短读长RNAseq方法在识别复杂转录本isoform方面存在固有限制,因为它们不能测序全长转录本。相反,转录本被片段化以进行测序,其产生的单个短reads无法跨越整个转录本。算法工具可用于从这些reads中组装完整的转录本,但不同的组装算法可能会导致相互矛盾的结果,整体组装质量良莠不齐。为了克服二代短读长RNAseq的这种限制,出现了基于三代测序平台的全长转录本测序,比如ONT平台全长转录组测序技术。研究表明,在看似同质的细胞群体中各个细胞在基因表达方面可能不同。细胞间异质性使免疫细胞成为深入分析转录多样性的靶标。研究目的:通过使用ONT技术对全长cDNA分子进行测序,探究小鼠B1a细胞的单细胞转录组多样性。
研究方法
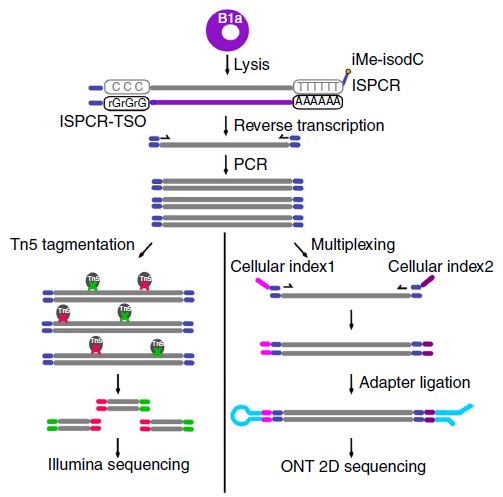
- 小鼠B1a细胞分选:野生C57Bl/6小鼠腹腔灌洗收集细胞,流式分选Ter119−CD3−CD4−CD8−Gr1−B220+IgM+CD11b−CD5+ B1a细胞。(注:B细胞根据其发育来源分为B1细胞和B2细胞,根据是否表达CD5分子B1细胞又分为B1a和B1b细胞2种亚型,其中B1a细胞为CD5+ B细胞,而B1b和B2不表达CD5分子。)
- Smartseq2单细胞全长mRNA扩增合成cDNA
- 7个B1a细胞全长cDNA分别进行二代Illumina转录组测序(73,086-351,876 150 bp reads/细胞)和三代ONT全长转录组测序(R 7.3 17,749-52,696/R9.4 57,874-128,726 ONT reads/细胞),二者间进行比较。
人工合成标准品 Spike-in RNA Variant Control Mixes (SIRVs, Lexogen,根据7个人类基因结构设计而成的,其中每个基因结构有6-18种转录本变异,因此总共有69种转录本,这些转录本全面的解决了可变剪接、可变转录起始点和终止位点、重叠基因和反义转录问题),分别进行二代Illumina转录组测序和三代ONT全长转录组测序,二者间进行比较。
研究结果
1、B1a细胞基因表达定量比较
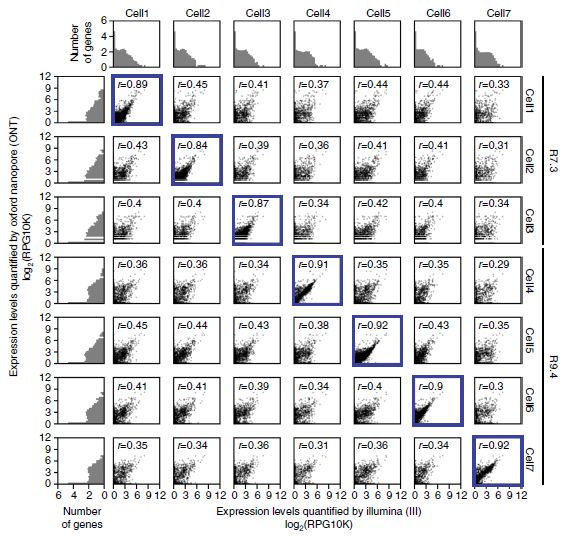
比较相同细胞的Illumina和ONT RNAseq基因表达定量结果,二者间具有高相关性(ONT R7.3芯片的Pearsonr相关系数≥0.84-0.89和升级版R9.4芯片为0.9-0.92),证实ONT RNAseq方法可复现Illumina基因表达定量。比较不同细胞中的Illumina和ONT RNAseq基因表达定量数据显示,Pearsonr≤0.45的低相关性,表明ONT RNAseq可以鉴定不同细胞间表达差异。
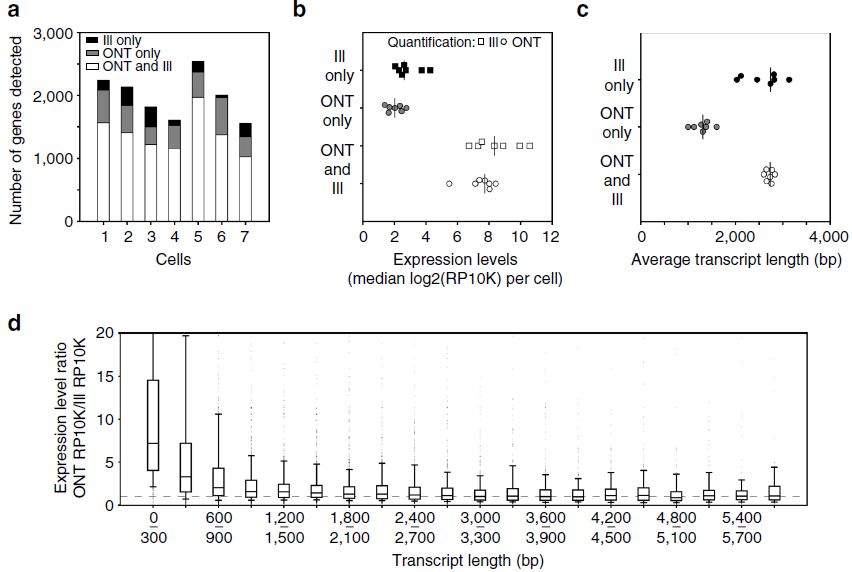
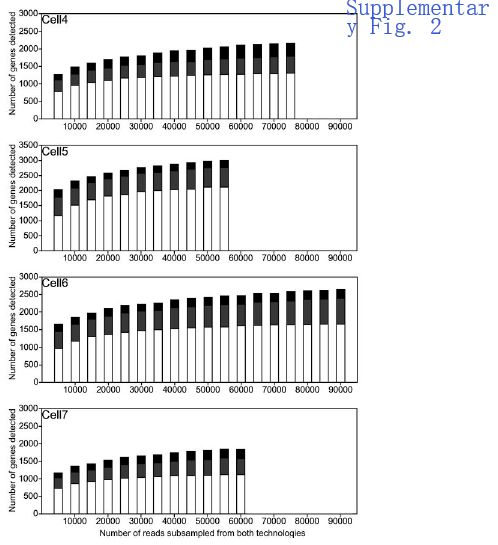
即使产生相对较少的reads数,ONT RNAseq基因表达定量也检测到了绝大多数Illumina RNAseq检测到的基因(下图a)。此外,7个细胞中的5个,基因表达检测已达到饱和(下图S2)。ONT或Illumina RNAseq单独检测到的基因表达水平较低,表明这些基因的表达水平接近两种技术的检测下限(下图b)。还观察到ONT RNAseq单独检测的基因由较短的转录本组成(下图c)。此外,长度<600bp并且通过ONT和Illumina RNAseq检测的基因在Illumina RNAseq数据中具有相对较低的表达水平(下图d)。虽然这与在基于Tn5的Illumina文库制备中强烈选择的较短转录本一致,但不能排除ONT RNAseq可能偏向于较短的转录本。为排除这种可能性,作者进一步选择合成转录本混合物SIRVs,分析转录本长度是否对ONT RNAseq表达定量有影响。
2、SIRVs合成转录本混合物2种平台比较
SIRV为已知长度、结构和序列的人工合成的Spike-in RNA对照混合物。当扩增单细胞级痕量RNA时,较低浓度组中的转录本drop-out(很多未检出表达),并且转录本定量显示每个浓度组内的变化(下图e,横坐标为4个不同浓度分组)。然而,重要的是,定量不受转录本长度的影响,除了短于500bp的转录本(下图f)。通常,ONT RNAseq定量与Spike-in 转录物浓度一致,转录本定量的组内变异在重复样本之间是可重复的(下图g)。这种组内变异可能是由于初始转录水平、系统扩增偏差或数据分析偏差导致。分析这些不同浓度的合成转录本使作者排除了ONT RNAseq有利于较短转录本定量的可能性,可对长度为500-2,500 bp的SIRV转录本进行大规模无偏倚定量。
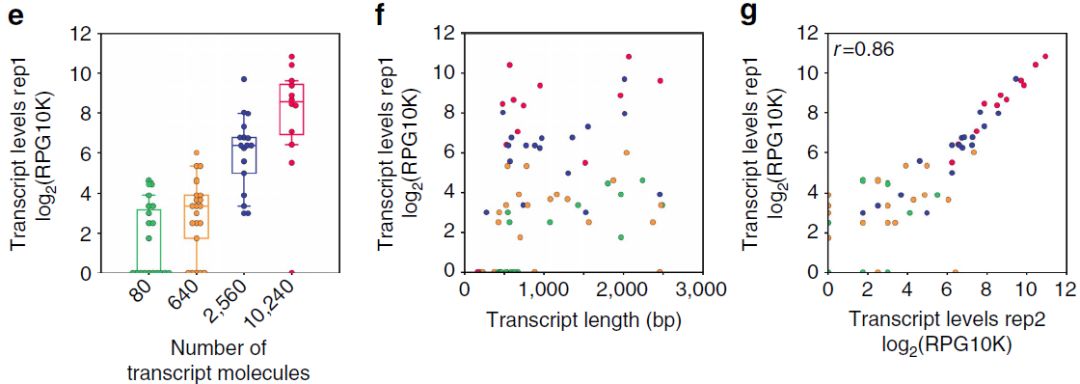
3、SIRVs isoform鉴定及定量
接下来作者评估了ONT全长转录组测序是否适用于鉴定SIRVs不同isoform及isoforms表达定量。利用Mandalorion pipeline对TSS、TES和剪切位点进行分类。作者检测到20个TSS位点和24个TES位点,它们都与实际的TSS和TES直接重叠,并且在SIRV转录本注释中存在的38个(/57个)实际TSS和41个(/59个)实际TES的60bp内。
此外,在SIRV基因组注释中检测到76个(/89个)5’剪接位点和73个(/93个)3’剪接位点。通过分析ONT reads实际剪接模式,作者检测到11个(/12个)备选3’剪接位点和12个(/14个)备选5’剪接位点,以及12个(/12个)内含子保留事件。
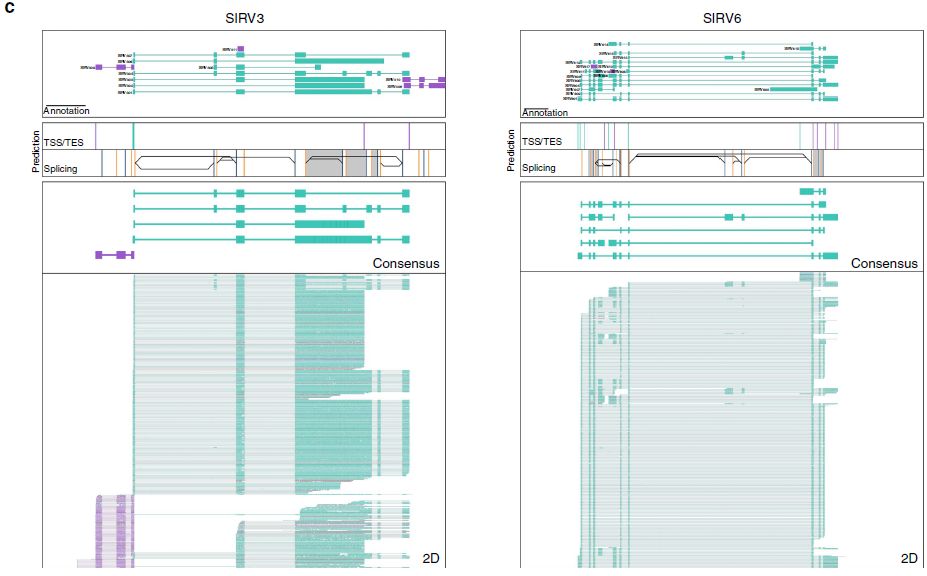
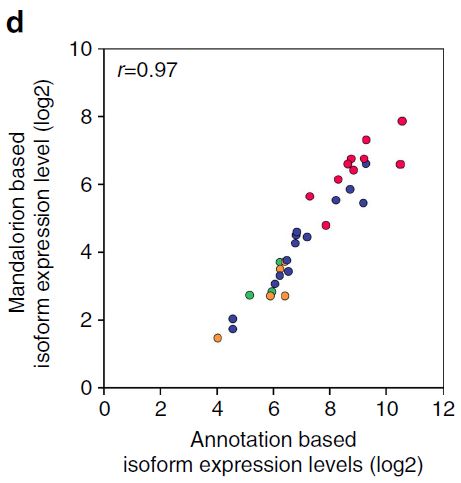
根据其TSS/TES和可变剪接位点的使用将ONT reads分类为isoform组,并生成一致性序列,共计33个一致性序列,与其对应的SIRV转录本之间具有97.8-100%相似性,且方向一致。26个一致性序列匹配2个高丰度组中存在的29个SIRV转录本之一(下图c)。不依赖于基因组注释的转录本isoform Mandalorion分类定量与reads直接与转录组比对得到的定量结果之间的高度相关性(下图d)。
4、鉴定单个B1a细胞isoform特征
通过对ONT全长转录组数据分析,鉴定到4234个TSS和3883个TES,仅有2476个TSS和2448个TES与小鼠基因组的Gencode数据库(vM10)中存在的TSS或TES重叠。(下图a)为了确定TSS和TES鉴定的z确性,作者分析了Fantom5数据库中 CAGE peak和polyA信号富集,Fantom5 CAGE峰来源于转录本5’末端的捕获和测序,因此应在TSS中富集。实际上,与TES(49/3883或1.3%)相比,高比例的注释(2356/2476或95%)和未注释(1052/1799或58%)TSS与高得分的Fantom5 CAGE峰重叠(下图b)。相反,注释和未注释的TES都高度富集polyA信号,而TSS则没有(下图c)。如预期的那样,大多数基因恰好包含一个TSS和一个TES。然而,696个基因含有1个以上的TSS或TES,表明存在一种以上的isoform(下图d)。总之,单个细胞ONT RNA-seq成功鉴定了数千个未注释的TSS和TES以及数百个具有差异TSS/TES使用的基因。
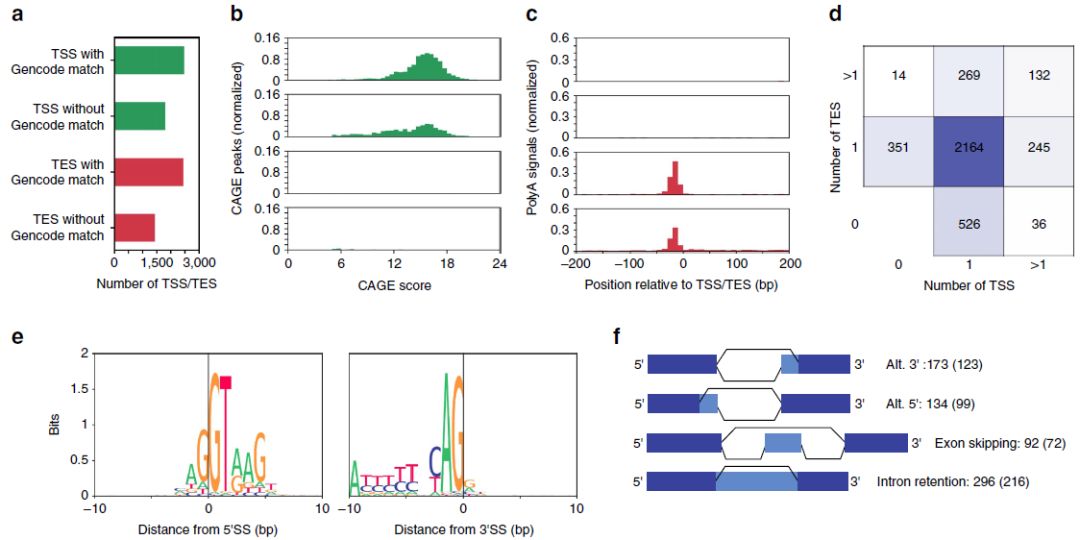
总共鉴定到24,887个5’剪接位点(SS)和24,756个3’剪接位点。绝大多数这些剪接位点由Illumina junction reads或GENCODE注释支持。24,298(97.6%)个5’SS和24,220(97.8%)个3’SS分别与GENCODE注释匹配。在与GENCODE注释不匹配的589个5’SS和536个3’SS中,分别有250(42.4%)个5’SS和216(40.2%)个3’SS由在Illumina junction reads支持。就算假设所有无GENCODE注释或Illumina reads支持的剪接位点都是假的(显然这是不可能的),该方法的错误发现率仅为1.3%(659/49,643)。ONT RNAseq在确定√确剪接位点方面相对成功(上图e为剪接位点碱基上下文context)。作者发现了296个内含子保留事件,134个可选的5’剪接位点和173个可选的3’剪接位点组合。大多数这些事件也在Illumina reads中观察到,illumina reads支持216个(/296个)内含子保留事件,99个(/134个)可选5’剪接位点,123个(/173个)可选3’剪接位点和72个(/92个)外显子跳跃事件(上图f)。
5、鉴定B1a细胞复杂isoform
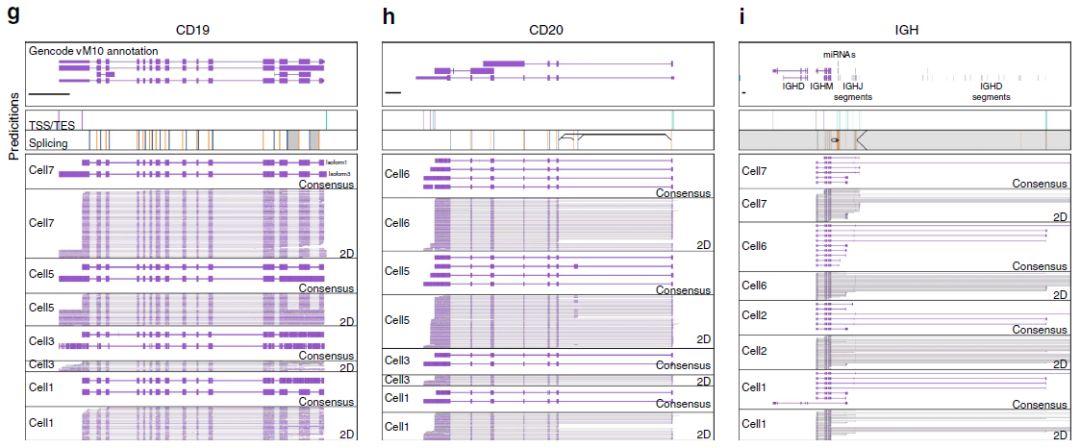
表达复杂isoform的基因定义为:含有可变TSS/TES和可变剪接位点的基因。共计鉴定了169种表达复杂isoform的基因。其中55个基因在细胞之间存在高度显著差异isoform使用,包括B细胞特异性表面受体CD19和CD20,抗体重链基因座(IGH)(下图g-i),CD37(下图CD37),以及CD2和CD79b,以及CD45。各个B1a细胞中,来自CD19的同种型显示出可变TSS和内含子保留事件的组合。另一方面,来自CD20的同种型显示出可选择性TES的组合,以及包括先前未注释外显子的外显子跳跃事件。IGH基因座更复杂,具有包含VDJ重组和IGHM恒定区外显子的典型isoform。观察到了含有IGHM恒定区外显子的isoform,但是源自(1)流产性DJ重组(2)I-外显子(3)IGHM转换区miRNA基因座(4)J-区段。最后,细胞1中的一种isoform来自IGHM I-外显子,但含有IGHD恒定区外显子。虽然之前已观察到IGH isoform多样性并且长期以来已知其参与类别转换,但ONT RNAseq在单细胞水平上测序全长cDNA的能力确实突出并证实了 IGH基因座特殊的转录多样性。
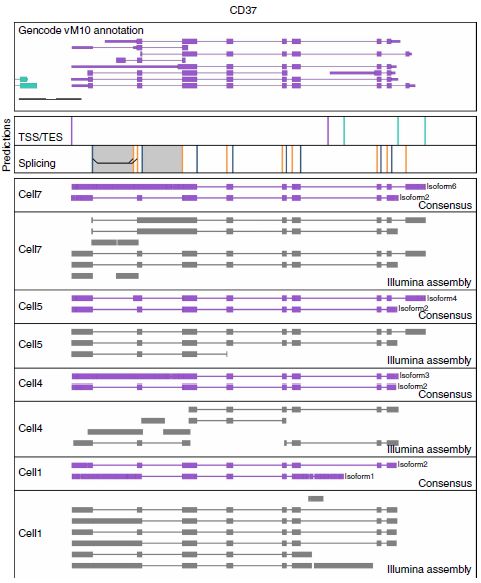
ONT全长转录组测序优于Illumina数据组装转录本isoform的优势在于从5’端到3’端测序整个cDNA分子的能力。虽然如果基因座仅表达单个isoform,使用Trinity组装Illumina数据可能会成功,但它似乎很难分析包含多个远距离替代特征的基因座的多种isoform。例如,ONT RNAseq在所分析的各个细胞中鉴定了CD37基因的几种不同isoform(上图CD37)。在大多数情况下,从单个细胞组装Illumina数据时,Trinity无法形成完整的重叠群或产生ONT RNAseq未检测到的重叠群。因此,CD37基因及其isoform鉴定突出了ONT RNAseq方法的优势,以确定复杂isoform多样性,超出了短reads技术的可能性。
小结
参考文献
Byrne, A., Beaudin, A. E., Olsen, H. E., Jain, M., Cole, C., Palmer, T., … Vollmers, C. (2017). Nanopore long-read RNAseq reveals widespread transcriptional variation among the surface receptors of individual B cells. Nature communications, 8, 16027. doi:10.1038/ncomms16027如果您的项目有任何,欢迎点击下方按钮咨询我们。







 京公网安备 11011302003368号
京公网安备 11011302003368号