之前小编已经对ATAC测序做了简单介绍(ATAC),首先我们简单回顾一下~~
ATAC测序,全称为 Assay for Transposase Accessible Chromatin using sequencing,是一种运用测序手段研究转座酶可接近的染色质的实验,染色质以紧密缠绕的形式存在,当基因要进行转录的时候,紧密缠绕的染色质则会松散开来,这时我们利用转座酶对松散开的区域进行酶切,从而对细胞中正在转录中的区域进行测序。
从ATAC所捕获的区域可以了解到,做ATAC测序主要的目的呢是研究正在转录的那部分DNA序列,测序的结果大部分是转录本区域的上下游的DNA序列,得到这些序列我们就可以对一些基因上的顺式调控元件进行研究,最常见的不外乎去研究转录因子结合的启动子区域。
那么做完ATAC测序我们还可以做哪些事情呢?
1.ATAC测序和转录组数据联合分析
ATAC测序结果,研究了该时空条件下发生转录的基因以及顺势调控元件的一些序列,那么我们就可以对这些基因进行分析。联合转录组测序结果,看ATAC上测到的一些丰度高的DNA序列区域,是否对应的转录本表达量也有增加,也可以找到对应的转录本相关基因的上游调控序列,从而整体分析从DNA到RNA的转录过程,进而对基因功能分析,再结合实验表型进行讨论,从而理清楚表观调控-表达-功能-表型这样一个过程[1]。
2.ATAC测序和ChIP-seq联合分析
在ChIP实验中,好多时候我们是用以研究转录因子所调控的那些基因的,那么我们有了ATAC测序之后是否就不需要做ChIP实验了呢?并不是~~就像虽然做转录组测序也可以知道基因表达量的上下调情况,但是我们仍然需要通过qPCR来进行分子生物学验证一样,ATAC测序之后也需要做ChIP-seq来做进一步的验证,通过ChIP的测序结果,来进一步对ATAC所预测到的一些转录因子结合区域进行验证[2~4]。
那么ATAC测序在动植物中的技术关键是什么呢?
ATAC测序和转录组一样,也是有时空特异性,但是由于ATAC测序建库的时候需要对细胞进行裂解提取细胞核,再通过转座酶对细胞核中正在转录组的染色体序列进行酶切,那么我们对实验处理中的细胞活性就有一定的要求—不能用冻存后的细胞来建库;另外,由于转座酶对DNA序列进行酶切的时候,只要是转座酶可以接近的区域都可以进行酶切,这就造成了测序数据中有好大一部分是线粒体的DNA序列,这些DNA我们认为是污染的部分,会尽量用探针进行除去,但是探针去除的效率也有限。如在小鼠胚胎不同发育时期的ATAC测序实验中,作者对探针去除线粒体DNA的效率进行了鉴定[2],所测结果如图1,可见核DNA最多的时候占比也仅仅有50%,那么相比较而言线粒体DNA的去除也就相当重要了;而植物细胞,由于其细胞壁结构不能直接通过酶对细胞进行裂解,并且测序结果中有好大一部分是叶绿体DNA污染,因此在做植物的ATAC测序时,则需要将植物细胞制备成原生质体,并且用梯度离心法来收集细胞核后再进行建库[5]。不管是线粒体DNA还是叶绿体DNA的污染,都可以在建库之后进行少量数据测序,通过简单的比对分析,来大概估测文库中的污染占比,从而决定是否正式进行测序。
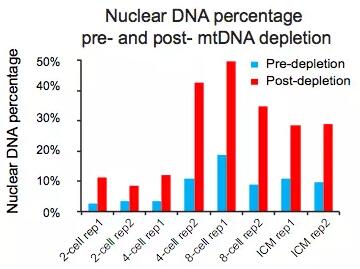
图1
参考文献:
[1]Ackermann A M, Wang Z, Jonathan S, et al. Integration of ATAC-seq and RNA-seq identifies human alpha cell and beta cell signature genes:[J]. Molecular Metabolism, 2016, 5(3):233-244.
[2]Wu J, Huang B, Chen H, et al. The landscape of accessible chromatin in mammalian preimplantation embryos.[J]. Nature, 2016, 534(7609):652.
[3]Denny S K, Yang D,Chuang C H, et al. Nfib Promotes Metastasis through a Widespread Increase in Chromatin Accessibility.[J]. Cell, 2016, 166(2):328-342.
[4]Qu, Kun, Zaba, et al. Individuality and Variation of Personal Regulomes in Primary Human T Cells[J]. Cell Systems, 2015, 1(1):51.
[5]Lu Z, Hofmeister B T, Vollmers C, et al. Combining ATAC-seq with nuclei sorting for discovery of cis-regulatory regions in plant genomes[J]. Nucleic Acids Research, 2016, 45(6).
[6]Gehrke A R, Schneider I, De l C E, et al. Deep conservation of wrist and digit enhancers in fish.[J]. Proc Natl Acad Sci U S A, 2015, 112(3):803-808.
[7]Acemel R D, Tena J J, Irastorzaazcarate I, et al. A single three-dimensional chromatin compartment in amphioxus indicates a stepwise evolution of vertebrate Hox bimodal regulation[J]. Nature Genetics, 2016, 48(3):336-341.
[8]Kaufman C K, Mosimann C, Fan Z P, et al. A zebrafish melanoma model reveals emergence of neural crest identity during melanoma initiation.[J]. Science, 2016, 351(6272):aad2197.
[9]Blythe S A, Wieschaus E F. Establishment and maintenance of heritable chromatin structure during early Drosophila embryogenesis[J]. Elife, 2016, 5.
[10]Lu Z, Hofmeister B T, Vollmers C, et al. Combining ATAC-seq with nuclei sorting for discovery of cis-regulatory regions in plant genomes[J]. Nucleic Acids Research, 2016, 45(6).
[11]Buenrostro J D, Wu B, Litzenburger U M, et al. Single-cell chromatin accessibility reveals principles of regulatory variation[J]. Nature, 2015, 523(7561):486-490.








 京公网安备 11011302003368号
京公网安备 11011302003368号