哒哒哒~~~新年刚过去了20天,我们又有项目文章见刊啦…….1月16日,由大连海洋大学和百迈客合作的“SLAF-based high-density genetic map construction and QTL mapping for major economic traits in sea urchin Strongylocentrotus intermedius”在线发表在《Scientific Reports》上,具体什么事呢?图谱君这就为您解析…….

海胆是海洋里一种古老的生物,与海星、海参是近亲,据科学考证,它在地球上已有上亿年的生存史。海胆生殖腺占海胆全重的8%-15%,其所含有二十碳烯酸占总脂肪酸的30%以上,可有效预防心血管疾病。本文利用SLAF测序技术,构建了高密度遗传图谱,对海胆经济性状相关QTL定位,开发分子标记,用于后续分子标记辅助育种。
1.材料与方法
亲本:日本自繁的中间球海胆二代雄性个体(♂);中国旅顺龙王塘繁育五代以上的雌性个体(♀)
作图群体:150个F1个体
测序:Illumina HiSeq 2500 SLAF测序
构图软件:HighMap
QTL定位:MapQTL V5.0
SNP标记检测:高分辨熔解曲线(HRM)
2.结果与分析
研究结果:
1.SLAF测序与基因分型
选择RsaI和HaeIII酶对基因组DNA酶切,构建SLAF测序文库。Illumina测序总计得到533.05 M 数据,Q30达到89.03%,GC含量为37.39%。2个亲本各分别得到13355278和9550588条reads,F1后代平均产生2629599条reads。
生物信息学分析,父本产生249076个SLAF标签,母本产生279416个SLAF标签,平均深度分别为32.62×和20.53×。对于F1作图群体,每个个体平均产生190361个SLAF标签,平均深度为8.80×。其中62772个多态性SLAF标签用于基因分型(图1)。除aa×bb类36647个SLAF标记被用于图谱构建,适合F1群体的后期连锁图谱构建,有效多态性率为7.99%。
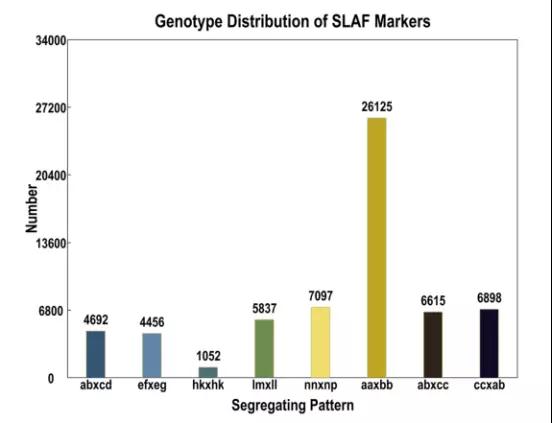
图1 标记分型
2.图谱构建
经过滤,得到可用于作图的 SLAF 标签 4678 个,通过两两标签间计算重组率和 MLOD 值,根据 MLOD 值将标签分为 21 个连锁群,最终确定上图标记共 4387 个,雄性图谱全长3116.10 cM,共983个标记,平均图距3.24 cM。雌性图谱全长3537.39cM,共3465个标记,平均图距为1.03cM。整合图谱全长1907.21 cM,平均图距为0.44 cM(图2)。上图率为 93.78%,上图标签偏分离比例 2.52%。
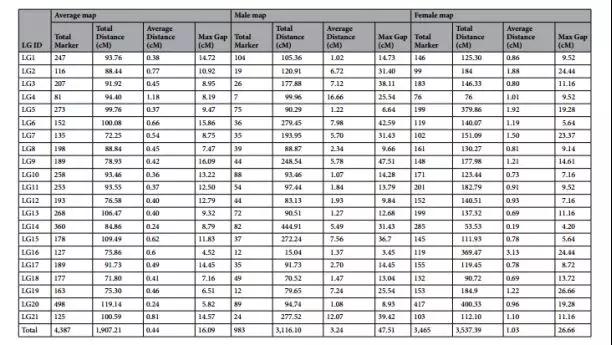
图2 图谱信息
3.QTL定位
在已构建的中间球海胆高密度遗传图谱基础上,结合中间球海胆 21 个连锁群的图谱和分型数据以及性状表型值进行性状关联分析。采用MapQTL 5.0 软件的区间作图(IM)算法分别定位中间球海胆生长性状(壳径、壳高、体重、口器重、性腺湿重、干性腺率、亮黄色差、亮橙黄色差)相关的 QTL。共鉴定出33个QTL位点,分别位于13个不同的连锁群。(LG1,LG2,LG3,LG4,LG5,LG6,LG7,LG10,LG12,LG14,LG15,LG17和LG20)。在LG6上,只检测到壳高相关的QTL,可解释17.80%的表型变异。口器重量QTL位于五个不同的LGs(LG1,LG2,LG3,LG7和LG17),最大的表型变异率解释达到18.60%。壳径相关QTL位于LG6,LG7,LG10,LG12,LG15和LG20,最大可解释18.10%的表型变异率。同样,在LG15上检测到体重和性腺湿重主效的QTL,分别解释了20.4%和37.2%的表型变异。所有的QTL都表明,LG15定位的频率最多,对表型的贡献率都很大,说明LG15存在控制中间球海胆生长性状的主效 QTL 位点,为后期研究的重点。
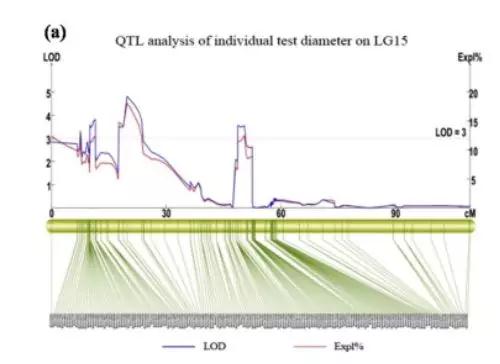
图3 QTL定位
4. HRM分析
HRM是一种快速,高通量和有效的鉴定SNP的方法。本文利用HRM技术对3个与生长性状相关的 SNP 标记在 另外3 个家系总共90个个体的分型结果进行遗传与多样性评估,发现SNP-29(marker19255)可较好分型,且壳高、壳径、活体重性状的贡献率均比较大,说明SNP-29与生长相关的性状基因连锁强度较大,因此后期可含有 SNP-29 标记的优势基因型应用于海胆高产分子标记辅助育种。
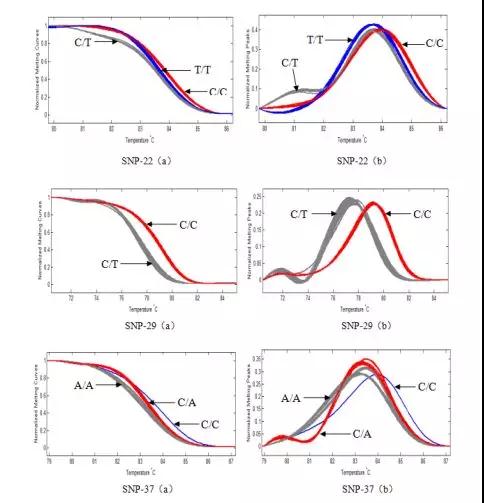
图4 HRM 验证
总结
文章到这里就结束了,总结一下:作者利用SLAF测序技术,构建了一个含有21个连锁群(LG),拥有4387个多态性特异性扩增片段(SLAF)的高密度遗传图谱。基于这个遗传图和表型数据,对8个经济性状进行基因定位。共鉴定出33个潜在的QTL位点,可以解释9.90%–46.30%的表型变异率。且开发了多态性SNP标记,用于后期标记辅助育种。
关于后期,SNP标记应用于后期标记辅助育种的检测手段,图谱君还要唠叨下,除了HRM,还有KASP哦,它是竞争性等位基因特异性PCR,基于引物末端碱基的特异匹配,在广泛的基因组DNA样品中(甚至是一些复杂基因组的DNA样品),来对SNPs和特定位点上的InDels进行精准的双等位基因判断,从而进行基因分型。可应用于QTL精细定位(定位区间内位点的验证,进一步缩小区间范围)、种子质量控制、大样本量的分子标记验证、分子辅助育种、种子资源鉴定等工作,可以在短时间内准确判断分子标记类型。。。。。。。
参考文献
Yaqing C, Jun D, Yuhui, et al. SLAF-based high-density genetic map construction and QTL mapping for major economic traits in sea urchin Strongylocentrotus intermediu [J]. Scientific Reports, 2018
胡思帆丨文案
许语辉 | 审核
图片来自网络,侵删
图谱君重出江湖,附Springer – Aquatic Genomics(432页)
扫描二维码立即下载干货哦~









 京公网安备 11011302003368号
京公网安备 11011302003368号