
期刊名称:Nature Communications
影响因子:16.6
合作单位:西南大学
研究物种:颠茄+曼陀罗
研究方法:基因组学、比较基因组学、转录组学
研究背景
茄科是被子植物中最大的科之一,包括番茄、马铃薯和茄子等重要的经济物种。除了栽培物种外,一些物种因其致命的毒性和药用价值也引起了关注。托品烷生物碱(Tropane Alkaloids,TAs)在茄科植物中广泛分布,而一些重要的药用托品烷生物碱(mecticinal tropane alkaloids,mTAs),如莨菪碱和东莨菪碱,只在少数茄科种属中合成。目前,已经在东莨菪碱生物合成途径解析方面开展了大量的研究工作,然而,关于茄科植物中TAs生物合成途径的演化机制井不清楚。
材料方法
基因组:
曼陀罗:55.88Gb Illumina+107X ONT+244.16 Gb Hi-C;
颠茄:145.27Gb Illumina+29.15X PacBio HiFi+320.93 Gb Hi-C;
转录组:Illumina NextSeq 500 platform;
代谢组:UPLC-MS。TAs测定
研究结果
1.颠茄和曼陀罗基因组组装注释
作者分别利用ONT和PacBio等测序技术对曼陀罗和颠茄进行测序和基因组组装,组装得到颠茄的基因组大小为~1.59Gb,contig N50为3.03Mb,scaffold N50为42.83Mb,其中99.50%的序列锚定在36个假染色体上。曼陀罗基因组大小约为1.84Gb,contig N50大小为105.17Mb,scaffold N50大小约为15609 Mb,97.42%的序列锚定在12个假染色体上。基于从头预测、同源性预测以及转录组数据,分别预测了70209个(颠茄)和32037个(曼陀罗)蛋白质编码基因。BUSCO分析显示两个物种都具有98.9%的完整性。
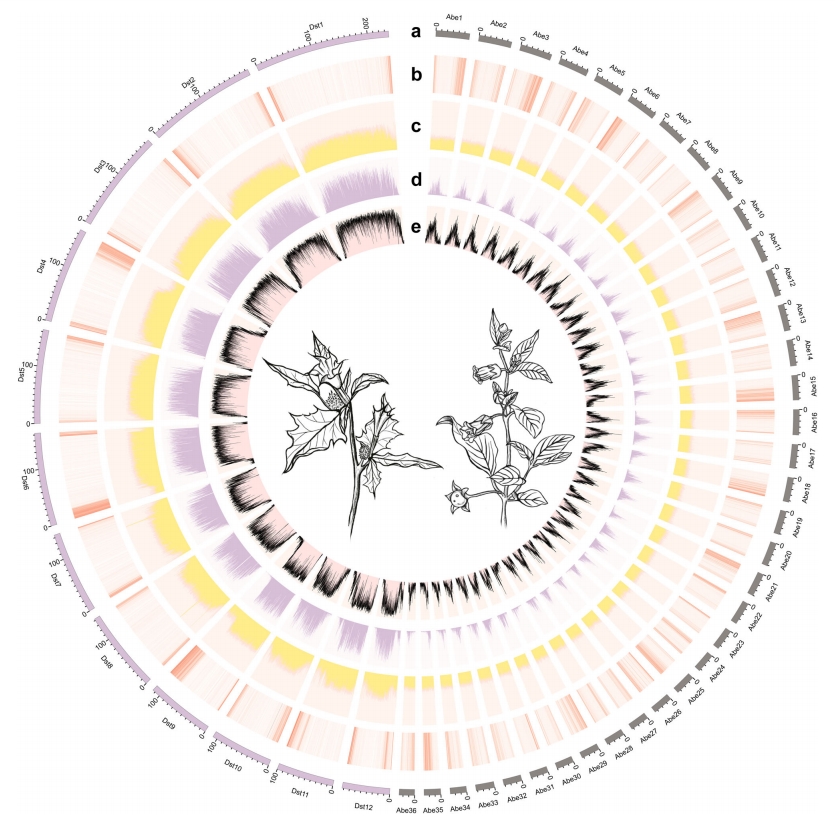
图1-颠茄和曼陀罗基因组特征
2.比较基因组和系统发育分析
作者利用颠茄和曼陀罗,以及8个茄科物种和4种其他被子植物的207个单拷贝基因进行系统发育分析。结果表明:颠茄和枸杞属于一个分支,而曼陀罗、辣椒、茄子、番茄和马铃薯属于另一个分支,估计矮牵牛在约43Mya(33-56Mya)从茄科物种分化而来,曼陀罗在约28Mya(23-37Mya)由顺茄和枸杞组成的分支分化而来,而颠茄在大约24Mya(18-31Mya)从枸杞分化而来。Ks分析显示,茄科物种与其他高等双子叶植物共享较古老的伽马古多倍体事件。对颠茄、曼陀罗和葡萄进行基因组共线性分析,对于葡萄基因组的各个区域,通常在曼陀罗基因组中能鉴定出3个与之匹配的区域,并且在颠茄和曼陀罗之间也有相似的现象。
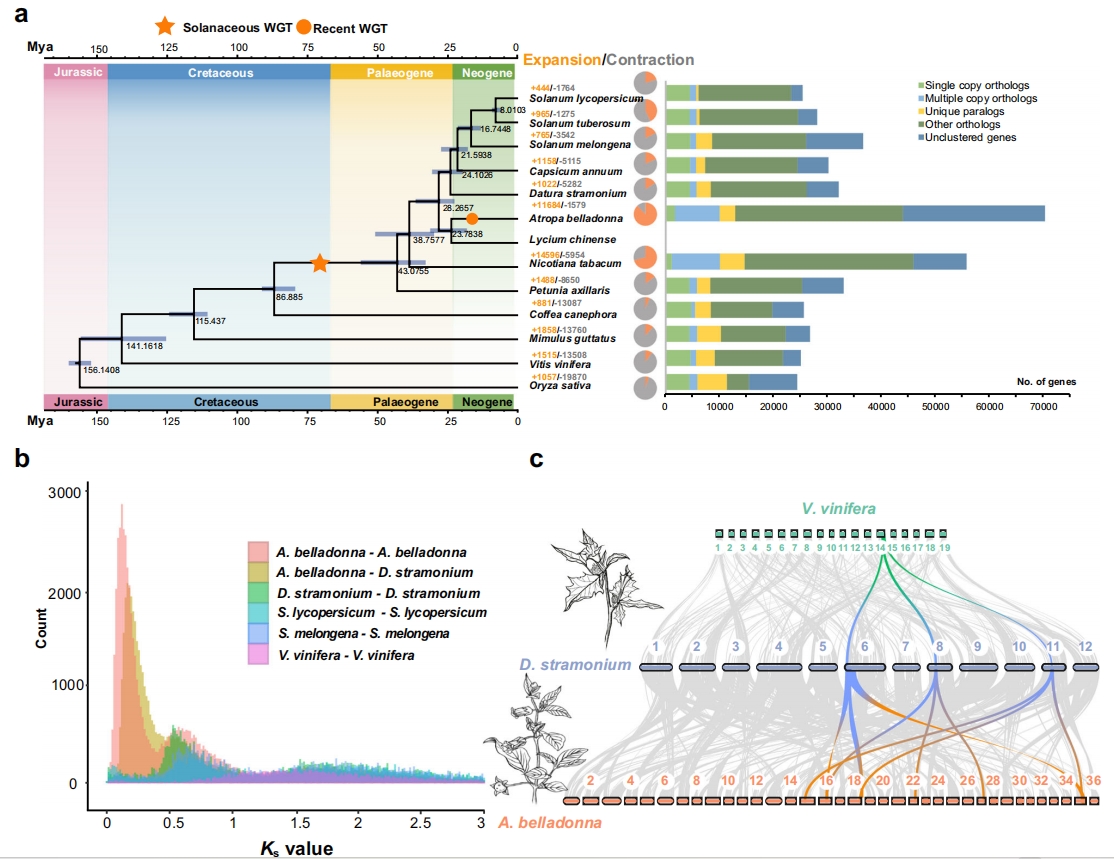
图2-13个物种的系统发育关系和分化时间
3.颠茄和曼陀罗具有保守的mTAs合成途径
通过与颠茄的功能特征基因对比,检索了9个茄科物种中与TAs和mTAs生物合成相关的每个基因家族的同源基因,并构建了各个家族的系统发育树。mTAs生物合成上游途径的大部分基因在茄科中是具有多拷贝且较保守,其系统发育关系与种间关系一致。研究者对含有mTAs的组织进行基因表达检测,发现在生物合成步骤中至少一个mTAs相关基因在颠茄和曼陀罗的根中高度表达。
4.基因簇的进化与TA在茄科植物中的广泛分布有关
鉴于TAs在茄科的广泛分布以及托品烷生物碱生物合成基因的高度保守性,作者进一步检测了这些基因的染色体位置,发现CYP82M3和TRI基因聚类在茄科除烟草以外的茄科代表物种基因组中。CYP82M3催化托品酮的形成,TRI随后将托品酮还原生成托品碱,托品碱是TAs的核心结构。推测该基因簇的进化与托品烷生物碱在该科的广泛分布有关。此外作者还发现,mTAs特异性基因LS和CYP80F1的缺失,决定了mTAs在茄科植物中的不均匀分布。
总 结
本研究基于三代PacBio、Nanopore、Hi-C等技术成功构建了2个具有代表性的产mTAs的物种:颠茄和曼陀罗染色体水平基因组。研究结果表明:它们在茄科中的亲缘关系较远,但这两个物种产生mTAs的生物合成途径是保守的。保守的基因簇和基因重复构成了TAs在该科中广泛分布的基础,导致mTAs的分支基因可能在茄科早期祖先物种中进化,但在大多数谱系中都已丢失,颜茄和曼陀罗是例外。此外,研究还鉴定了一种细胞色素P450,它能将莨若碱修饰成去甲莨菪碱。此研究结果为了解茄科植物中TAs的生物合成提供了基因组学基础,并将有助于通过合成生物学方法进行mTAs的生物技术生产。
Zhang F,Qiu F,Zeng J,et al.Revealing evolution of tropane alkaloid biosynthesis by analyzing twogenomes in the Solanaceae family.Nature Communications.2023.






 京公网安备 11011302003368号
京公网安备 11011302003368号