纳米孔测序是一种由ONT(Oxford Nanopore Technology)研发的单分子测序技术。在转录组测序应用中,相比于传统二代RNA-Seq测序技术,长读长的纳米孔RNA测序可以在无需打断的条件下得到全长序列并进行定量,同时直接RNA测序还可以检测多种碱基修饰,且测序无需扩增,减少了PCR过程引入的碱基偏倚。
ONT测序技术在多个方面具有非常强悍的优势,然而,一份合格的下机数据才是科研成功研究的基础,为保证得到准确的转录组结构分析和定量结果,需要对测序数据进行严格的质控评估。那么我们今天一起学习一下《Summary statistics and QC tutorial》,ONT官方提供的对测序raw data进行全面数据质控的教程。
介绍
此教程适用于指导对单个nanopore测序芯片产出的数据进行评估,评估的主要内容如下所示:
1、测序产出(测序得到多少reads,多大数据量);
2、测序数据的质量和长度分布;
3、如果加入了barcode序列进行混样建库,测序数据在不同样品的分布。
准备
直接到教程的github页面下载或通过git命令下载:
git clone https://github.com/nanoporetech/ont_tutorial_basicqc.git QCTutorial
后续分析会用到下载目录QCTutorial下的以下内容:
1) Nanopore_SumStatQC_Tutorial.Rmd:Rmarkdown文件,说明文档和用于执行分析。
2) RawData/lambda_sequencing_summary.txt.bz2:示例文件,Guppy对测序reads进行碱基识别生成的相关信息文件。
3) RawData/lambda_barcoding_summary.txt.bz2:示例文件,用于区分混样建库时多样品的barcode信息。
4) environment.yaml:指定分析所需软件包及计算环境的文本文档。
5) config.yaml:配置文件,用于指定分析所需的输入。
2、创建Conda环境
为了方便执行分析所需软件包及其依赖的安装及管理,需要安装Conda并创建用于此分析的环境。
1) Conda安装(Python3版本的Miniconda):
wget https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh
bash Miniconda3-latest-Linux-x86_64.sh
bash
2) 创建Conda环境及环境激活(第1步中下载的environmen.yaml用于环境初始化):
创建环境:conda env create –name BasicQC –file environment.yaml
激活环境:source activate BasicQC
分析
进行分析之前需先准备配置文件,通过修改准备步骤下载的config.yaml中相应的参数来完成,需要修改的内容主要有:
| 修改内容 | 内容说明 | 示例 |
|---|---|---|
| inputFile | 碱基识别的统计信息 | sequencing_summary.txt.bz2 |
| barcodeFile | 混样建库的barcode信息 | barcoding_summary.txt.bz2 |
| basecaller | 碱基识别工具 | Guppy 2.1.3 |
| flowcellId | 测序芯片ID | FAK41706 |
注:如为单样品测序无barcode信息,则barcodeFile部分为空。
准备完成后,可以通过命令行启动分析,命令如下:
R –slave -e ‘rmarkdown::render(“Nanopore_SumStatQC_Tutorial.Rmd”, “html_document”)’
如果习惯图形界面操作,也可以通过Rstudio载入Rmarkdown文件执行分析:
结果
上述分析完成后会将分析结果存放至HTML文件,可用浏览器打开Nanopore_SumStatQC_Tutorial.html进行查看。对单个芯片约1M reads分析的部分结果展示如下(结果来自教程,碱基识别使用Guppy 2.1.3,根据识别序列的平均质量值将其分为pass和fail两种,质量值阈值默认为7):
1、总结
展示了数据产出的总体情况(如下图,本分析中碱基识别共产出991,715条序列,14.6G碱基)。

2、质量长度
此部分展示了对识别出的所有序列质量和长度信息的统计结果,包括序列的平均长度,N50和平均质量,序列长度和质量的密度分布等

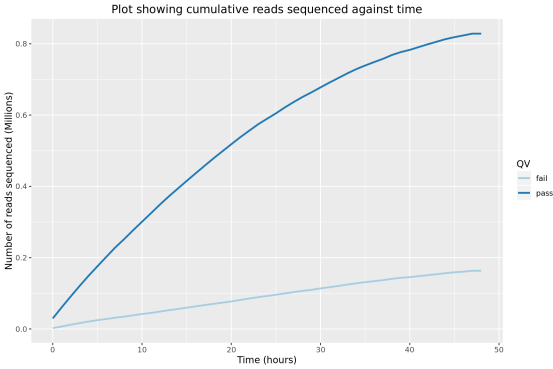
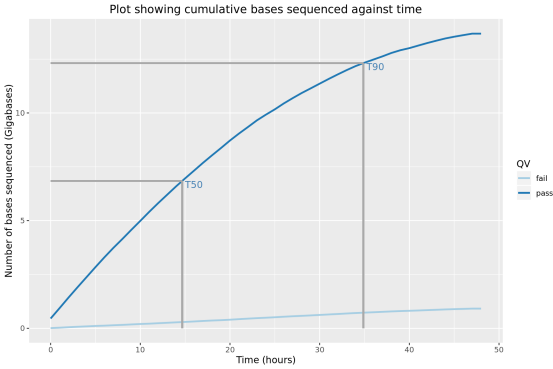
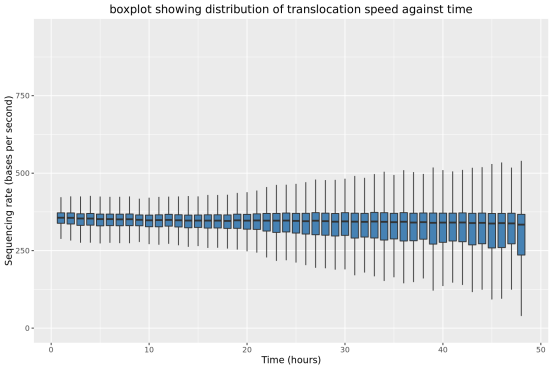
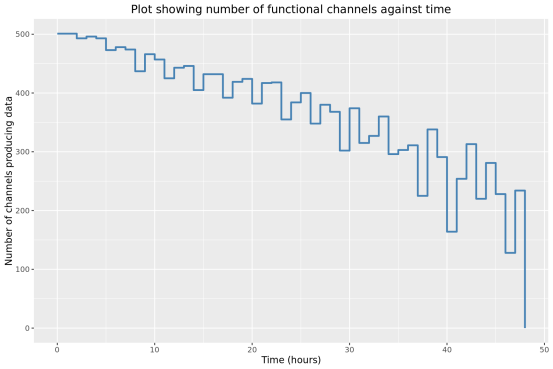
3、测序表现
此部分内容统计了随测序时间变化,测序累计序列个数,碱基个数,测序速度和有效工作纳米孔数等指标的变化情况。
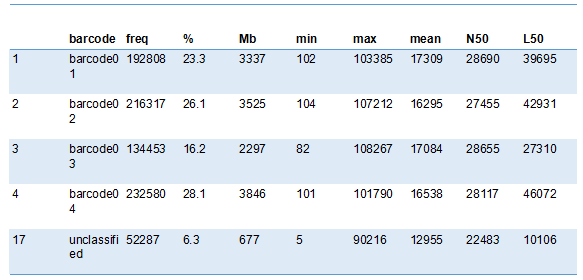
4、区分混样
在加入barcode序列混样测序的情况下,barcode识别区分的结果展示如下,包括barcode识别效率,区分的文库个数及每个文库中序列个数占比和长度信息等。

上面展示了分析结果的部分内容,更多细节的内容可参考底部的相关链接。
rawdata的质控评估只是整个信息分析的开始,是为了对测序数据有大致的整体认识,以便更好地指导后续分析。然而分析的每个环节都会对最终结果产生影响,因此每一步的处理都要深思熟虑。
小编寄语
2018年8月牛津纳米孔公司与百迈客公司达成长期合作,拥有MinION、GridION X5和PromethION三种型号全套纳米孔测序仪。至今已积累了丰富的项目经验,全长转录组成功案例先后发表在《Plant Biotechnol J》、《J Hazard Mater》、《Biotechnol Biofuels》、《Sci Rep》、《Fish & Shellfish Immunology》等国际知名期刊,已发表文章研究物种分别有杨树、吴松草、风筝果、甘薯、野生甘薯、兔子、跳甲、花羔红点鲑和辣椒,覆盖领域分别为林木、哺乳动物、昆虫、水产和作物等。
如您有任何全长转录组等相关问题,欢迎点击下方按钮,我们将竭尽全力为您答疑、设计方案和提供高分成功案例等。
参考链接:
https@//github.com/nanoporetech/ont_tutorial_basicqc(@换成:)
https@//community.nanoporetech.com/knowledge/bioinformatics(@换成:)







 京公网安备 11011302003368号
京公网安备 11011302003368号