研究背景
棉花是重要的经济作物,提供了高质量的棉纤维用于工业生产和人类使用。不断增长的工业需求给棉花增产提出了更高的要求。单铃重是重要的产量构成因子之一,如何在不降低其他纤维品质的前提下提高产量是育种家孜孜以求的目标。通过MAS辅助育种能够直接对基因型进行选择。基于遗传图谱的QTL定位研究可以定位到相应的功能基因或者与功能基因紧密连锁的分子标记,因此能极大地促进MAS研究。常规的SSR标记很难构建饱和的遗传图谱,而利用SNP标记则可以解决这个问题。利用高通量测序技术能够对全基因组开发SNP标记,使高密度遗传图谱构建成为可能。
本研究利用SLAF-seq技术对包含196个子代的陆地棉RIL群体构建高密度遗传图谱,并结合多年多点单铃重的表型数据进行QTL定位。同时针对QTL定位区域进行候选基因的挖掘。
材料和方法
本文亲本是0-153和sGK9708,0-153纤维品质较好,而sGK9708产量潜力高且适应性广。双亲单铃重性状差异极显著。F6:8重组自交系196个。方法:利用SLAF-seq技术构建高密度遗传图谱并进行QTL定位。QTL定位软件:Windows QTL Cartgrapher 2.5。
结果分析
共获得443.56M reads共计87.89GB数据。数据Q20为82.24%,GC含量为34.47%。亲本共开发SLAF标签5.3万个,深度分别为78.66X和102.13X。子代平均开发5万个SLAF标签,平均深度为14.5X。基于SLAF标签共开发出160876个SNP,其中亲本中多态性有23519个,多态率为14.62%。适合棉花作图的SNP标记(aaxbb型)18318个,去除低质量、低深度和极显著偏分离的标记后后剩余5521个SNP用于遗传图谱构建。构建了棉花A基因组和D基因组连锁群26个,共3259.37cM的遗传图谱,平均图距0.78cM。其中A基因组包含标记3550个,遗传距离1838.37个,D基因组包含标记个1971个,遗传距离1971cM。
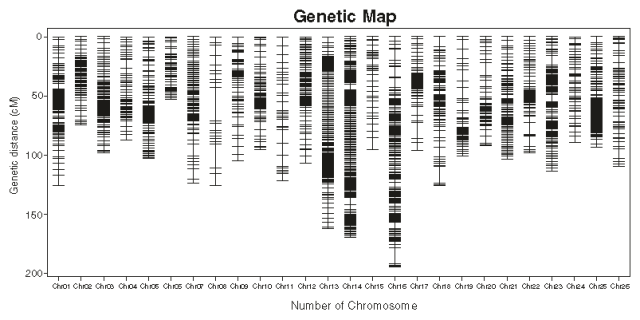
(1)遗传图与棉花物理图谱共线性分析发现图谱覆盖度良好,绝大多数的SNP与物理图谱的共线性良好,相对于A基因组而言,D基因组的共线性更好。
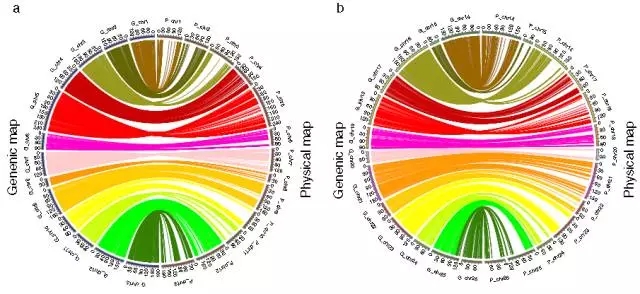
(2)重组热点分析发现26条连锁群中,21条含有重组热点区域,A套中9条LG含有重组热点,D套中12条LG含有重组热点。所有LG中,13号含有重组热点最多,有196个。
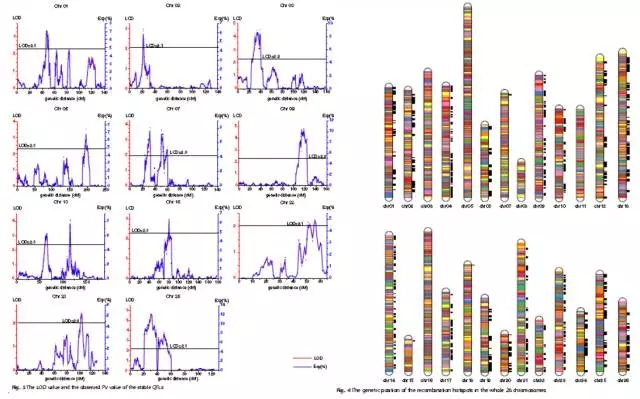
结合多年多点的表型数据进行QTL定位,共检测到11个不同环境下146个单铃重的QTL位点,其中16个能在至少3个环境中重复检测到。这16个QTLs中qBW-chr13-7能在7种环境下检测到,包含26个SNP标记,解释表型变异率在6.13%-14.7%之间。结合参考基因组共鉴定出344个候选基因,其中340个基因含有注释信息,并利用GO,KEGG和KOG数据分别进行基因富集分析。为更进一步精细定位和MAS育种奠定基础。
参考文献
Construction of a high-density genetic map by specific locus amplified fragment sequencing (SLAF-seq) and its application to Quantitative Trait Loci (QTL) analysis for boll weight in upland cotton (Gossypium hirsutum.).
BMC plant biology,2016.








 京公网安备 11011302003368号
京公网安备 11011302003368号