文章发表在Scientific Reports上,利用SLAF-seq技术构建的高密度遗传图谱定位珍珠蚌珍珠品质相关性状QTLs,相信本篇文章能够给珍珠蚌育种工作带来进步,以后珍珠品质和产量势必会得到提高,喜爱珍珠或者珍珠面膜的女士们可以花费得更少,买买买!!!
好了,回到文章本身,先看看文章的材料情况:
亲本:来自鄱阳湖的母本X来自洞庭湖的父本(来自千岛湖的蚌蚌们表示不服)
子代:200个F1子代
再看文章的结果:
基本测序结果分析:
在自然状态下很难保证所有F1群体均是目标亲本的子代,所以作者进行了亲自鉴定的工作,最终剩下157个正牌的F1子代;
共获得了139,113,148个reads,其中Q值大于30以上的高质量数据占88.64%,GC含量为37.45%。父母本reads数分别为9,025,115和8,571,660,F1子代平均为773,990。与之对应的高质量SLAF-tag数为239,704个,其中父母本中开发的SLAF-tag数分别为201,805和206,806个,深度为19.25X和20.70X;子代中平均SLAF-tag为123,038个,平均深度为3.11X;
聚类分析后共获得多态性的SLAF-tag132,542个,其中可成功进行二等位编码的SLAF-tag108,004个,选择适合F1群体作图的多态性标记后,利用测序深度和完整度过滤,最终剩余4,508个多态性SLAF标记用于遗传图谱的构建;
遗传图谱构建:
结合506个SSR标记和4,508个SLAF标记,进行遗传图谱的构建,成功构建了珍珠蚌19条连锁群,共含有标记4,920个,中性图总图距为2,713cM,平均图距为1.81cM。雄性图包括3,233个标记,总图距2,561cM,雌性图包含标记3,123个,总图距2,810cM;

图1 珍珠蚌高密度遗传图谱
遗传图谱比较统计分析:
作者之前利用SSR标记做的遗传图谱和利用SLAF-seq构建的遗传图谱进行了比较。两张遗传图谱均构建了珍珠蚌19条连锁群,但是标记数,图距,分辨率均不同。SSR标记包含了506个标记,总图距1,922.3cM,平均图距3,99cM,大的Gap数较多,而基于SLAF-seq的图谱在以上指标上均显著优于SSR标记图谱。
QTL定位分析:
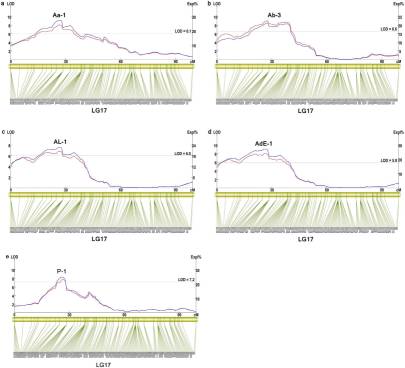
共定位到了26个和珍珠品质相关的QTL,分别位于第1,4,8,14,15和17号连锁群上,PVE范围为8.45%-22.8%。在1号连锁群上定位到3个壳宽QTLs,在第1和第15号连锁群上分分别定位到1个控制壳重的QTL。第8号染色体上定位到体重相关的7个QTL,另外,作者也定位到了大量其他性状的QTL。同时对第17号染色体上的4个和贝壳珍珠层颜色相关的QTL进行简单的统计验证。
文章亮点总结:
1.材料:珍珠蚌需要生长到一定年限才能有珍珠产生,本文材料非常难得,是能获得较好结果的基础;2.有SSR遗传图谱的构建,并与SLAF标记进行了整合,图谱密度高;
3.利用遗传图谱定位到了大量和珍珠质量相关的QTL,相关标记可以进一步开发用于分子标记辅助育种,具有重要的经济价值;
4.部分QTL区域的标记进行简单的统计学验证,使QTL定位结果的可靠性增加。
本期文献解读就到这里,提前透露下,图谱君最近正在运作另外一篇水产物种的图谱构建,QTL并且利用一种高(涨)逼(姿)格(势)的方法进行QTL验证的文章,敬请期待~~~
参考文献:
Bai Z Y, Han X K, Liu X J, et al. Construction of a high-density genetic map and QTL mapping for pearl quality-related traits in Hyriopsis cumingii[J]. Scientific Reports, 2016, 6.







 京公网安备 11011302003368号
京公网安备 11011302003368号