胰腺癌是消化道常见恶性肿瘤之一,晚期极易出现转移,其中胰腺癌肝转移很常见。据柳叶刀杂志记载,胰腺癌确诊后的五年生存率约10%,是预后很差的恶性肿瘤之一。所以临床上获得一种marker用于对胰腺癌患者做出早期诊断尤为重要。
中文题目:Hi-C+ATAC+Chip+RNA-seq多组学揭示胰腺癌转移过程中3D表观图谱
英文题目:High-resolution Hi-C maps highlight Open Access multiscale 3D epigenome reprogramming during pancreatic cancer metastasis.
期刊:Journal of Hematology & Oncology
IF:17.388
发表时间:2021.8
作者:Bo Ren
研究背景
近年来胰腺癌的发病率稳步上升。尽管在大多数人类癌症的治疗方面取得了重大进展,但胰腺癌因为其远端转移导致其仍然是一种致命的恶性肿瘤。目前,远处转移患者的5年生存率仅为3%,远低于局部病变患者。越来越多的表观遗传学知识表明,DNA甲基化和组蛋白修饰与胰腺癌病理生物学和亚型相关,并且它们在胰腺癌进展过程中发生显著变化。然而,作为表观遗传学信息的一个组成部分,3D表
基因组在胰腺癌生物学中的作用,尤其是在其转移中的作用尚不清楚。
本文中,作者通过Hi-C、ChIP-seq、ATAC-seq和RNA-seq研究了来自正常胰腺上皮、原发性和转移性胰腺癌的细胞的高分辨率3D表观基因组图谱,来识别参与胰腺癌转移的关键基因。
结果
通过多组学技术,作者发现转移性胰腺癌细胞的Compartment A/B、TAD和loop环发生显著变化,表明在胰腺癌转移期间,大染色质组蛋白H3赖氨酸9(H3K9)修饰的异染色质的重新编程、可伴随compartment和TAD的重新编程,胰腺癌转移过程中增强子重编程可伴随loop环的重新编程。同时,作者找到了胰腺癌转移关键的候选基因LIPC在转移癌中与特异性增强子成环,并在胰腺癌转移病灶中显著上调。GEO数据集和的IHC分析表明,与原发性胰腺癌和正常胰腺组织相比,LIPC在肝转移中的表达更高。体内体外实验均显示,LIPC可以促进胰腺癌的迁移、侵袭和EMT(上皮细胞-间充质转化)过程。该文章通过多组学分析构建了一个完整的胰腺癌转移过程的3D基因组图谱,扩展了对胰腺癌转移过程中基因调控机制的认知。
材料与方法
材料:正常胰腺细胞(HPNE)、原发胰腺癌细胞(PANC-1)、肝转移(Capan-1)的胰腺癌细胞;
方法:Hi-C、ATAC-seq、Chip-seq、RNA-seq、TCGA+GEO
研究内容
一、Compartment重排与胰腺癌转移的关系
首先作者利用Hi-C数据通过Pearson相关矩阵的主成分分析确定了胰腺癌转移过程中的Compartment A/B及其变化。作者发现转移和正常/原发性胰腺癌细胞之间的Compartment切换更为显著[Capan-1与HPNE中的26.70%和Capan-1与PANC-1中的30.75%,正常胰腺导管和原发癌细胞之间的间隔切换16.17%]。
为了研究这些表观遗传学特征与A区和B区的划分之间的关联,作者测试了组蛋白修饰的相对水平和跨区的染色体可及性。发现A区具有更高的活性组蛋白修饰,包括H3K4me3、H3K27ac和H3K36me3。H3K9me3在HPNE和PANC-1细胞的B区室中显著富集,而Capan-1细胞的B区室具有较高水平的H3K27me3富集。
作者对Compartment利用PC1值进行聚类分析,以确定正常和原发癌细胞的B区室转变为转移癌细胞的A区室(图1a),并且位于这些区室中的基因表现出更高的表达(图1b)。GO富集分析结果显示,这些基因富含Ras/small GTPase 、 oxidation–reduction term,而这些是胰腺癌进展所必需的。这些结果提示,Compartment重排可能促进胰腺癌的转移。
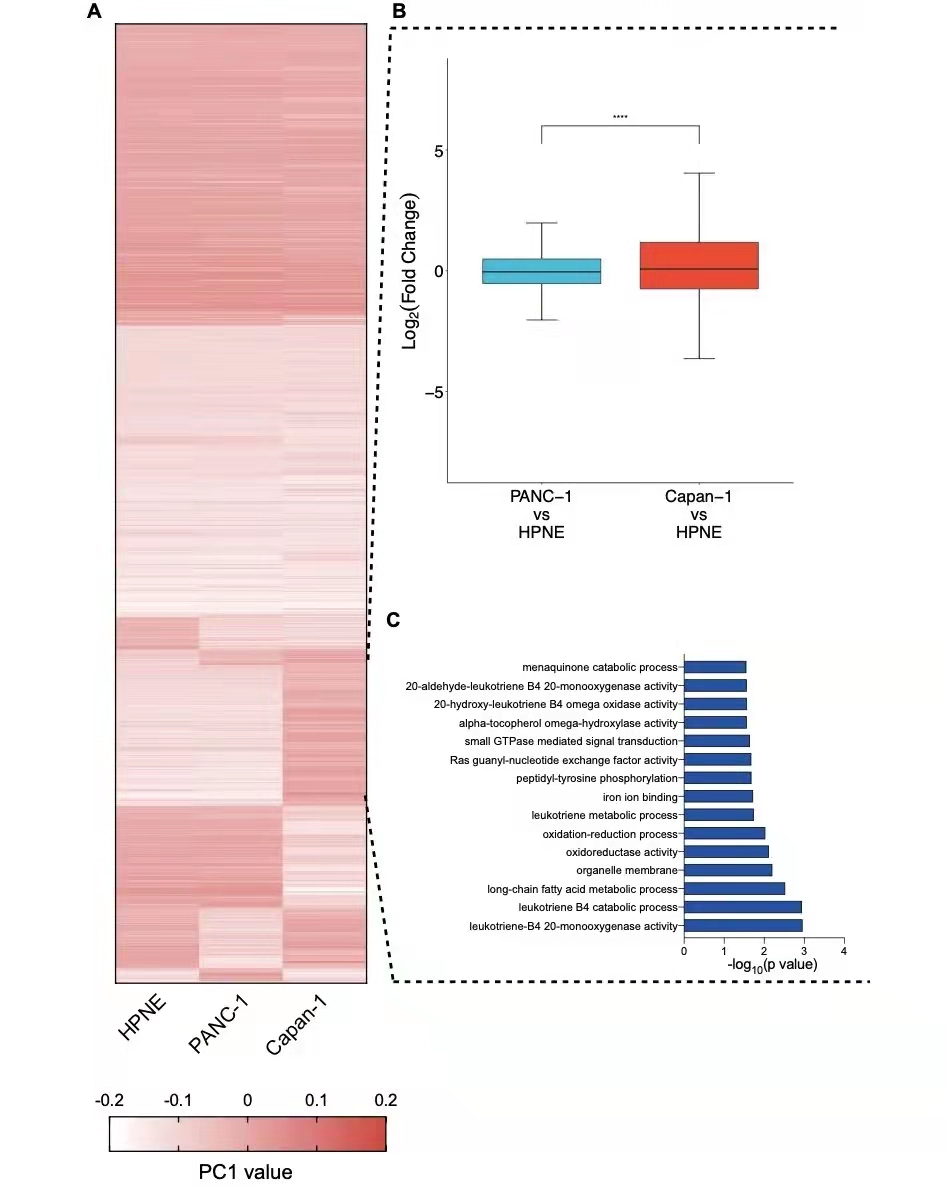 图1
图1
二、胰腺癌转移过程中组蛋白修饰变化与TAD分裂
作者在HPNE细胞中鉴定出6398个平均大小为237kb的TAD,在PANC-1细胞中鉴定出6449个平均大小为247kb的TAD,在Capan-1细胞中鉴定出8580个平均大小为185 kb的TAD。有趣的是,Capan-1细胞中的TAD的大小变得比HPNE中的(p<0.0001,Wilcoxon秩和检验)和PANC-1细胞(p<0.0001,Wilcoxon秩和检验)中的小得多(图2a)。同时发现正常和原发癌细胞的TAD边界数量几乎相同,但在胰腺癌转移期间显著增加,这与转移癌细胞中TAD大小的减小相一致,表明胰腺癌转移过程中新的TAD边界形成。
通过Chip数据分析,与转移癌相比,原发癌具有更高的H3K27me3富集的特异性TAD,表明转移过程中,非活性的TAD转变为活性的TAD。例如BCAT1基因,是重新编程支链氨基酸代谢所必需的,位于新的富含H3K36me3的TAD中(图2d),并且其表达量显著升高升高(图2e)。根据TCGA数据集显示,BCAT1在有淋巴结转移的胰腺癌样本中BCAT1表达上调(图2f)(p=0.02,Student t检验),并且BCAT1表达与总生存率呈负相关(图2g)(p=0.028,对数秩检验)。总之,这些结果表明,在胰腺癌转移过程中,结构域的分裂可能与表观遗传状态的改变相结合。
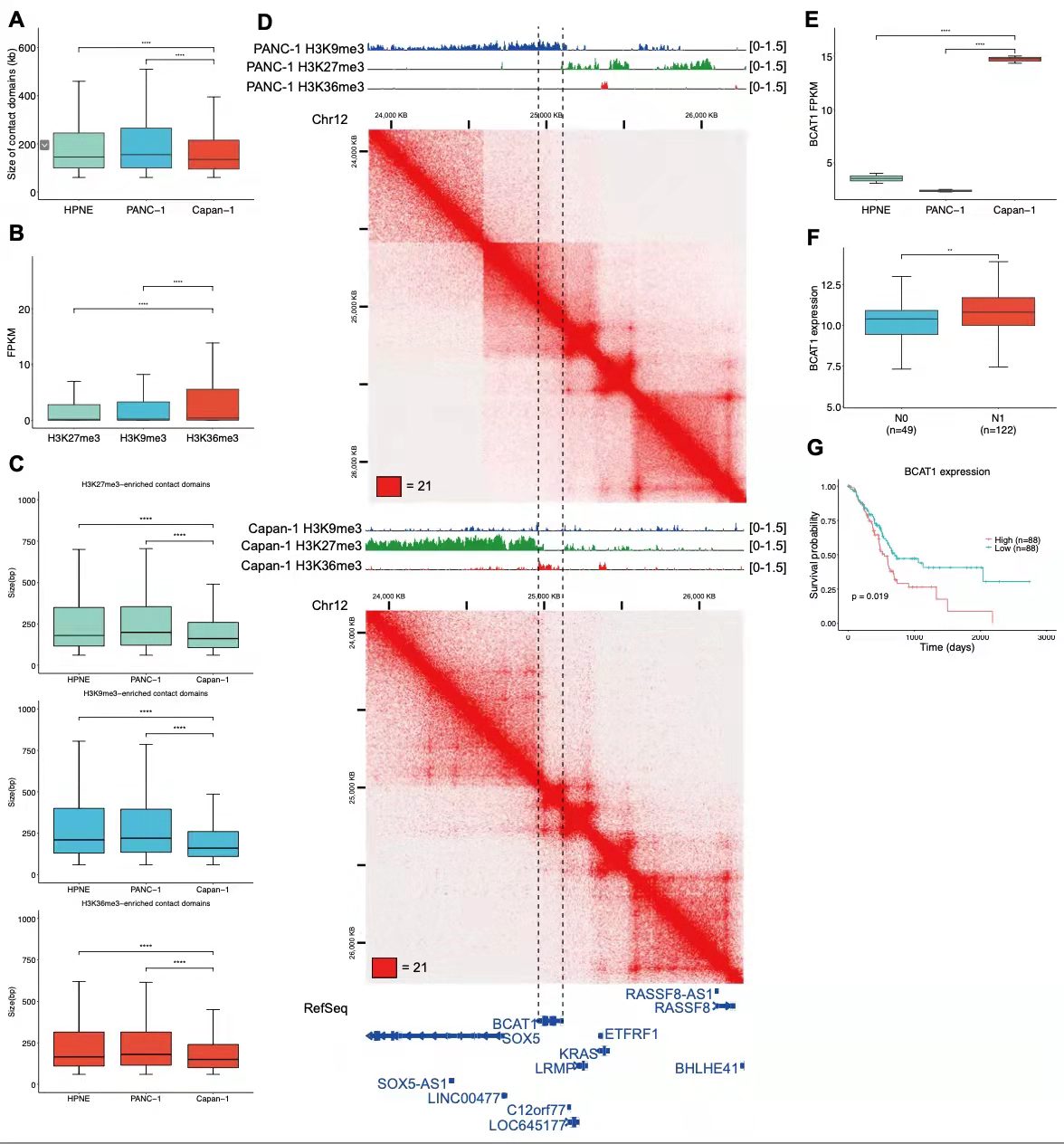
图2
接下来,作者重点关注原发性和转移性癌细胞之间的共同结构域。提取了位于共同结构域中的基因,发现这些基因中的大多数在转移癌样本中上调,并且GO分析显示显著上调的基因(log2FC≥ 1和调整p值≤ 0.05)在血小板衍生生长因子受体信号通路中富集(图3b)。此外,作者利用TCGA胰腺癌患者队列的基因表达数据和长期随访数据来确定这些基因的表达与患者生存率之间的相关性。分析表明,这些基因的高表达显著降低了患者的生存率(p=0.0175,对数秩检验)(图3c)。例如,TGFA基因,位于原发癌富集H3K27me3的结构域中,但位于转移癌的富集H3K36me3的结构域中。而这个基因的高表达显著降低了患者的生存周期(图3f)。也就是说,共同结构域中的染色质状态改变也可促进胰腺癌转移。
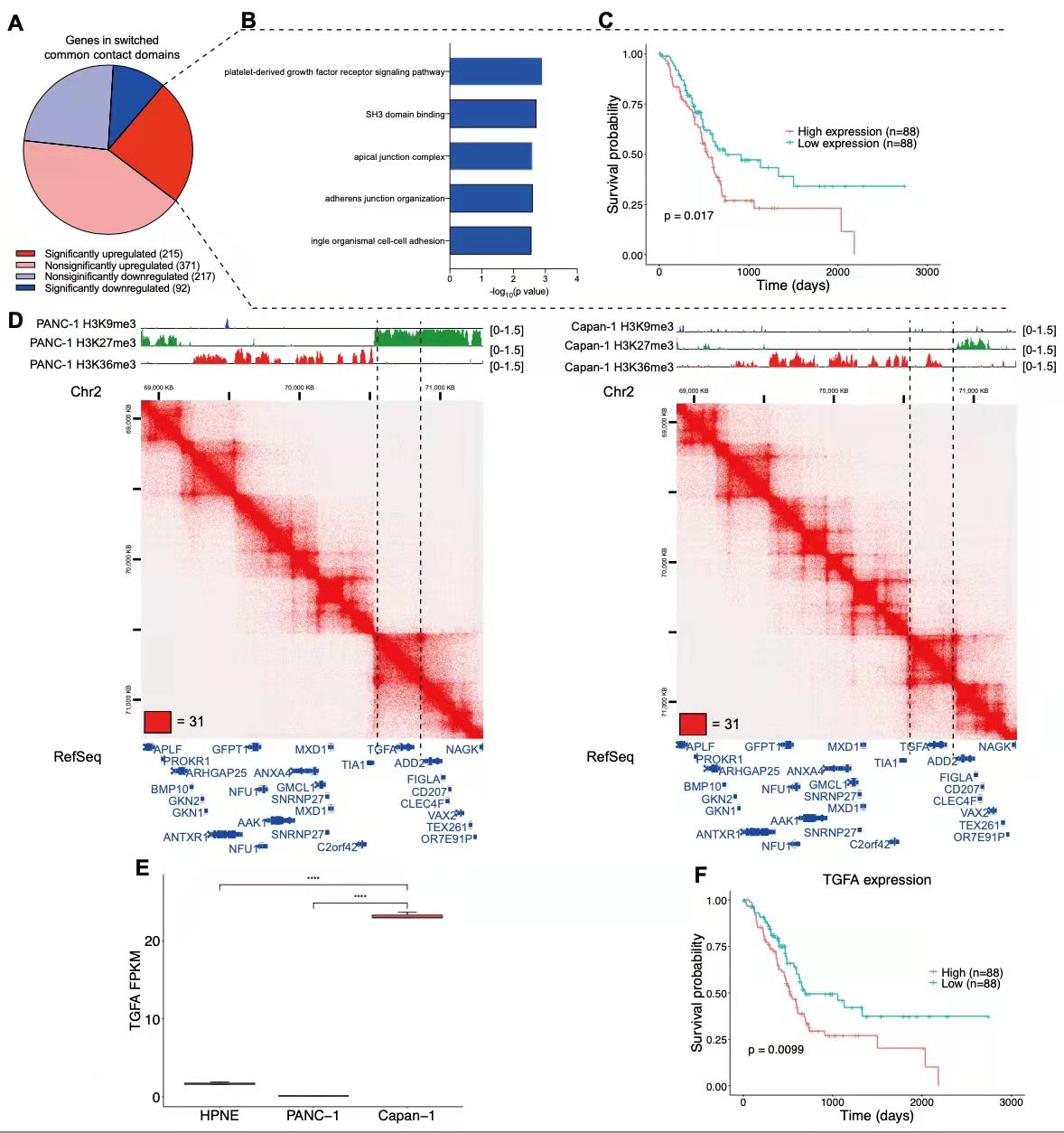 图3
图3
三、与转移特异性增强子相关的基因与胰腺癌转移和不良预后相关
通过loop环的分析,loop数量与TAD总体趋势类似,在转移细胞癌中显著增加。与原发癌细胞相比,大多数启动子与Capan-1特异性增强子成环的基因表达上调,同时,这些基因的表达水平明显高于与PANC-1特异性增强子连接的基因。作者对显著上调基因富集分析发现:这些基因在细胞迁移和血管生成方面显著富集(图4c),表明这些基因与胰腺癌转移相关;而且生存分析发现,这些基因的高表达与不良预后显著相关(图4d)。这些结果表明,胰腺癌转移过程中环的变化可上调转移相关基因。
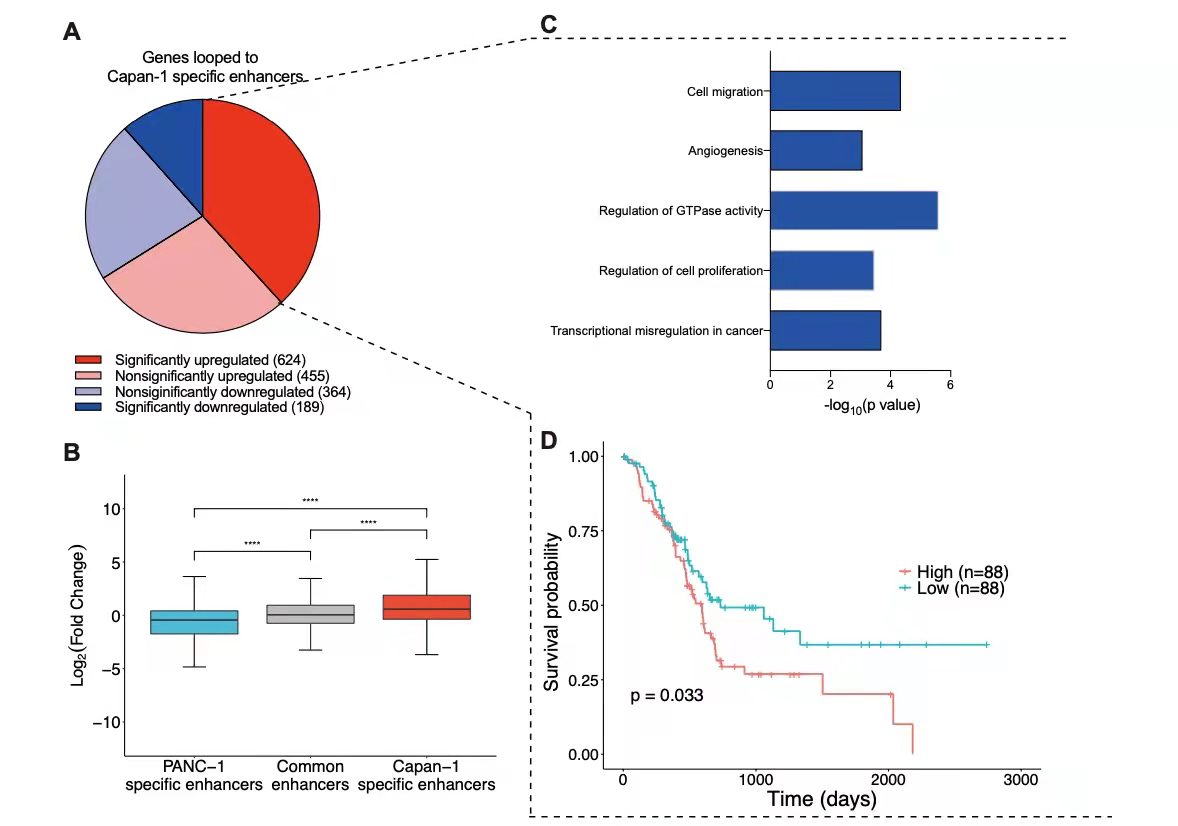
图4
四、Hi-C鉴定LIPC为胰腺癌转移促进基因
利用高分辨率Hi-C数据,作者筛选了了600多个与胰腺癌转移相关的环重编程基因。结合GEO数据,定了3个交叉的关键基因,它们与Capan-1特异性增强子形成环,并在胰腺癌转移组织中上调(图5a)。作者重点关注脂肪酶C(LIPC)基因,其在所有对照组中的FC变化最大(Capan-1与PANC-1中为11.44,GSE71279中为1.54,GSE63124中为7.96,GSE42952中为4.34)。H3K27ac芯片序列分析显示有7个候选增强子位于LIPC基因座旁。这些增强子与Capan-1中的LIPC基因环接,但不与PANC-1中的LIPC基因环接(图5b),导致Capan-1中LIPC的表达升高。qPCR结果显示,Capan-1中所有候选增强子和LIPC启动子的表达水平升高(图5c)。此外,3C qPCR检测到Capan-1中LIPC启动子和增强子之间有更强的相互作用(图5d)。
为了测试LIPC对胰腺癌转移的影响,作者过表达和干扰试验,LIPC的过度表达促进了胰腺癌细胞的迁移和侵袭,但LIPC的敲除抑制了胰腺癌细胞的迁移和侵袭。原位异种移植肿瘤模型显示,与Lv shNC组相比,Lv shLIPC组的肿瘤负荷显著降低。LIPC沉默组的原发肿瘤重量、肝脏重量和有肝转移的胰腺癌细胞也较低(图5f-h)。
IHC实验表明,肝转移性胰腺癌组织的临床标本中的LIPC表达。肝转移中LIPC的表达明显高于原发性胰腺癌。同样,原发性胰腺癌中LIPC的表达高于癌旁正常胰腺组织(图5i)。
以上结果表明,在胰腺癌转移过程中,环重编程导致LIPC上调,并从临床和功能上证实了LIPC在胰腺癌转移中的重要作用。
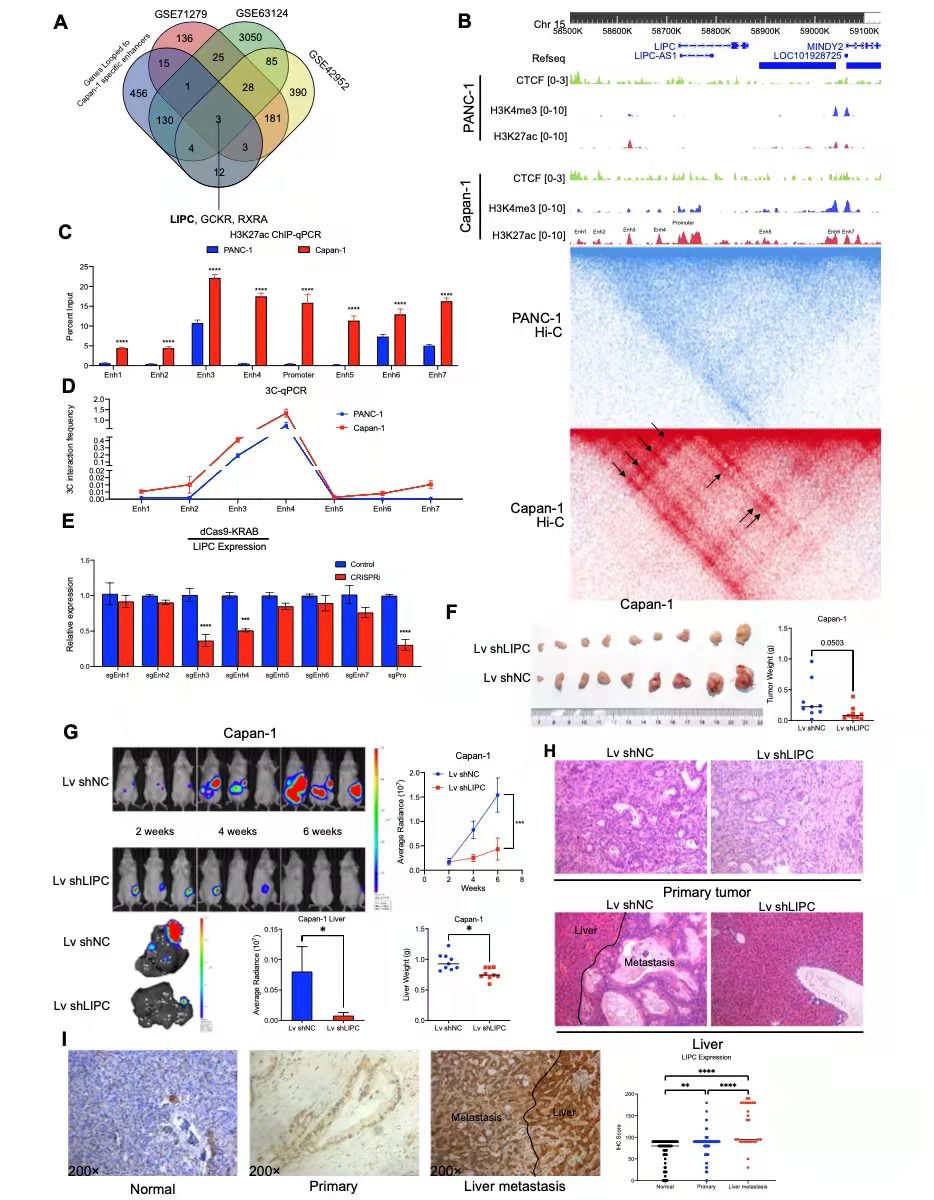 图5
图5
五、参与转移特异性增强子环接至启动子的转录因子
在证明转移特异性增强子的基因环与胰腺癌转移相关后,作者想知道哪些因素有助于这些增强子启动子环的形成。作者整合了ATAC-seq数据,分析了转移癌中参与增强子启动子成环的特异性增强子,确定TFs的motif,如SP1、EGR1和KLF5(图6b)。相反,参与增强子启动子成环的原发性胰腺癌特异性增强子的TFsmotif,如E2F1、MAZ和ZFX(图6b)。位于相应启动子的的这些motif分析结果与位于细胞类型特异性增强子的motif分析结果相似(图6c)。值得注意的是,所有NDR都有CTCF和CTCFL motif,这与CTCF是介导染色质环的重要染色质结构蛋白这一事实一致。
先前的研究表明KLF5对胰腺癌的进展至关重要,而在本次研究中,KLF5的motif在转移特异性增强子和相应的启动子处富集(图6b-c)。RNA-seq分析显示KLF5在转移癌细胞中显著上调(图6d)。此外,通过TCGA数据分析,发现胰腺癌样本中KLF5的表达高于正常样本(图6e),并且KLF5的高表达与胰腺癌患者的不良预后相关(图6f)。利用多组学数据,作者确定了转移癌细胞中KLF5启动子区域的特有的几个增强子(图6g,黑色箭头)。总之,转移特异性增强子环到几个关键TFs的启动子,这些TFs可以介导额外的增强子启动子成环来上调与胰腺癌转移相关的基因。
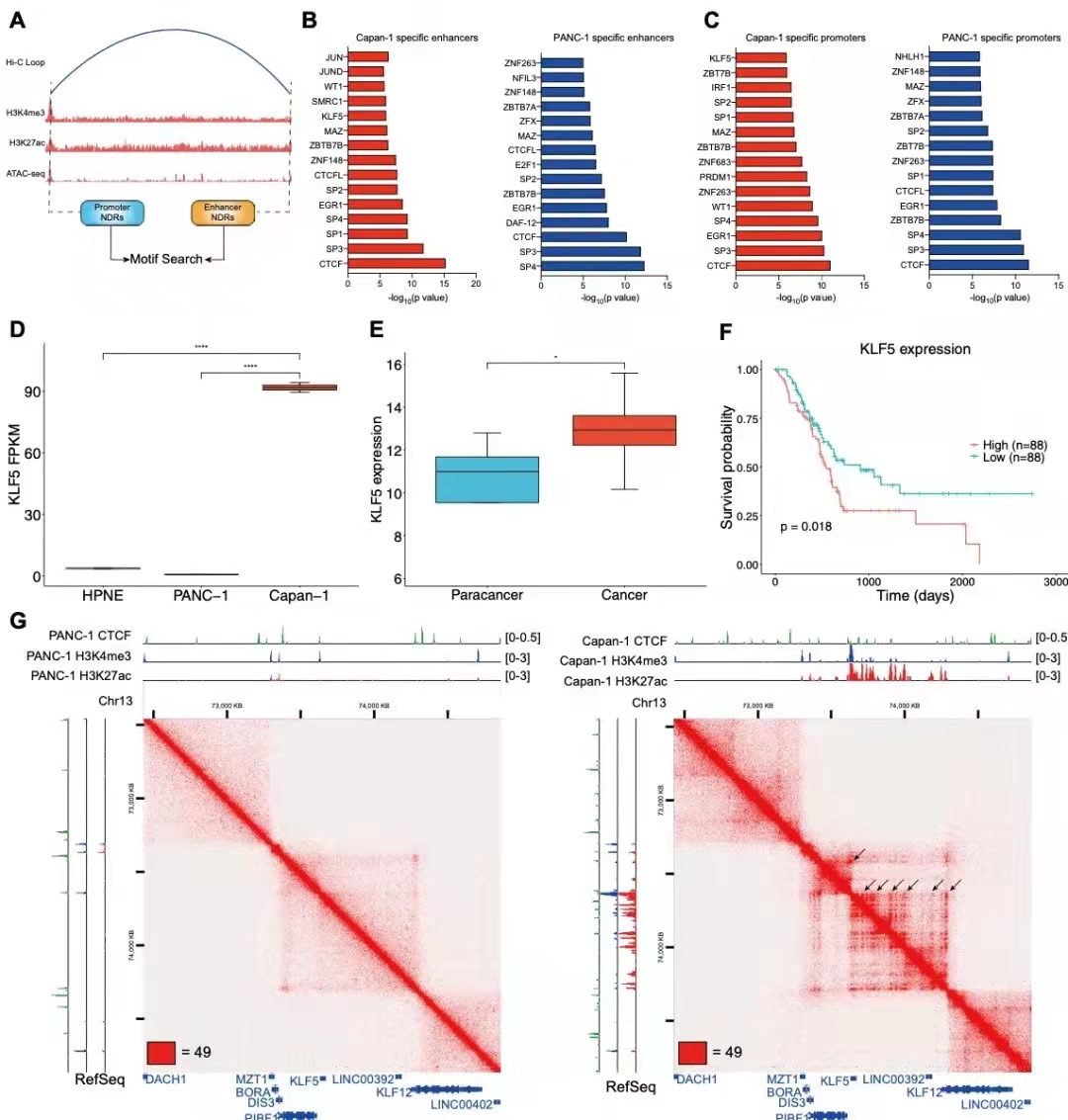
图6
结论
作者通过Hi-C图谱强调了来源于肝转移的胰腺癌细胞在不同染色质级别上表现出显著差异。发现转移性胰腺癌细胞的Compartment重排更为明显。此外,在胰腺癌转移过程中,TAD在基因组和表观基因组上发生改变,其变得更小和更多,这与组蛋白修饰的变化有关。同时,作者发现3D表观基因组重编程确实存在,并鉴定了600多个与胰腺癌转移特异性相关的基因。并通过各种实验,确定了关键候选基因LIPC在转移癌中与特异性增强子成环,并在胰腺癌转移病灶中显著上调。LIPC可以促进胰腺癌的迁移、侵袭和EMT(上皮细胞-间充质转化)过程,这些结果强调了LIPC在胰腺癌转移中的重要作用,并证明3D表观基因组重编程可以上调转移促进基因以促进胰腺癌转移。
推荐阅读
【成功案例】上海交通大学医学院利用多组学技术(RNA-seq、ATAC-seq和Hi-C)联合确定脊索瘤治疗新靶点CA2







 京公网安备 11011302003368号
京公网安备 11011302003368号