自2018年5月高质量亚洲棉基因组文章见刊起,百迈客Hi-C项目成功案例相继见刊势如破竹。短短2年间,成功案例已突破40余篇,更是有超过5篇合作案例刊登于杂志Nature Genetics,其中不乏二倍体、同源多倍体、异源多倍体等复杂且高质量基因组的获得,高阶文章的连续发表印证了百迈客Hi-C技术在科研领域中的优势。
Tips:
Hi-C是染色质区域捕获(Chromosome conformation capture)与高通量测序(High-throughput sequencing)相结合而产生的一种新技术。通过甲醛处理将DNA与蛋白质交联在一起从而固定DNA的构象,并进行酶切、生物素引入、酶连和提取,然后对处理好的核酸进行文库构建和高通量测序,最终通过对测序数据进行分析即可揭示染色体片段间的交互信息。该技术仅利用单个个体就可以将基因组序列定位到染色体,从而进行染色体水平的研究。
至于百迈客Hi-C的研究效果究竟如何?快速速随小编一同概览系列高阶文章吧~
PART ONE

影响因子:27.603
中科院分区:
大类:生物 1区[Top]
小类:遗传学 1区
————————————————————————————————
ONE
2020年4月13日,武汉大学朱玉贤院士团队关于草棉、亚洲棉和陆地棉基因组研究成果发表在国际期刊Nature genetics。该研究利用了北京百迈客生物技术有限公司的Hi-C技术分别将草棉和亚洲棉的95.69%及92.18%序列挂载至13条染色体上,完成了高质量棉花基因组图谱[1]。
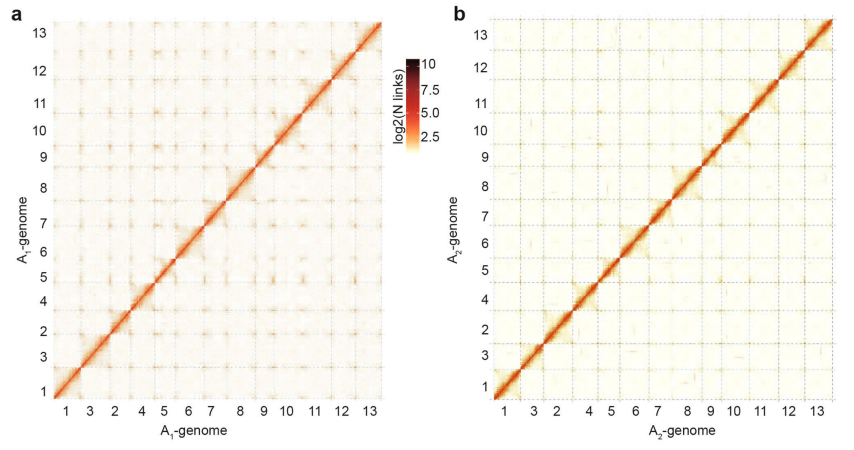
Hi-C辅助草棉、亚洲棉基因组染色体挂载
TWO
2019年9月30日,福建农林大学基因组研究中心明瑞光教授团队联合多个单位共同合作的红苞凤梨研究成果在国际知名期刊Nature Genetics在线发表。该研究获得了513 Mb基因组序列,并通过百迈客Hi-C实验技术助力,最终将456 Mb基因组定位到25条染色体上(挂载率约88.8%),完成了菠萝基因组学领域的再一次突破[2]。
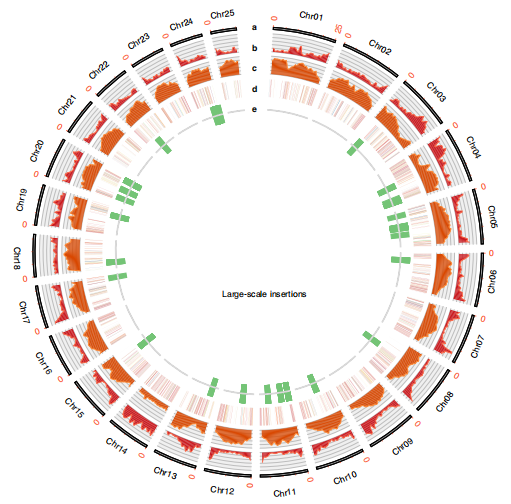
红苞凤梨基因组Circos图
THREE
2018年12月4日,华中农业大学作物遗传改良国家重点实验室张献龙教授课题组首次完成了高质量异源四倍体陆地棉(Gossypium hirsutum)和海岛棉(Gossypium barbadense)基因组组装,相关研究成果刊登于国际知名期刊Nature Genetics。文中利用北京百迈客生物科技有限公司的三代PacBio测序技术、Hi-C染色体挂载技术(海岛棉及陆地棉染色体挂载率分别高达97.68%和98.94%)获得了高质量栽培种异源四倍体棉基因组[3]。
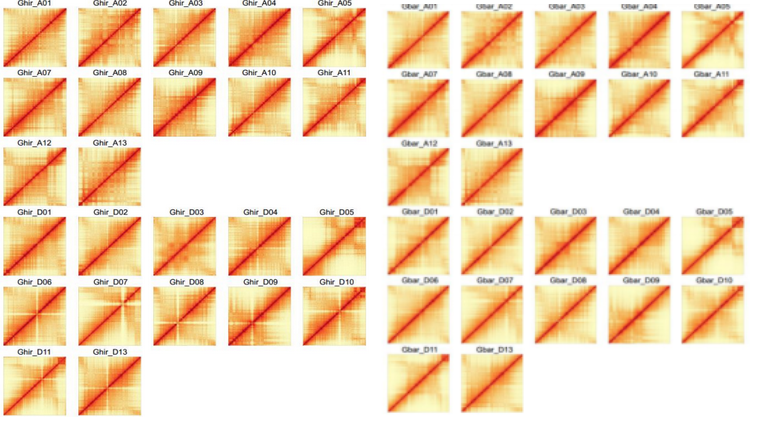
陆地棉和海岛棉基因组Hi-C热图
(Wang M et al.,Nature Genetics.2018)
FOUR
2018年10月8日,福建农林大学基因组研究中心明瑞光课题组首次完成了同源多倍体甘蔗基因组密码破译,该研究成果刊登于Nature Genetics。文中利用二代BAC文库测序,三代PacBio测序及北京百迈客生物科技有限公司的Hi-C建库测序技术,并结合明瑞光课题组独立研发的ALLHiC算法成功地将甘蔗基因组组装到染色体水平,高质量甘蔗基因组的获得印证了百迈客Hi-C研究在科研领域中的优势[4]。
同源多倍体甘蔗基因组Hi-C热图
FIVE
2018年5月8日,中国农业科学院棉花研究所所长李付广研究员、武汉大学朱玉贤院士、中国农业科学院棉花研究所杜雄明研究员、中国农业科学院农业基因组研究所所长黄三文研究员、林涛博士与北京百迈客生物科技有限公司关于亚洲棉的合作成果发表在Nature Genetics上。利用三代PacBio测序+Hi-C技术完成了亚洲棉基因组的重新组装,将基因组Contig N50从72 kb提升到1.1 Mb,并将91.98%的序列挂载至染色体水平上,为亚洲棉后续的群体遗传学等相关研究奠定了基础[5]。
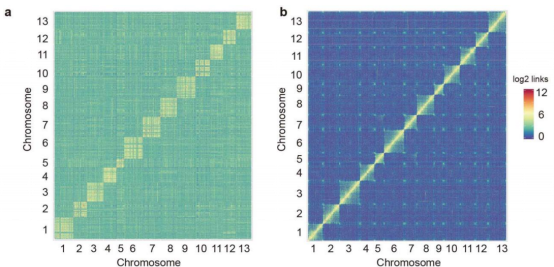
两版亚洲棉基因组Hi-C热图比对(左旧;右新)
PART TWO
影响因子:15.84
中科院分区:
大类:工程技术 1区[Top]
小类:材料科学:综合 1区|纳米科技 1区|化学综合 1区
——————————————————————————————————
殷冬梅教授团队在Wiley知名期刊Advanced Science上发表题为“Comparison of Arachis monticola with diploid and cultivated tetraploid genomes reveals asymmetric subgenome evolution and improvement of peanut”的研究进展,首次揭示了异源四倍体野生种Arachis monticola在花生从野生二倍体到栽培四倍体的重要驯化地位;证实了异源四倍体花生中A和B亚基因组的单系起源以及在驯化过程中的亚基因组功能分化;在多倍体基因组的进化、作物的驯化以及提高花生产量和抗性等方面具有重要价值。
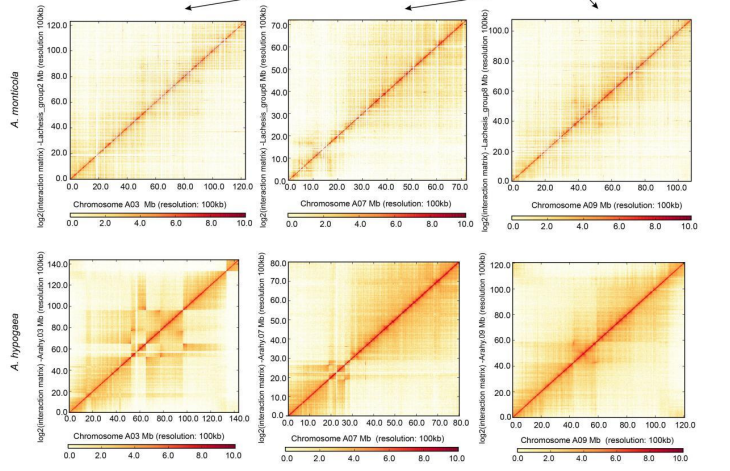
Arachis monticola自身以及与A. hypogaea Hi-C互作热图
PART THREE

影响因子:13.256
中科院分区:
大类:生物 1区[Top]
小类:植物科学 1区
——————————————————————————————
论文题目:Musa balbisiana genome reveals subgenome evolution and functional divergence
研究物种:香蕉(Musa balbisiana)野生种Pisang Klutuk Wulung (B-基因组, 2n=2x=22)
合作单位 :中国热带农业科学院热带生物技术研究所
发表时间:2019年7月
染色体数:11
Hi-C测序深度及挂载率:~138×;87.27%
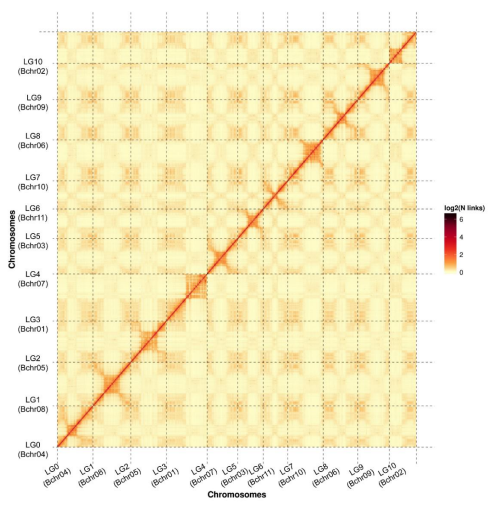
香蕉基因组Hi-C热图
PART FOUR

影响因子:12.121
中科院分区:
大类:综合性期刊 1区[Top]
小类:综合性期刊 1区
———————————————————————————————
ONE
论文题目:Genomic and transcriptomic insights into molecular basis of sexually dimorphic nuptial spines in Leptobrachium leishanense
研究物种:雷山髭蟾(Leptobrachium Leishanense)
合作单位 :华中师范大学
发表时间:2019年12月
染色体数:13
Hi-C测序深度及挂载率:~44×;3.31 Gb(92.9%)
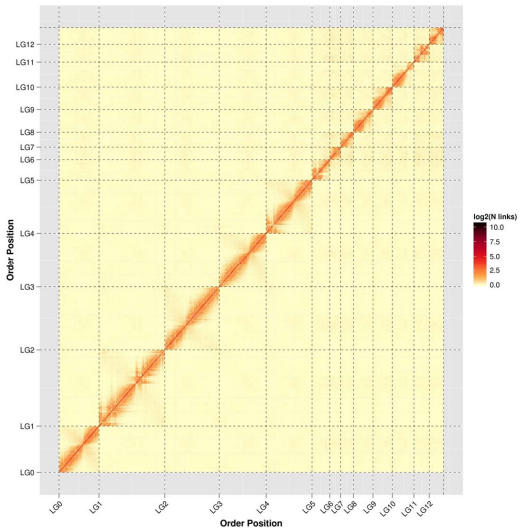
雷山髭蟾基因组Hi-C热图
TWO
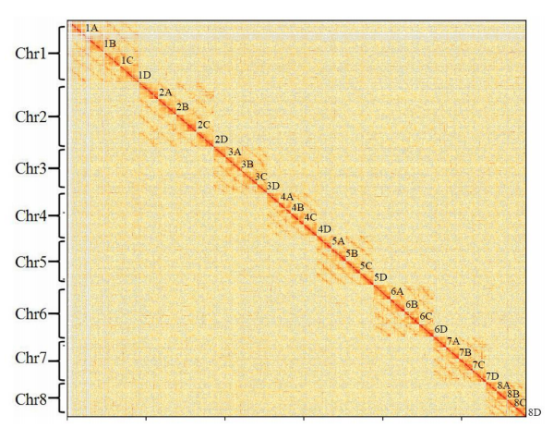
论文题目:Extensive intraspecific gene order and gene structural variations in upland cotton cultivars
研究物种:陆地棉(Gossypium hirsutum L.)TM-1及ZM24
合作单位 :中国农业科学院棉花研究所
发表时间:2019年7月
染色体数:26
Hi-C挂载率:TM-1由先前遗传图的79.2%,提升至97.4%;ZM24挂载率为93.2%
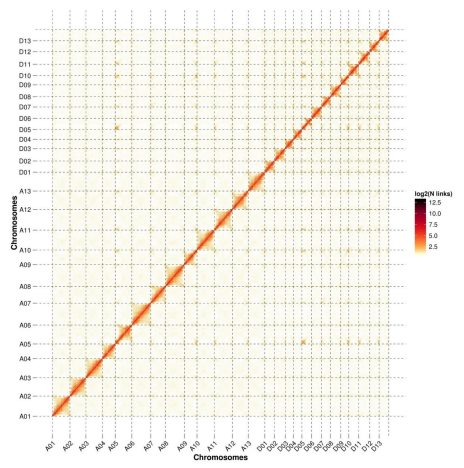
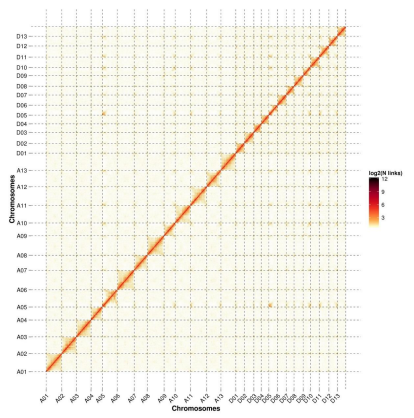
陆地棉TM-1及ZM24基因组Hi-C热图
PART FIVE

影响因子:12.084
中科院分区:
大类:生物 1区[Top]
小类:植物科学 1区|生化与分子生物学 1区
————————————————————————————————
ONE
论文题目:The reference genome of tea plant and resequencing of 81 diverse accessions provide insights into genome evolution and adaptation of tea plants
研究物种:茶树(Camellia sinensis var. Sinensis)
合作单位 :安徽农业大学
发表时间:2020年4月
染色体数:15条
Hi-C挂载率:86.73%
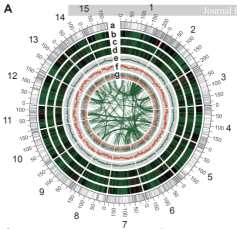
茶树(舒茶早)基因组Circos图
TWO
论文题目:The Coix genome provides insights into Panicoideae evolution and papery hull domestication
研究物种:薏苡“大黑山”(Coix lacryma-jobi L.)
合作单位 :四川农业大学
发表时间:2019年11月
染色体数:10
Hi-C挂载率:98.8%
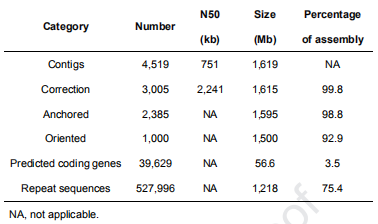
薏苡基因组信息列表
THREE
论文题目:A Chromosome-Scale Genome Assembly of Paper Mulberry (Broussonetia papyrifera) Provides New Insights into Its Forage and Papermaking Usage
研究物种:构树(Broussonetia papyrifera)
合作单位 :中国科学院植物研究所
发表时间:2019年2月
染色体数:13
Hi-C测序深度及挂载率:~38.56×;99.25%
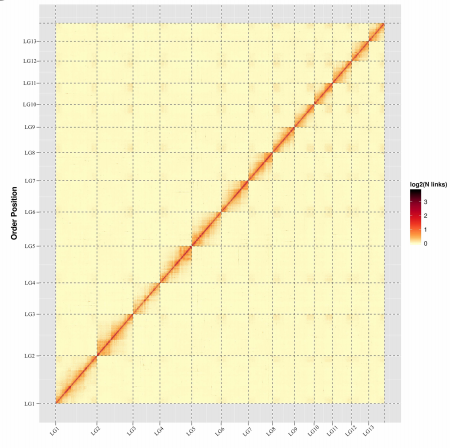
构树基因组Hi-C热图
FOUR
论文题目:Population Genomic Analysis and De novo Assembly Reveal the Origin of Weedy Rice as an Evolutionary Game
物种名称:杂草稻(Oryza sativa f. spontanea)
合作单位:沈阳农业大学水稻研究所
发表时间:2019年1月
染色体数:12
Hi-C测序深度及挂载率:~93×;99.19%
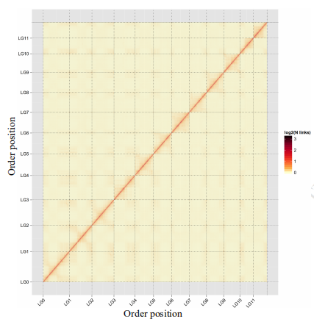
杂草稻基因组Hi-C热图
PART SIX

影响因子:11.093
中科院分区:
大类:生物 1区[Top]
小类:遗传学 1区|生物工程与应用微生物 1区|生化与分子生物学 1区
————————————————————————————————
论文题目:The subgenomes show asymmetric expression of alleles in hybrid lineages of Megalobrama amblycephala ×Culter alburnus
物种名称:翘嘴鲌(Culter alburnus);团头鲂(Megalobrama amblycephala)
合作单位:湖南师范大学、北京农林科学院、中南大学等
发表时间:2019年10月
染色体数:24
Hi-C挂载率:团头鲂93.86%
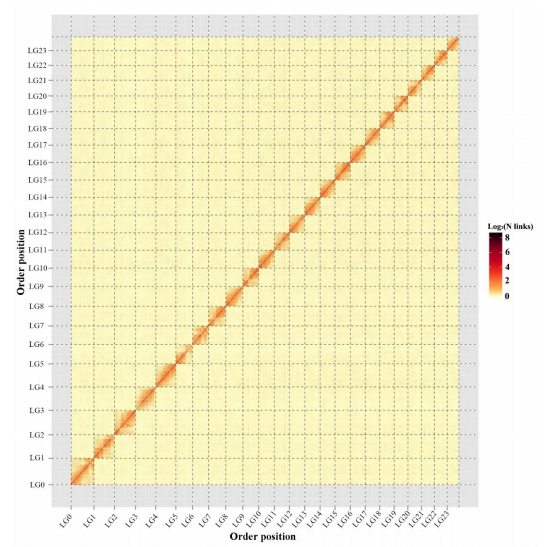
团头鲂基因组Hi-C热图
PART SEVEN

影响因子:8.154
中科院分区:
大类:生物2区[Top]
小类:植物科学 1区|生物工程与应用微生物 1区
————————————————————————————————
ONE
论文题目:The high-quality genome of diploid strawberry (Fragaria nilgerrensis) provides new insights into anthocyanin accumulation
物种名称:黄毛草莓(Fragaria nilgerrensis)
合作单位:沈阳农业大学
发表时间:2020年1月
染色体数:7
Hi-C测序深度及挂载率:~55.88×;97.89%
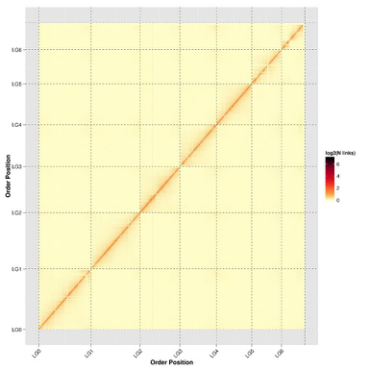
黄毛草莓基因组Hi-C热图
TWO
论文题目:The high-quality genome of diploid strawberry (Fragaria nilgerrensis) provides new insights into anthocyanin accumulation
物种名称:突尼斯石榴(Punica granatum L.)
合作单位:中国农科院郑州果树研究所
发表时间:2019年9月
染色体数:8
Hi-C测序深度及挂载率:~55×;97.76%
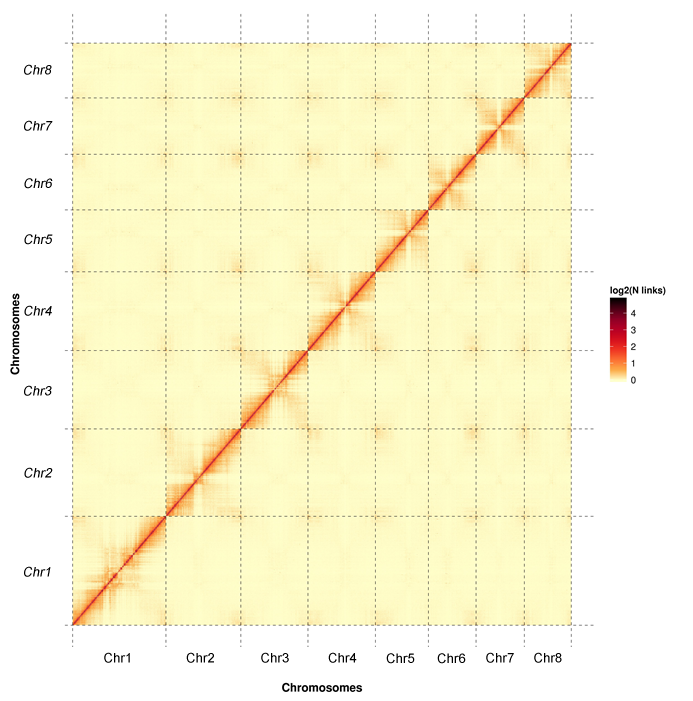
突尼斯石榴Hi-C热图
TREE
论文题目:Genome sequencing of the Australian wild diploid species Gossypium austral highlights disease resistance and delayed gland morphogenesis
物种名称:澳洲棉(Gossypium austral)
合作单位:河南大学
发表时间:2019年9月
染色体数:13
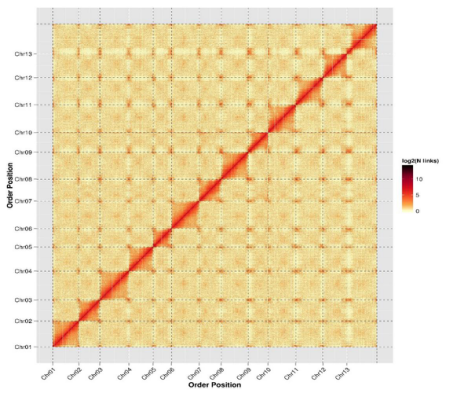
澳洲棉基因组Hi-C热图
PART EIGHT

影响因子:7.051
中科院分区:
大类:生物 1区[Top]
小类:遗传学 1区
————————————————————————————————
论文题目:Tung Tree (Vernicia fordii) Genome Provides A Resource for Understanding
Genome Evolution and Improved Oil Production
物种名称:油桐(Vernicia fordii)
合作单位:中南林业科技大学
发表时间:2020年4月
染色体数:11
Hi-C挂载率:95.15%
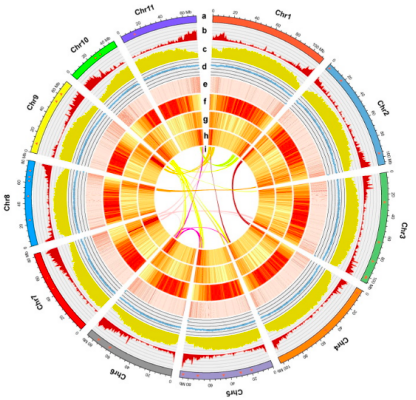
油桐基因组circos图
PART NINE

影响因子:6.286
中科院分区:
大类:生物 1区[Top]
小类:进化生物学 2区|生化与分子生物学 2区|生态学 1区
——————————————————————————————
ONE
论文题目:Chromosome-level genome assembly of the East Asian common octopus (Octopus sinensis) using PacBio sequencing and Hi-C technology
物种名称:中华真蛸(Octopus sinensis)
合作单位:中国水产科学研究院黄海水产研究所
发表时间:2020年6月
染色体数:30
Hi-C测序深度及挂载率:77.93×;96.41%
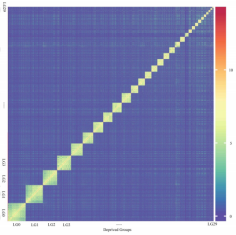
中华真蛸基因组Hi-C热图
TWO
论文题目:Chromosome-level genome assembly of the greenfin horse-faced filefish (Thamnaconus septentrionalis) using Oxford Nanopore PromethION sequencing and Hi-C technology
物种名称:绿鳍马面鲀(Thamnaconus septentrionalis)
合作单位:中国水产科学研究院黄海水产研究所
发表时间:2020年5月
染色体数:20
Hi-C测序深度及挂载率:~84.6×;99.44%
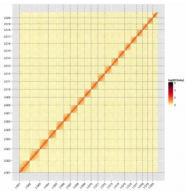
绿鳍马面鲀Hi-C热图
THREE
论文题目:Genomic analyses of a “living fossil”: The endangered dove-tree
物种名称:珙桐(Davidia involucrata)
合作单位:四川大学
发表时间:2020年1月
染色体数:21
Hi-C测序深度及挂载率:112 ×;88.07%
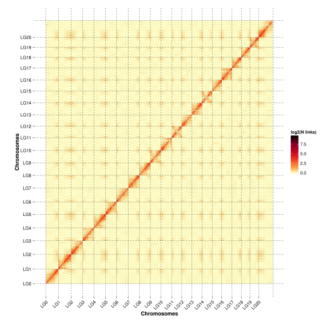
珙桐基因组Hi-C热图
FOUR
论文题目:Chromosome-level genome assembly of the predator Propylaea japonica to understand its tolerance to insecticides and high temperatures
物种名称:龟纹瓢虫(Propylaea japonica)
合作单位:中国农业科学院棉花研究所
发表时间:2020年1月
染色体数:10
Hi-C测序深度及挂载率:162 ×;98.3%
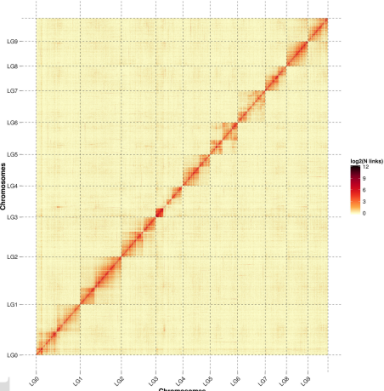
龟纹瓢虫Hi-C热图
FIVE
论文题目:Comparative genome/transcriptome analysis probes Boraginales’ phylogenetic position, WGDs in Boraginales, and key enzyme genes in the alkannin/shikonin core pathway
物种名称:车前叶蓝蓟(Echium plantagineum)
合作单位:南京大学
发表时间:2019年10月
染色体数:8
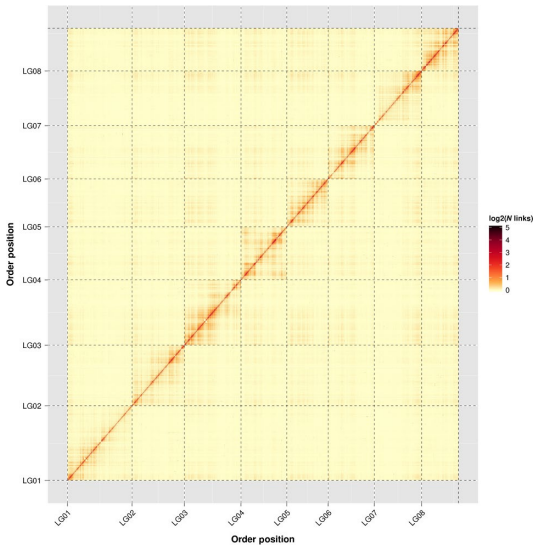
车前叶蓝蓟基因组Hi-C热图
PATR TEN
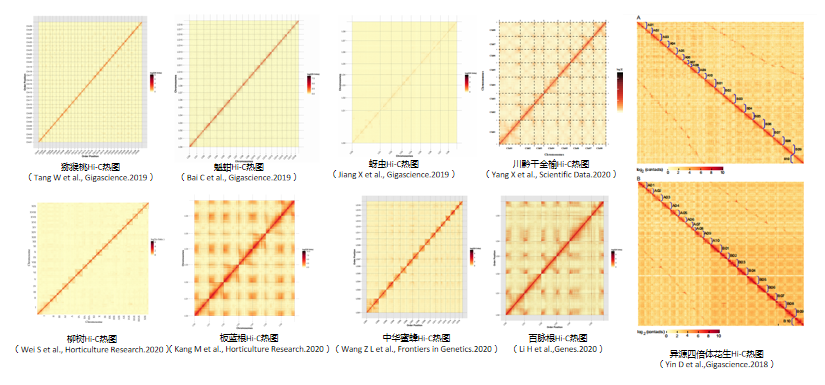
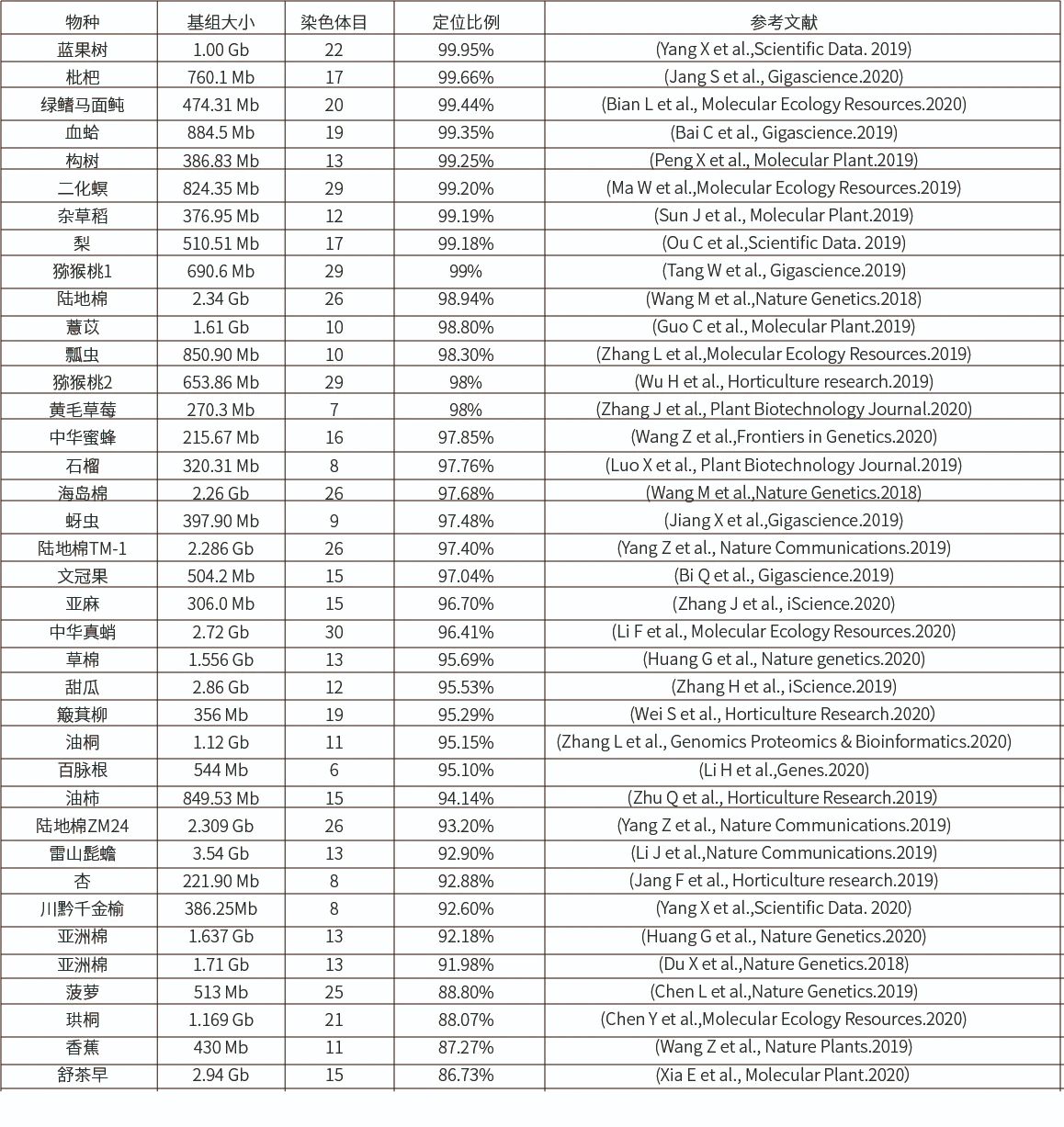
更多百迈客Hi-C挂载成功案例如下所示
文:豆沙包
排版:市场部
参考文献:
[1]Huang G , Wu Z , Percy R G , et al. Genome sequence of Gossypium herbaceum and genome updates of Gossypium arboreum and Gossypium hirsutum provide insights into cotton A-genome evolution[J]. Nature Genetics, 2020, 52(5).
[2]Chen L Y , Vanburen R , Paris M , et al. The bracteatus pineapple genome and domestication of clonally propagated crops[J]. Nature Genetics, 2019, 51(10):1-10.
[3]Wang M, Tu L, et al. Reference genome sequences of two cultivated allotetraploid cottons, Gossypium hirsutum and Gossypium barbadense.[J]. Nature genetics, 2019.
[4]Zhag J, Zhang X, et al. Allele-defined genome of the autopolyploid sugarcane Saccharum spontaneum L[J]. Nature Genetics, 2018, 50.
[5]Du X , Huang G , He S , et al. Resequencing of 243 diploid cotton accessions based on an updated A genome identifies the genetic basis of key agronomic traits[J]. Nature Genetics, 2018.







 京公网安备 11011302003368号
京公网安备 11011302003368号