
2018年12月20日,西安交通大学刘恩岐教授团队与北京百迈客生物科技有限公司合作,通过纯二代测序技术完成了喜马拉雅旱獭基因组组装,获得了喜马拉雅旱獭完整基因组草图,并对其冬眠及环境适应性机制进行深入研究,该研究成果刊登于Cell子刊iScience。喜马拉雅旱獭基因组文章具体研究内容如下:
英文题目:Hypoxic and cold adaptation insights from the Himalayan marmot genome;
中文题目:喜马拉雅旱獭基因组揭示其缺氧和冷适应机制;
发表杂志:iScience;
发表时间:2018.12.20;
合作单位:西安交通大学;
摘要
喜马拉雅旱獭(喜马拉雅土拨鼠)是一种冬眠哺乳动物,栖息在喜马拉雅山脉的高海拔地区。本研究成功组装了喜马拉雅旱獭基因组,组装基因组大小2.47 G。系统发育分析表明,喜马拉雅旱獭与蒙古旱獭大约在198万年前开始分化。旱獭在冬眠期间,与肝脏中脂肪酸代谢相关基因,参与补体和凝血级联相关基因的转录发生变化,以及脑干细胞的多能性途径发生改变。两个受选择基因Slc25a14和ΨAamp,在旱獭低海拔和高海拔种群之间显示出明显的基因分型差异。作为假基因,ΨAamp可能具有生物活性,通过竞争性miRNA结合影响Aamp的稳定性。这些发现揭示了喜马拉雅旱獭适应极端环境的分子和遗传基础。
研究背景
喜马拉雅旱獭(喜马拉雅土拨鼠)是旱獭属的一种大型松鼠,广泛分布在印度,尼泊尔和巴基斯坦喜马拉雅地区以及中国青藏高原的海拔1,900-5,000米处。青藏高原以其气压低,气候寒冷,资源有限的极端环境而闻名。喜马拉雅旱獭具有几个独特的生物学特征,如冬眠,深洞挖掘,厚皮毛和增大的体积,这可能与其对恶劣环境选择压力的演化相关。
许多野生动物,如牦牛,藏羚羊和藏獒,栖息在青藏高原。这些野生动物的高海拔适应机制引起了广大研究者极大的兴趣,尽管不同物种经历了相似的选择压力,但可能涉及到不同的适应途径和相关基因。与其它高原地区的哺乳动物不同,喜马拉雅旱獭冬季在群体中冬眠,冬眠洞穴特别深,有些深度超过10米。哺乳动物的冬眠是代谢抑制和休眠的季节性状态,其特征在于体温,新陈代谢,心率和氧消耗量的降低。喜马拉雅旱獭在冬季面临严重的缺氧和寒冷压力。
本研究通过高深度测序成功组装了雄性喜马拉雅旱獭基因组,对来自高海拔和低海拔地区的20只喜马拉雅旱獭和其它4只旱獭进行重测序,并对冬眠/觉醒状态的喜马拉雅旱獭进行RNA测序。本研究提供了有关高海拔适应和冬眠遗传机制的线索,同时基于喜马拉雅旱獭的生物学研究为研究旱獭进化,高地疾病,冷适应性提供了宝贵的资源。
材料方法
● 材料
一只来自中国青海省西宁市的2岁雄性喜马拉雅旱獭;
● 基因组denovo策略
二代小片段文库(mean insert sizes: 180 bp和500 bp)和mate-end文库(mean insert sizes: 3 Kb, 4 Kb, 5 Kb, 8 Kb, 10 Kb, 15 Kb和17 Kb);Illumina HiSeq 4000;测序深度:~206x;
● 研究方法
基因组组装注释:
1. 基因组调研图:Genome size = 21-mer number / 21-mer depth,JELLYFISH,预估基因组~2.33 Gb;
2.基因组组装:ALLPATHS-LG;SSPACE;GapCloser(V1.12 for SOAP de novo) ,组装基因组~2.47 Gb,contigN50=80.46 Kb,scaffoldN50=1.50 Mb;
3.基因组完整性评估:a,转录组基因完整性预测(旱獭肾,胰腺,肾上腺,肝脏和大脑组织混合),Trinity,BLAT,97.90% unigenes支持组装好的参考基因组;b,CEGs评估:CEGMAv.2.3,BLAT,完整性99.60%;c,BUSCO评估:BUSCOv1.22 (mammalia_odb9),完整性88.34%;
4.重复序列注释:采用同源预测和从头预测的策略对喜马拉雅旱獭基因组进行重复序列预测,软件包括Repeat Scout,LTR-FINDER,MITE和PILER,鉴定了~1.149 Gb重复序列,占旱獭基因组的46.5%,LINE和LTR分别占喜马拉雅旱獭基因组的20.05%和9.22%;
5.非编码RNA(ncRNA)预测:tRNA-scan-SE;miRBase(Release 21);miRDeep2;
6.假基因预测:tBLASTN,GeneWise,鉴定出1,479个假基因;
7.蛋白质编码基因预测和功能注释:利用从头预测(Augustus),同源预测(人,鼠,兔,GeneWise和GeMoMa)及转录组预测(BLAT,PASA,TopHat,Cufflinks,Transdecoder),EVidence Modeler(EVM),对喜马拉雅旱獭蛋白编码基因进行预测,共计预测了21,468个蛋白质编码基因,占整个蛋白质编码基因集的99.38%;
8.基因组注释质量评估:通过比较喜马拉雅旱獭基因组的蛋白质编码基因结构特征与3个注释好的基因组(人:GRCh38.p7;鼠:GRCm38.p4;兔:OryCun2.0),发现喜马拉雅旱獭和其它物种非常相似,表明喜马拉雅旱獭蛋白质编码基因集的具有很好的质量。
比较基因组学分析:
1. 直系同源基因和旁系同源基因鉴定:treeFam,all-to-allBLASTP,MUSCLE,Mafft;
2.系统发育树构建与分化时间估算:利用15个物种(小鼠,大鼠,仓鼠,喜马拉雅旱獭,松鼠,鼠兔,兔子,人类,黑猩猩,山羊,绵羊,牛,猪,驴和马)的4,573个单拷贝直系同源基因构建系统发育树,MUSCLE,phyML,分化时间估算PAML MCMCtree;
3.比较基因组学分析:Treefam,Cafe。
群体进化分析:
1.群体选择:旱獭4个种群(长尾旱獭,蒙古旱獭,灰色旱獭和黄腹旱獭)进行全基因组重测序,揭示旱獭群体进化;为了进一步揭示喜马拉雅旱獭高原适应性机制,本研究中选择了20只个体,包括10只低海拔个体(8只雄性和2只雌性,中国青海~1900m处)和10只高海拔个体(5只雄性和5只雌性,中国青海~4500 m处),进行全基因组重测序,测序深度:~10x;
2.SNP calling:BWA,samtools,Picard,GATK3.0,SnpEff;2.旱獭系统发育关系分析:松鼠作为外群,构建喜马拉雅旱獭,灰色旱獭,黄腹旱獭,长尾旱獭,蒙古旱獭和高山旱獭系统发育树,MUSCLE(v3.349),phyML,MCMCtree;
3.种群历史动态分析:5只旱獭和3种喜马拉雅动物(金色的金丝猴,牦牛和大熊猫),PSMC模型,Mummer3.0;
4.群体结构和遗传关系分析:PCA(EIGENSOFT6.0),Structure(Admixturev1.22);
5.群体高原适应性选择分析:Fst,π;
6.群体受自然选择区域分析:Ka/Ks,t-test;
7.Slc25a14蛋白的同源建模分析:Bio3D和PyMOL1.8;
8.ΨAamp进化与插入时间分析:MEGA7.0,EMBOSSpackage (v6.6.0.0)。
RNA分析:
1.绑定ΨAamp的miRNAs分析:BLASTN;
2.RT-PCR分析;
3.差异表达基因分析:TopHat,Cufflinks。
研究结果
1)喜马拉雅旱獭基因组组装与注释
采用全基因组鸟枪法(whole genome shotgun strategy)及二代测序(Illumina HiSeq 2500),测序了1只来自中国青海西宁的雄性喜马拉雅旱獭,通过对构建17个DNA文库(paired-end 和mate-pair libraries)测序,共计利用了508.41 Gb高质量测序数据进行基因组de novo,基因组覆盖度206x,组装基因组大小~2.47 Gb,contig N50=80 kb,scaffold N50=1.5 Mb,进一步验证了组装基因组的完整性和z确性,喜马拉雅旱獭重复序列含量约占基因组的46.52%(与人基因组相似,基因组重复序列占46.1%),喜马拉雅旱獭与地松鼠有相似的GC含量和CpG频率,在喜马拉雅旱獭基因组中共计鉴定出4,905个非编码RNAs和1,479个假基因,基于同源性预测,从头预测和转录组预测结合的方法,预测了21,609个蛋白质编码基因,其中99.4%被功能注释。基因模型特征与模式动物相似,表明喜马拉雅旱獭基因组的高度z确性。
2)旱獭基因组进化分析
为了确立喜马拉雅旱獭系统发育地位,研究人员比较了喜马拉雅旱獭与其它14种哺乳动物的基因编码序列,这些哺乳动物跨越灵长目,啮齿目,兔形目,奇蹄目和偶蹄目。系统发育分析显示喜马拉雅旱獭(Himalayan marmot)和地松鼠(ground squirrel)在同一个分支中(图1A),这一结果与土拨鼠从地松鼠进化而来的数据一致。喜马拉雅旱獭和地松鼠之间的分歧时间估计约为980万年前(图1 A)。通过比较喜马拉雅旱獭与人,小鼠和兔的蛋白质序列,共鉴定了四种哺乳动物共有的10,358个同源基因家族,及235个喜马拉雅旱獭特有的基因家族,在这些基因家族中,与调控摄食行为和脂肪酸代谢过程相关的基因家族显著增多。每个基因家族的基因变异数作为与物种适应性分化密切相关的的机制,通过与地松鼠比较,推断出在喜马拉雅旱獭中大量扩张和收缩的基因家族分别有221和118个(图1B)。扩张的基因家族主要富集摄食行为相关的功能基因类别(GO:2000253,摄食行为的正调控; GO:0002021,嗅觉感知等),缺氧适应(GO:0003300),心肌肥大; GO:0007596,凝血等)和能量代谢(GO:2000507,能量稳态正调控等)。基于快速进化的基因经过了正向选择这一假设,本研究中共计鉴定了78个受正向选择的基因(PSGs),这些PSGs被认为是参与全身动脉血压调节功能类别(GO:0043281),G2 DNA损伤checkpoint(GO:0031572),甘油三酯代谢过程(GO:0006641)和心肌收缩(GO:0060048)等。喜马拉雅土拨鼠中特定扩张的基因家族和PSGs推断出其在基因组水平上对恶劣环境的适应性演化。
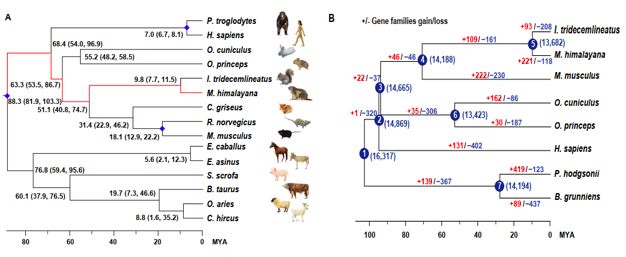
图1、喜马拉雅旱獭基因组进化研究
3)旱獭种群分化与种群历史动态分析
为了在全基因组角度上阐述旱獭进化的情景,研究人员收集了4种旱獭,包括蒙古旱獭(M. sibirica),灰色旱獭(M. baibacina),长尾旱獭(M. caudate)和黄腹旱獭(M. flaviventris),并对每个个体进行全基因组重测序,覆盖度~10x(图2 A)。系统发育分析表明,喜马拉雅旱獭和蒙古旱獭是姊妹种,其分化时间~1.98 MYA(图2B),二者的杂合度相近(图2 C),然而,喜马拉雅旱獭和蒙古旱獭的有效群体大小(Ne)的历史动态趋势显示出完全不同的模式,表明它们对其特定栖息地的适应是独立发生的,并且这些适应性涉及多方面的生物学问题,包括季节性,饮食习惯和社会行为。黄腹旱獭的杂合度高于其它旱獭(图2 C),基于PSMC模型分析发现,其广泛的分布和种群大小正好相符。出乎意料的是,基于全基因组序列和Y染色体基因序列的分析发现阿尔卑斯旱獭 Alpine marmot (M. Marmota)(~5.45 MYA)的分化早于黄腹旱獭,进一步挑战了旱獭可能起源于北美的争论。
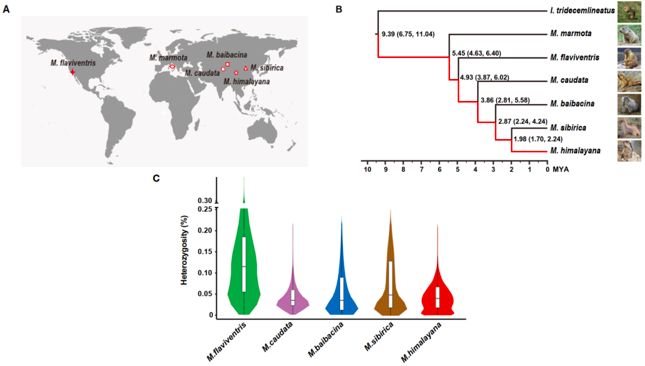
图2、旱獭的地理位置,系统发育关系和杂合度分析
后续利用PSMC模型来检验4个“喜马拉雅”物种的祖先群体的有效群体大小,包括喜马拉雅旱獭,大熊猫,牦牛和仰鼻猴。在两个*大的冰川期间,喜马拉雅旱獭的有效群体大小(Ne)急剧下降:Xixiabangm冰期(XG,1.17~0.8 MYA)和Naynayxungla冰期(NG,0.78~0.50MYA)。在此期间,其它3个喜马拉雅物种群体大小也呈现下降趋势。随后,与其它哺乳动物相比,喜马拉雅旱獭经历了更长的瓶颈期,这可能是由于喜马拉雅旱獭的体型相对较小。然而,喜马拉雅旱獭的有效群体大小(Ne)并未受到最后冰川最大值(LGM,〜2万年前)的严重影响,这表明喜马拉雅旱獭已经适应了恶劣的环境。
4)喜马拉雅旱獭冬眠机制的转录组分析
与其它高原哺乳动物不同,喜马拉雅旱獭在冬季冬眠。为了阐述旱獭冬眠的分子机制,研究人员分析了来自喜马拉雅旱獭肝脏和大脑的RNA-seq数据,以表征在冬眠/觉醒周期中基因表达的变化。差异表达的基因(DEGs)显着富集在肝脏中脂肪酸代谢,萜类骨架生物合成和初级胆汁酸生物合成的途径(图3 A)。并清楚地观察到在冬眠状态下,参与脂肪酸降解的基因的上调及参与脂肪酸合成的基因的下调,暗示了一个√确的脂质代谢调节机制(图3 B)。此外,还发现在觉醒状态下,包含与药物代谢相关的基因-细胞色素P450,氨基酸的生物合成和碳水化合物分解代谢相关基因的总体下调。
在脑部,差异表达基因(DEGs)主要富集于补体和凝血级联途径以及调节干细胞多能性的信号传导途径等(图3 C)。之前有研究指出,由于冬眠者的大脑暴露在接近冰点的温度并且血流量减少,因此淤滞引起血栓的风险增加。研究人员观察到参与补体和凝血级联,细胞色素P450的异生素代谢和昼夜节律的基因的显着下调。此外,最近的一项研究表明,与冬眠者iPSCs分化的神经元保留了内在的抗寒特征,调节干细胞多能性的信号通路中的两个主要转录因子Sox2和Myc在冬眠期间显着上调(图3 D),其维持干细胞的自我更新能力并保护脑部免受冷诱导的损伤。同时,在觉醒期间参与调节干细胞多能性的基因(Lifr,Bmpr2和Acvr2b等)的激活(图3 D)可以促进干细胞分化以修复受损细胞。干细胞多能性的时序性调控可能是喜马拉雅旱獭在极端环境胁迫下生存的一种显着策略。
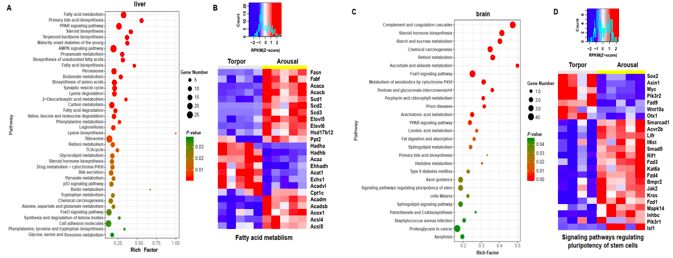
图3、冬眠期差异表达基因分析
根据前期的研究,喜马拉雅旱獭与地松鼠和黑熊有共同的途径,包括脂质和葡萄糖代谢,解毒,补体和凝血级联,以及昼夜节律。然而,研究人员发现在这两个冬眠者(地松鼠和黑熊)和喜马拉雅旱獭之间存在一小部分预测的共有差异表达基因(DEGs)和反常表达模式基因。这些结果表明,喜马拉雅旱獭可能利用多样化的坏境适应性策略在高度季节性或不可预测的环境中生存。
5)高原适应性的遗传进化研究
为了更好地了解喜马拉雅旱獭的高原适应性机制,研究人员从极高海拔(海拔> 4,500 m,n = 10)和相对低海拔(<1,900 m,n = 10)收集了20只喜马拉雅旱獭。研究人员首先测量了血液参数,发现与低海拔喜马拉雅旱獭相比,高海拔喜马拉雅旱獭的一些血液相关性状,尤其是红细胞计数,血红蛋白浓度和平均红细胞体积显着增加。接下来,对每个喜马拉雅旱獭进行了全基因组测序,并在喜马拉雅旱獭基因组中搜索了具有高种群分化(Fst)和多样性比率(qp,low/qp, high)的区域,最终鉴定了分布在喜马拉雅土拨鼠基因组不同scaffolds 中的24.84 Mb受选择的区域。这些区域含有383个功能基因,与非选择性基因相比,这些基因具有更高的Ka / Ks值(P <0.01),主要功能类别包括“缺氧反应”,“DNA修复”,“血管生成”,“心脏功能”,“脂肪酸代谢”,“细胞周期”,“发热”和“钙信号通路”。参与缺氧诱导因子-1(HIF-1),血管内皮生长因子(VEGF)或其它缺氧相关信号通路的10个功能基因,包括Slc25a14,Nox-1,Hmox1,Vegfr2,Atg16l2,Bex1,Ptgr2,Gprasp1,Fam46d 和Chd3,在高海拔和低海拔群体之间显示出明显的基因分型差异(图4 A)。在Slc25a14中发现了非同义替换(由Ser28取代Phe28,F28S),其显示出*强的分化信号(FST = 0.73)(图4 A和B)。基于同源建模的Slc25a14的三维结构显示,F28S替换发生在N末端的loop区域,并且由于氨基的电荷和极性的变化而增加其与相邻的α-螺旋的距离(图4 C)。为了进一步评估这一变体的功能影响,研究人员将突变体Slc25a14与其在不同哺乳动物中的直系同源基因进行比对,并发现Phe28在比对的其它动物中高度保守。该突变体功能影响的预测证明F28S取代是有害的,所有这些结果都表明F28S突变可能是高海拔群体Slc25a14选择性清除的结果。为了推测旱獭高海拔适应和冬眠的遗传适应性机制,研究人员在选择性清除区域鉴定到了116个差异表达基因,在这些基因中,25个差异表达基因与HIF-1途径相关,主要属于DNA修复,血管生成,脂肪酸代谢,细胞凋亡/细胞周期和发热的类别相关。在冬眠/觉醒周期中,大量选择性清除基因具有差异表达,表明这两种生物学过程之间存在共同的遗传影响(图4 D)。在这些基因中,Bex1, Apln, kcne1l, Med12, Dad1和Fgf16在冬眠阶段肝脏或脑中显着上调(图4 D),进一步证明这些基因参与冬眠和高原适应性,先前的研究报道了这些基因参与许多生理过程,包括神经元分化,肝再生,血管生成和能量代谢,而这需要进一步的实验验证。
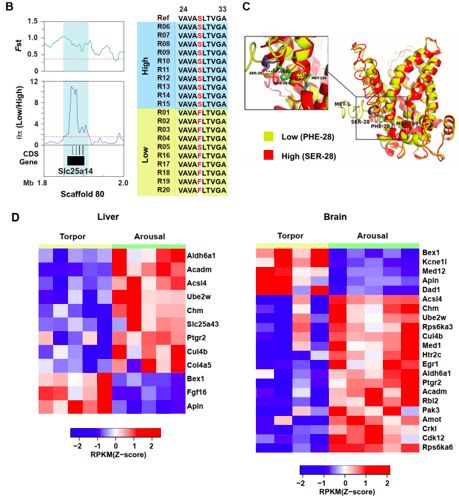
图4、高海拔喜马拉雅旱獭的群体选择和冬眠期间差异表达基因的重叠分析
6)ΨAamp在旱獭高海拔适应中的演化和潜在作用
假基因逐渐被认定为是适应性表型多样化的重要调节因子,与选择性清除信号相关,研究人员鉴定了27个参与极高海拔适应的候选假基因,占全基因组范围内假基因的1.83%。这一比率几乎与具有1.78%的功能基因的比率相当,表明它们在极端环境的适应性中的作用不可忽略。与蛋白质编码基因中发现的群体基因型差异一致,研究人员在高海拔群体中观察到受选择的假基因中几乎固定的突变,例如ΨAamp,ΨAdl1和ΨRnf114。在这些假基因中,血管相关的迁移细胞蛋白假基因(ΨAamp)具有完整的基因结构并显示出*高的Fst值(Fst = 0.67),但ΨAamp未被检测为受选择性基因(图4 A和5 A)。ΨAamp的结构显示它是一个整合的加工假基因,其mRNA通过逆转录被插入基因组中(图5 B)。这个假基因含有过早终止密码子(第208个核苷酸,C> T),缺失/插入和移码突变,使其不能翻译成功能性的蛋白,Aamp和ΨAamp的共线性分析表明在不同哺乳动物中Aamp是高度保守的。然而,在地松鼠和喜马拉雅旱獭中发现并保存了高度保守的共线性位点(图5 B),进一步的系统发育分析表明,ΨAamp和Aamp属于两个独立的分支,基于序列分歧插入时间的估算暗示假基因的插入事件发生在约22.64~25.40 MYA,在喜马拉雅旱獭和地松鼠分化之前(图 5 C)。这些发现支持古老的假基因对于松鼠具有特异性并从共同的祖先遗传而来这一假设,然而,与地松鼠相比,喜马拉雅旱獭显示Aamp和ΨAamp之间的序列距离较小,这表明存在选择性压力来维持旱獭的遗传元素。
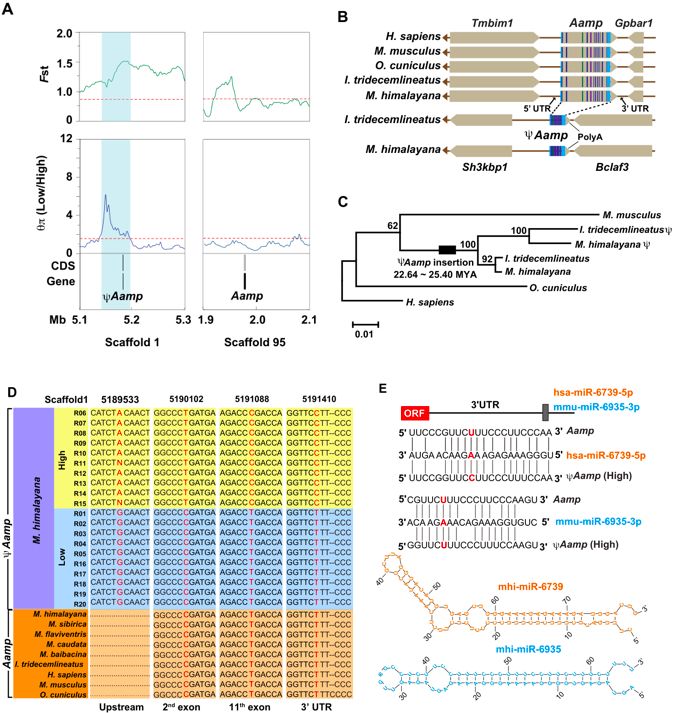
图5、ΨAamp高海拔适应性中的演化和潜在作用
作为重要的调节区域,3’UTR是动物中经典的靶向miRNA基因座,该区域的突变可能影响mRNA丰度和表型分化,在高海拔的群体中上游区域,研究人员发现了2nd外显子, 11th外显子、ΨAamp的3’UTR区中的4个固定的替代(图5 D),在3’UTR区内,发现了完全保守的匹配,其跨越T> C替换,对于ΨAamp/ Aamp-靶向hsa-miR-6739-5p和mmu-miR-6935-3p(图5 E)。Pre-miRNA结构模拟显示它们在喜马拉雅旱獭基因组中具有稳定的发卡结构(图5 E),此外,在喜马拉雅旱獭肝脏中检测到了ΨAamp和 Aamp的转录。在高海拔的群体中Aamp的表达水平降低。这些结果支持这样的假设:ΨAamp中 3’UTR区的突变可能降低miRNA组合效率,从而影响Aamp在高海拔群体中的稳定性。
讨论
在本研究中,成功组装了一只雄性喜马拉雅旱獭基因组草图,为研究其基因组进化和独特的生理特征提供了宝贵的基因组资源,包括缺氧适应和能量稳态。系统发育分析表明,喜马拉雅旱獭的形成可能是由于青藏高原的隆起造成的。在最后一次冰川季期间,喜马拉雅旱獭很好地适应了极端环境。
对高海拔和低海拔群体的遗传多样性有了更深入的了解,丰富了哺乳动物极端环境适应性的认知。在Slc25a14基因座处发现的强烈的群体分化信号和明显的基因型差异意味着其在喜马拉雅旱獭对缺氧的适应性反应中起到了重要的作用,Slc25a14通过调节线粒体功能和氧化剂产生具有神经保护作用,同时还涉及维持代谢率和适应性体温调节。有趣的是,研究人员发现ΨAamp在高海拔群体中通过竞争性miRNA介导Aamp表达可能具有生物学活性,Aamp以多种细胞类型表达,主要定位于血管内皮细胞的细胞质和膜中。Aamp的敲除损害了VEGF诱导的内皮细胞迁移和血管生成,高海拔群体中Aamp的下调可能是在极度缺氧条件下防止过度血管生成的保护性策略。由于喜马拉雅旱獭是一种冬眠动物,高比例的受选择基因的筛选支持了高原适应性和冬眠之间的密切联系,这些基因在冬眠/觉醒周期中差异表达。与此同时,干细胞信号通路的补体和凝血级联和多能性可能与大脑的抗寒性保护策略有关。
喜马拉雅旱獭基因组的特征及本文提供的其它旱獭的全基因组重测序数据为阐明其进化事件和环境适应性提供了广泛的视角。其独特遗传特征的鉴定将有助于其潜在的医学应用。
如果您的项目有任何问题,欢迎点击下方按钮咨询我们,我们将免费为您设计文章方案,并赠送您100元生信培训在线课程代金券,助您勇攀科研高峰!
相关阅读
- 百迈客助力四倍体海岛棉及陆地棉基因组发表于Nature Genetics
- 百迈客助力甘蔗基因组Nature Genetics发表
- 亚洲棉科研成果Nature Genetics在线发表
- 《Molecular Plant》百迈客大白菜遗传进化研究








 京公网安备 11011302003368号
京公网安备 11011302003368号