
2018年12月4日,华中农业大学作物遗传改良国家重点实验室张献龙课题组首次通过三代测序(PacBio)+光学图谱(BioNano)+Hi-C技术完成了异源四倍体陆地棉(Gossypium hirsutum)和海岛棉(Gossypium barbadense)基因组组装。该研究成果刊登于Nature Genetics,文中利用北京百迈客生物科技有限公司的三代PacBio测序组装及Hi-C染色体挂载技术,组装获得了高质量栽培种异源多倍体棉基因组。>>下载文献全文
中文题目:三代异源四倍体陆地棉和海岛棉基因组破译;
发表杂志:Nature Genetics;
合作单位:华中农业大学作物遗传改良国家重点实验室;
摘要
异源四倍体陆地棉和海岛棉(Gossypium hirsutum和Gossypium barbadense)长期以来一直在世界范围内种植,由于其具有天然可再生纺织纤维。虽然以前利用二代测序技术组装的陆地棉和海岛棉基因组在棉花研究中应用较高,但是其基因组高度片段化且不完整。本研究中进行了异源四倍体基因组的升级,利用了三代测序组装技术(single-molecule real-time sequencing)+光学图谱(BioNano optical mapping)+Hi-C染色体挂载技术(high-throughput chromosome conformation capture techniques),实现了异源四倍体陆地棉G. hirsutum acc. Texas Marker-1 (TM-1) 和海岛棉G. barbadense acc. 3-79基因组的组装。与之前的二代基因组相比,三代陆地棉和海岛棉基因组具有高度连续性,高度重复区(如着丝粒)具有更高的完整性。比较基因组学分析确定了广泛的结构变异可能发生在多倍化后,在14条染色体臂内和臂间的倒位十分突出。研究中构建了渐渗系群体,以导入从海岛棉到陆地棉的有利染色体片段。从而使得研究人员可以识别与优质纤维质量相关的13个数量性状基因座。 这些资源将加速棉花的进化和功能基因组研究,并为未来的纤维改良育种计划提供新的信息。
研究背景
棉花是世界上*大的天然纺织纤维来源,每年纤维产量的90%以上来自异源四倍体棉花(G. hirsutum和G. barbadense),它起源于大约1-2百万年前的异源多样化事件,随后是数千年的不对称亚基因组选择。陆地棉(G. hirsutum)由于其高产而在全世界种植。G. barbadense以其卓越的纤维质量而受赞誉。为了培育产生纤维更长,更细和更强韧的陆地棉(G. hirsutum)品种,一种合理有效的方法是将海岛棉(G. barbadense)的优良纤维性状引入陆地棉。基因组学启动的育种策略需要对基因组组织进行详细而有力的理解。
材料方法
材料:高度纯合陆地棉(Texas Marker-1;TM-1) ;海岛棉(3-79);
基因组denovo策略:三代测序组装(PacBio RS II,SMRT)+光学图谱(BioNano optical mapping)+Hi-C染色体挂载;
研究方法:基因组组装:Canu (version 1.3) ,BLASR (version 1.3.1) ,BWA (version 0.7.10-r789) ,Pilon (version 1.22) ;光学图谱纠错:核酸内切酶Nt.BssSI23,AutoDetect,IrysSolve;Hi-C染色体挂载:核酸内切酶HindIII,BWA(version 0.7.10-r789),LACHESIS,HiC-Pro;基因组完整性评估:BUSCO评估;TE注释:PASTEClassifier (version 1.0);RepeatMasker (version 4.0.6);基因预测和注释:Genscan,Augustus (version 2.4),GlimmerHMM (version 3.0.4),GeneID (version 1.4)和SNAP (version 2006-07-28);GeMoMa (version 1.3.1);假基因组预测:GenBlastA (version 1.0.4),GeneWise (version 2.4.1);
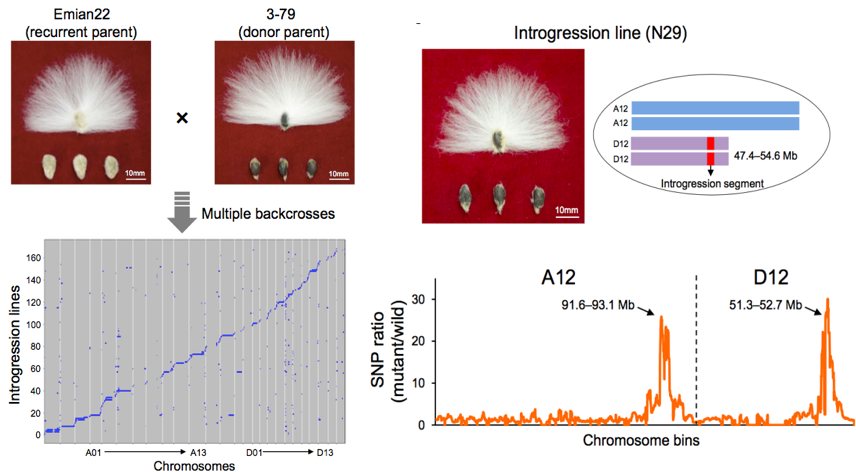
着丝粒区域鉴定:blastn,SPSS software (version 17.0) ;基因组共线性分析:MUMmer (version 3.23),GATK(version 3.1.1),Samtools(version 0.1.19) ,MCScanX package;结构变异检测:MUMmer3 (version 3.23);二倍体棉重测序SNPs鉴定:Trimmomatic (version 0.32),BWA;168个CSSLs群体SNPs鉴定:染色体片段置换系(CSSLs)的构建(图1),测序深度(6X),插入片段:350 bp,BWA,GATK和Samtools;CSSLs群体QTLs定位与表达分析:QTL IciMapping (version 4.0) ;TopHat2 (version 2.0.13) ;Cufflinks (version 2.2.1);STRUCTURE (version 2.3) ;TASSEL software (version 5.0) ;
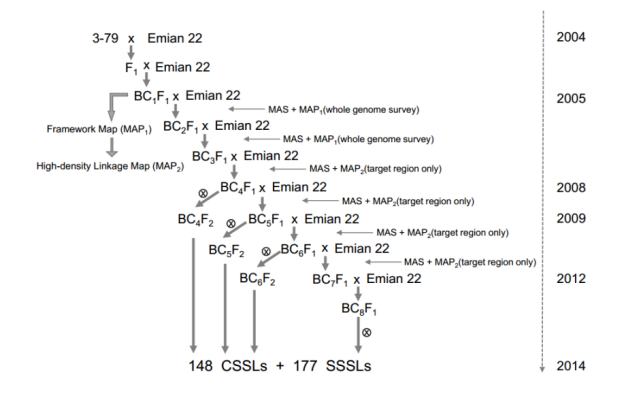
图1 棉花渐深系构建流程
研究结果
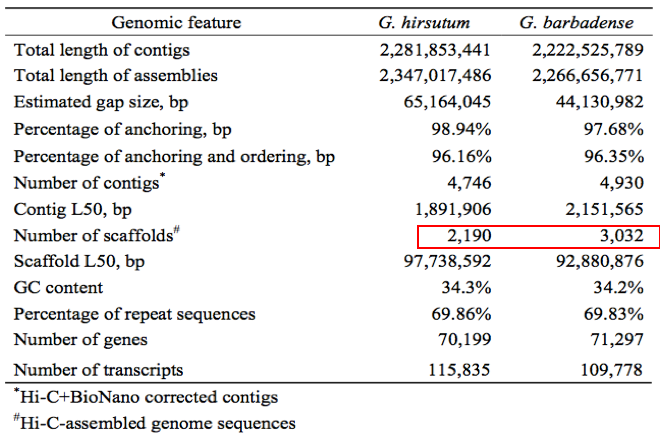
组装结果验证:将重新组装的陆地棉与海岛棉基因组与已发表的遗传图谱进行比对,结果显示,每条染色体都具有高度共线性(Gossypium hirsutum 98.86%;Gossypium barbadense 96.92%);进一步通过36个已有的BAC文库及二代Illumina和mate-pair文库的回比评估,并通过对陆地棉的BUSCO数据集中的1,440个高度保守的核心蛋白中的1,415个(98.2%)和对海岛棉的1,420个(98.6%)的鉴定,支持了基因区组装的完整性。与之前发表的二代基因组相比,基因组连续性显著提高(陆地棉高出55倍,海岛棉高出90倍),进一步实现了gap填充,基因组高杂合区的√确组装。本研究中陆地棉与海岛棉基因组的迭代更新为后续四倍体棉花的研究提供了新版参考基因组。
表1 陆地棉和海岛棉基因组组装注释
染色体结构变异分析:高质量的参考基因组使得研究人员能够通过对两种种质的直接比较基因组分析来鉴定大的结构变异。发现有170.2 Mb的基因组序列被鉴定为G. hirsutum和G. barbadense之间的反转,包括120.4 Mb的At亚基因组和49.8 Mb的Dt。有趣的是,在异染色质中,第四条和第十一条染色体中显示出了染色体臂内倒位。研究中在A06染色体中发现了4个大的倒位变异,包括3个染色体臂内倒位(in1, in3 and in4)和1个染色体臂间倒位(in2),通过Hi-C数据在断点周围离散的染色质相互作用(图3),突出了Hi-C技术识别大规模染色体重排的优势。光学图(BioNano optical maps)谱数据进一步支持了这些反转断裂位点(图3)。此外,发现在D12染色体上,存在1个大的染色体臂间倒位。在棉花中这些染色体臂间/染色体臂内的大量变异需要进一步探索其生物学功能,如在拟南芥,小麦和人中所述。研究人员同时还检测到3,820个染色体易位(1,074个染色体内易位,占据3.8 Mb;2,746个染色体间易位,占6.8 Mb)。
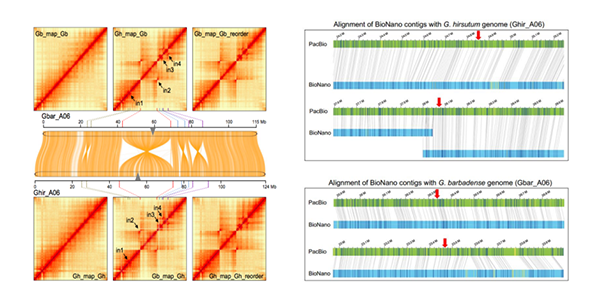
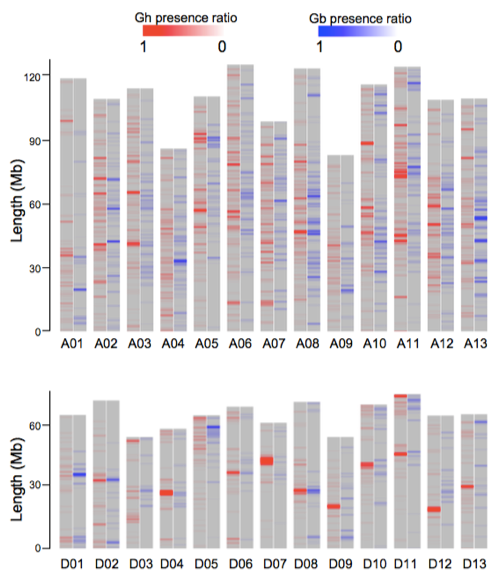
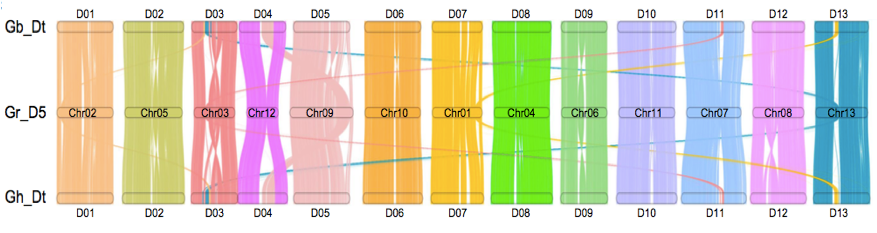
四倍体陆地棉和海岛棉基因组的组装使得研究人员能够进一步探索四倍体棉亚基因组和其二倍体祖先之间的基因组差异。首先通过对具有D型基因组的13份二倍体材料进行重测序分析,分析显示在两种四倍体棉D-亚基因组具有相同的二倍体祖先种雷蒙德氏棉G. raimondii(D5 genome,D亚基因组供体);进而利用二倍体雷蒙德氏棉G. raimondii直接进行基因组比较分析,发现四倍体陆地棉和海岛棉与雷蒙德氏棉相比,都有一些独特的结构变异,如在海岛棉染色体D05和陆地棉D12中均存在大的染色体臂间倒位,暗示这些变异出现在多倍化之后(图5)。研究人员还观察到两个四倍体相对于G. raimondii共有一些结构变异,例如染色体D09中两个四倍体发生了大的反转(图5)。研究中同时运用了二倍体祖先种亚洲棉G. arboreum(A2 genome,A亚基因组供体)的Hi-C数据,与陆地棉和海岛棉A亚基因组进行比对,检测棉由二倍体到四倍体多倍化过程中发生的结构变异,Hi-C具体矩阵图显示在13条染色体中发生了大规模的染色体重排,其中发部分变异为两个四倍体的棉的A亚基因组共有。研究中发现染色体A06中*大的染色体臂间倒位(in2)在陆地棉中是特有的(图3 左),表明这种结构变异可能在染色体多倍化后发生。因此得出结论,二倍体棉花中的A基因组在异源多倍化后被重组,导致不同四倍体(陆地棉和海岛棉)中发生了大染色体倒位。
通过研究发现了在陆地棉(G. hirsutum acc. TM-1)和海岛棉(G. barbadense acc. 3-79)间存在广泛的遗传变异,进一步推测这些变异的一部分可能是造成表型差异的原因,包括纤维性状。为了利用这些变异进行定向育种,研究人员构建了一个渐渗系群体,旨在引入有利的变异,控制从G.barbadense到G. hirsutum等重要农艺性状的形成,如纤维质量。研究人员通过分子标记对168份渐渗系材料进行测序,并鉴定了涵盖所有26条染色体的466个基因渗入片段(图6)。研究人员发现了在染色体D12中含有渐渗片段的渐渗系,其具有有限的绒毛纤维,类似于其供体亲本G.barbadense 3-79(图6,图7上),基因渗入片段的位置与无绒天然突变体G. hirsutum Xuzhou142fl的图谱测序所示的位置相同,然而其遗传基础以前未被充分了解(图7下)。这些结果表明,陆地棉(G. hirsutum)中无绒毛突变体的遗传变异与海岛棉(G. barbadense)中的数量性状基因座(QTL)共定位。该渐渗片段与天然纤维突变体的特征将有助于比较分析海岛棉和陆地棉之间的绒毛纤维起始机制。
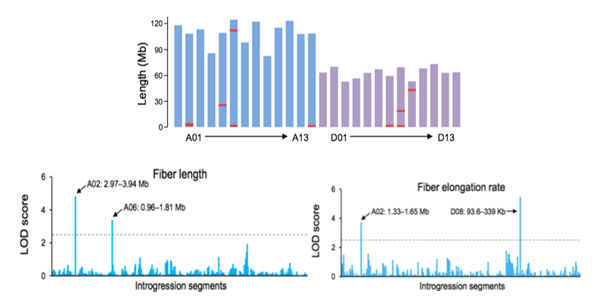
为了鉴定海岛棉(G. barbadense)中优质纤维质量的有益等位基因,研究人员对渐渗群体中的纤维品质相关性状进行了QTL分析。研究中共计鉴定了5个性状的13个QTLs位点,其中控制纤维长度位点2个,控制纤维强度位点4个,马克隆值位点2个,纤维伸长率位点2个,纤维均匀度位点3个(图8 a-c)。在这些QTLs位点中,9个位点之前未被鉴定出,通过检验13个QTLs中的基因表达水平,研究人员检测到了235个在纤维发育过程中高度表达的基因,同时还整合了基因组变异数据来预测候选基因,而这些基因值得进一步进行精细定位以确认对这些性状具有重要影响的基因。研究人员发现A02染色体上的1个QTL位点与纤维长度相关,在这个QTL中,一个未鉴定过的基因(Ghir_A02G003440),编码预测的糖基磷脂酰肌醇锚定的脂质转移蛋白,该基因在纤维伸长期的表达水平和深入系中纤维长度呈负相关,并可能与海岛棉中长纤维的发育相关。这些QTL数据为海岛棉基因组片段的详细功能分析提供了框架,并应通过基因渗入育种进一步开发具有优质性状的栽培棉花。
总结
回顾整个2018年,百迈客NG文章收获满满,于2018年5月与中国农业科学院棉花研究所杜雄明研究员合作成功完成了亚洲棉基因组的升级,又在2018年10月与福建农林大学基因组研究中心明瑞光课题组合作,完成了同源多倍体甘蔗基因组密码的破译。同时,于2018年巨资投入,引进三代测序Nanopore单分子纳米孔测序仪,成为了国内动植物基因组denovo研究第一家。
另外,还可以获赠100元在线培训课程代金券!
相关阅读








 京公网安备 11011302003368号
京公网安备 11011302003368号