一般做完RNA的差异表达分析后,会有一堆的结果与数据。那么,怎么样让你的分析结果在paper中以更加直观形象(好看炫酷)的方式展示出来呢?
前面我们讲过热图函数pheatmap()参数讲解。下面我们介绍另外一种展示方式:火山图(Volcano Plot)。
首先,我们要注意两个重要概念:
FDR(False Discovery Rate)
在差异表达分析过程中,采用统计学方法对原有假设检验得到的p值进行校正,并最终采用校正后的p值,即为FDR。
FC(Fold Change)是两个/组样品间基因表达水平的比值,是表示差异倍数的变量。
第1步 数据准备
差异表达分析结果数据示例:第一列为基因名称,第二列为差异表达分析算法得到的p值,第三列为矫正后的p值,即FDR,第四列为log2FC值。绘制火山图只需要最后两列就行。
数据准备
| gene_symbol | PValue | FDR | log2FC |
|---|---|---|---|
| RAB22A | 0.731093877 | 0.915536119 | -0.043559185 |
| DDX20 | 0.455372338 | 0.779014647 | -0.099300804 |
| CILP2 | 0.415756716 | 0.749566482 | 1.420386794 |
| 项目 #4 | 0.003274032 | 0.056770217 | 6.182603532 |
| RP11-618P17.4 | 0.351633414 | 0.709160705 | -0.108742132 |
第2步 R代码画图

最后绘制出来的火山图如下,是不是比干巴巴地在文章里面贴一个差异表达分析结果的表格好看多了?
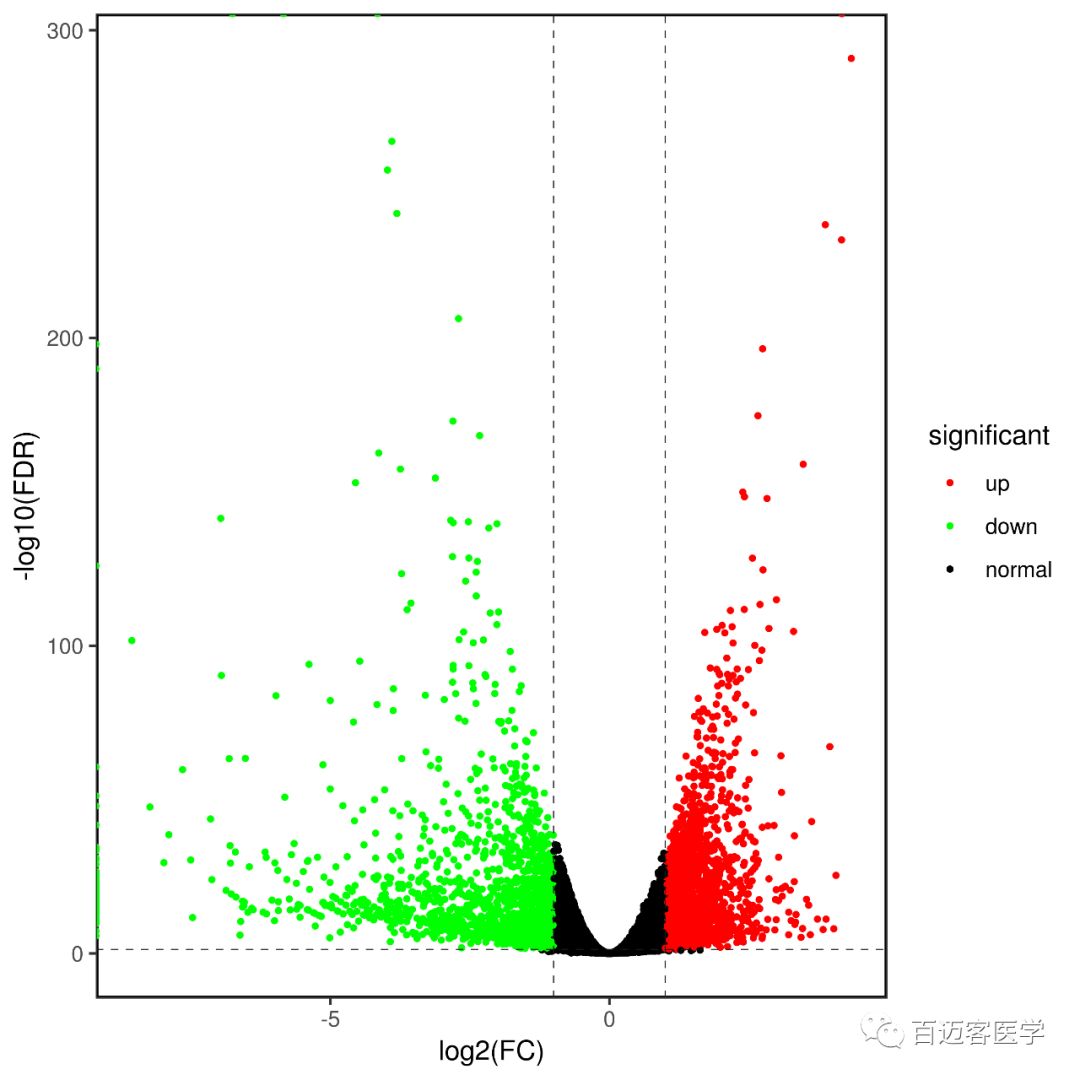 注:
注:
一个点表示一个基因, 横坐标表示某一个基因在两样品中表达水平差异倍数的对数值;纵坐标表示FDR的负对数值。横坐标绝对值越大,说明基因在两样品间的表达水平倍数差异越大;纵坐标值越大,表明差异越显著,筛选得到的差异表达基因越可靠。图中绿色的点代表表达下调基因, 红色的点代表表达上调基因, 黑色代表非差异表达基因。

关注百迈客云,随时随地学习
最近文章







 京公网安备 11011302003368号
京公网安备 11011302003368号