1、打开脚本文件
source(‘xxx’) #’xxx’脚本路径, 例如’C:/Users/R/Desktop/qq.R’
2、创建Seurat对象并绘制空间聚类图
FilePath : 存放“barcode.tsv.gz、barcode_pos.tsv.gz、feature.tsv.gz、matrix.mtx.gz”的文件夹路径
barcode_pos_file : “barcode_pos.tsv.gz”的文件路径
out_path : 输出文件目录
png_path : he染色(.png格式),如果是 .tiff 格式需要进行格式转换,注意转换时分辨率可适当调小,以避免 .png 格式的图片太大而读取失败
需注意的是Seurat对象名称必须是“object”
object <- Create_object(
FilePath = ‘E:/AAAWork/BSTViewer_project/subdata/L13_heAuto/’,
barcode_pos_file = ‘E:/AAAWork/BSTViewer_project/subdata/L13_heAuto/barcodes_pos.tsv.gz’,
out_path = ‘C:/Users/R/Desktop/temp/Cluster/’,
png_path = ‘C:/Users/R/Desktop/temp/he.png’,
min.cells = 10, #一个基因至少在n个细胞中表达才被保留,可自行调整,默认值10
min.features = 100, #一个细胞至少有n个基因才被保留,可自行调整,默认值100
dims = 1:30, #选择多少pc进行后续分析,可自行调整,默认1:30
resolution = 0.5, #设置下游分析的“粒度”,值越高得到的聚类数目越多,可自行调整,默认0.5
point_size = 3, #点的大小,可自行根据矩阵文件level调整,level越小需设置的值越小
width = 12, #输出图片的宽度,可自行调整,默认12
height = 5, #输出图片的高度,可自行调整,默认5
Cluster = T, #是否进行聚类分析,默认F
label = T #是否输出带标签的聚类图
)
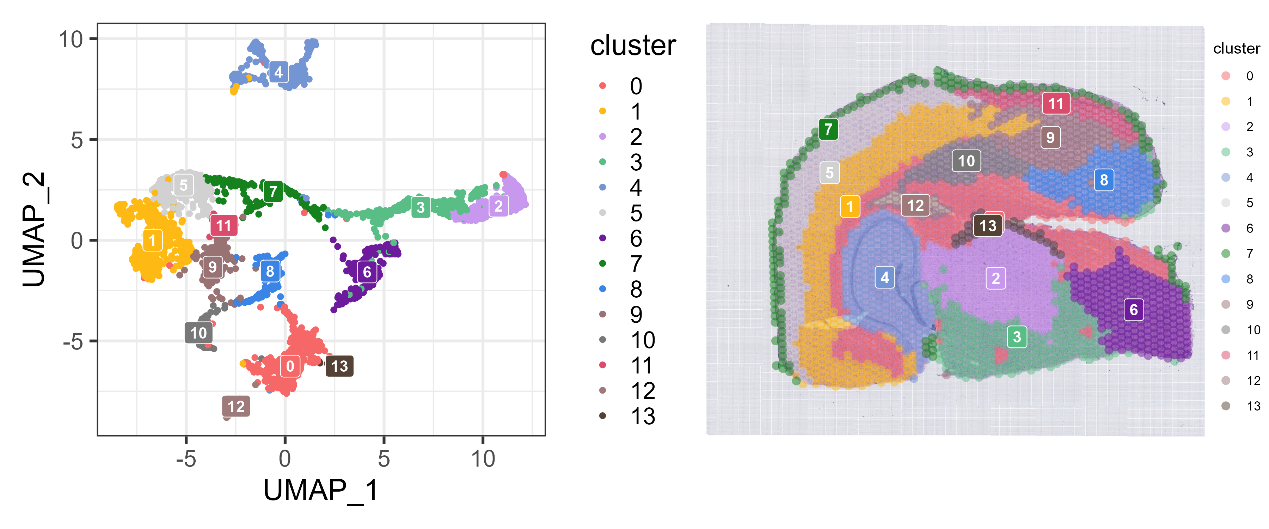
umap_cluster_label
3、UMI统计
object <- Create_object(
FilePath = ‘E:/AAAWork/BSTViewer_project/subdata/L13_heAuto/’,
barcode_pos_file = ‘E:/AAAWork/BSTViewer_project/subdata/L13_heAuto/barcodes_pos.tsv.gz’,
out_path = ‘C:/Users/R/Desktop/temp/UMI_stat/’,
png_path = ‘C:/Users/R/Desktop/temp/he.png’,
point_size = 3, #同上
width = 12, #同上
height = 5, #同上
UMI_stat = T) #是否统计UMI
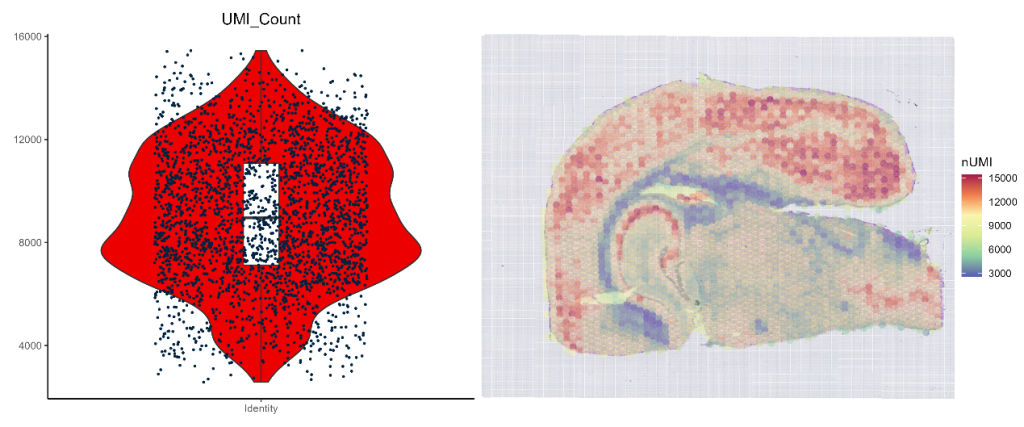
UMI_viol_heatmap
4、nFeature 统计
object <- Create_object(
FilePath = ‘E:/AAAWork/BSTViewer_project/subdata/L13_heAuto/’,
barcode_pos_file = ‘E:/AAAWork/BSTViewer_project/subdata/L13_heAuto/barcodes_pos.tsv.gz’,
out_path = ‘C:/Users/R/Desktop/temp/Gene_stat/’,
png_path = ‘C:/Users/R/Desktop/temp/he.png’,
point_size = 2, #同上
width = 12, #同上
height = 5, #同上
nFeature_stat = T) #是否统计nFeature
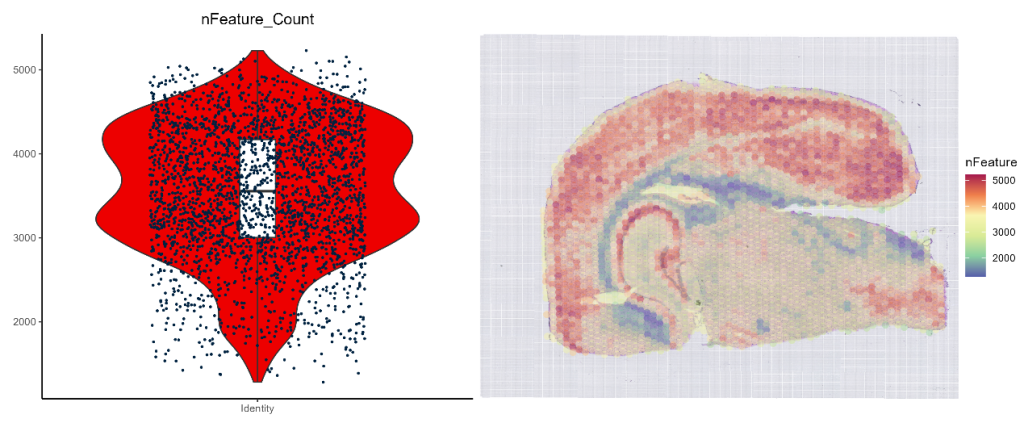
nFeature_viol_heatmap
5、输出每个cluster的marker gene,并绘制单个基因的表达热图
object <- Create_object(
FilePath = ‘E:/AAAWork/BSTViewer_project/subdata/L13_heAuto/’,
barcode_pos_file = ‘E:/AAAWork/BSTViewer_project/subdata/L13_heAuto/barcodes_pos.tsv.gz’,
out_path = ‘C:/Users/R/Desktop/temp/Single_gene_1/’,
png_path = ‘C:/Users/R/Desktop/temp/he.png’,
point_size = 2, #同上
Gene_stat = T, #是否进行mark gene绘制
top_gene = 1, #每个cluster取top多少mark gene, 可自行调整,值不宜设置太大
min.pct = 0.25, #一个基因在任何两群细胞中的占比不能低于多少, 可自行调整
logfc.threshold = 0.25, #差异倍数阈值, 可自行调整
markpic_width = 8, #小提琴图和tsne图宽
markpic_height = 12, #小提琴图和tsne图长)
热图列表
6、绘制某个或某些基因聚类图
object <- Create_object(
FilePath = ‘E:/AAAWork/BSTViewer_project/subdata/L13_heAuto/’,
barcode_pos_file = ‘E:/AAAWork/BSTViewer_project/subdata/L13_heAuto/barcodes_pos.tsv.gz’,
out_path = ‘C:/Users/R/Desktop/temp/Test/Single_gene_2/’,
png_path = ‘C:/Users/R/Desktop/temp/he.png’,
point_size = 2.6, #同上
Gene_stat = T, #是否进行mark gene绘制
Custom_gene = T, #是否进行自定义gene绘制
alpha_continuous = c(0.5,1) #根据基因表达量调整透明度范围
gene_list = c(‘Hpca’)) #绘制的基因名,可以输入多个
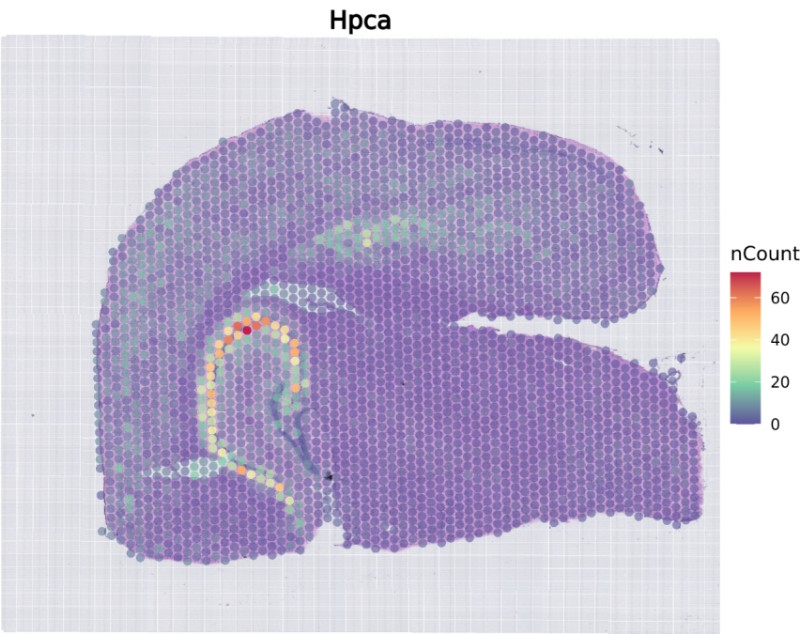
Hpca
选择黑色背景突出显示
object <- Create_object(
FilePath = ‘E:/AAAWork/BSTViewer_project/subdata/L13_heAuto/’,
barcode_pos_file = ‘E:/AAAWork/BSTViewer_project/subdata/L13_heAuto/barcodes_pos.tsv.gz’,
out_path = ‘C:/Users/R/Desktop/temp/Test/Single_gene_2/’,
png_path = ‘C:/Users/R/Desktop/temp/he.png’,
point_size = 2.6, #同上
Gene_stat = T, #是否进行mark gene绘制
Custom_gene = T, #是否进行自定义gene绘制
dark_background = T, #黑色背景
gene_list = c(‘Hpca’)) #绘制的基因名,可以输入多个
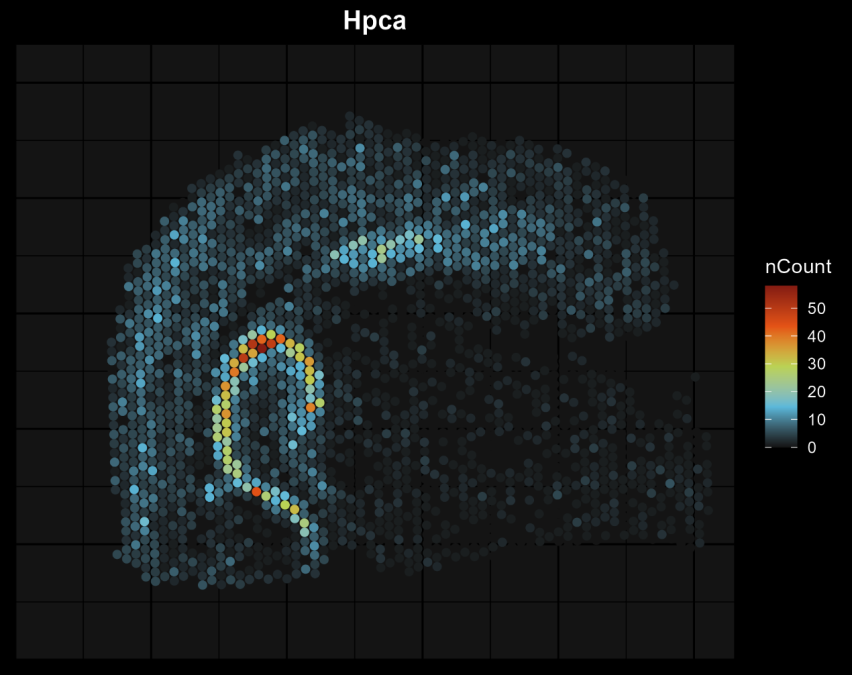
Hpca
7、绘制单个cluster聚类图
object <- Create_object(
FilePath = ‘E:/AAAWork/BSTViewer_project/subdata/L13_heAuto/’,
barcode_pos_file = ‘E:/AAAWork/BSTViewer_project/subdata/L13_heAuto/barcodes_pos.tsv.gz’,
out_path = ‘C:/Users/R/Desktop/temp/TestL6/single/’,
png_path = ‘C:/Users/R/Desktop/temp/he.png’,
point_size = 2, #同上
Single_cluster = T #是否绘制单个cluster图)
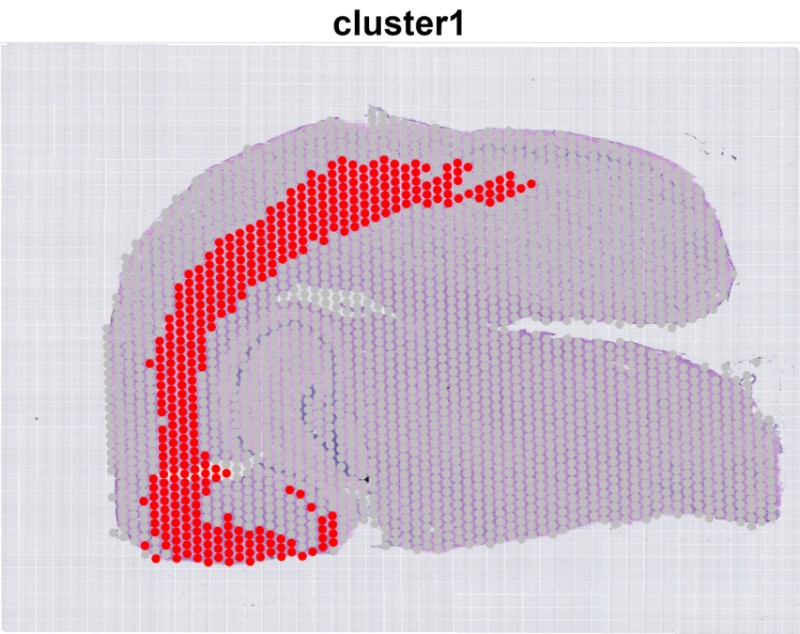
cluster1
单个cluster列表







 京公网安备 11011302003368号
京公网安备 11011302003368号