RNA 速率这个概念最初是在2018年提出的,可以通过测序结果与mRNA剪切动力学相关联,利用新转录的未剪接的前体 mRNA 和成熟的剪接 mRNA 可以在常见的scRNA-seq、stRNA-seq 流程中区分的事实,可以恢复定向动态信息,从而探索方向轨迹的演化。目前RNA速率分析可广泛应用于免疫微环境、发育模式、遗传进化等方向。那么今天我们一起来学习一下RNA速率分析流程及方法有哪些。
RNA速率分析1、获得重复序列注释文件
软件依赖:RMBlast、TRF ——> conda 安装即可
数据依赖:Repbase数据库
链接:https://pan.baidu.com/s/1F4E5-6XaVni1I_S97TEIDg 提取码:zq7t
下载后解压得到一个Libraries的文件夹
Repeatmasker下载:wget http://www.repeatmasker.org/RepeatMasker-open-4-0-6.tar.gz
下载后解压,将第二步 数据依赖 中Libraries文件夹下的内容拷贝到Repeatmasker目录下Libraries文件夹中
cp Libraries/* /path/to/Repeatmasker/Libraries/
安装可参考以下教程:
基因组重复序列检测:RepeatMasker的安装及使用可自行搜索安装
运行Repeatmasker:
/share/nas1/guochao/biosoftware/RepeatMasker/RepeatMasker -pa 60 -species “Mus musculus” -poly -html -gff -dir repeat /path/to/genome.fa
-species : 物种拉丁名
-dir : 输出结果目录,需提前创建好!
RNA速率分析2、构建loom文件
安装velocyto:pip install -i https://pypi.tuna.tsinghua.edu.cn/simple velocyto
具体安装方法可自行搜索
处理bam文件
perl脚本:
链接:https://pan.baidu.com/s/1F4E5-6XaVni1I_S97TEIDg 提取码:zq7t
perl am_add_flag.pl <BSTViewer_project/subdata/L1_heAuto/barcodes.tsv.gz> <sample/barcode.tsv> <BSTViewer_project/level_matrix/level_1/barcodes_cluster.tsv.gz>
:BSTMatrix输出结果 01.fastq2BcUmi目录下xxx.bc_umi_read.tsv.id
: BSTMatrix输出结果05.AllheStat目录下BSTViewer_project/subdata/L1_heAuto/barcodes.tsv.gz
<sample/barcode.tsv>: BSTMatrix输出结果样本名称目录下barcode.tsv文件
:BSTMatrix输出结果05.AllheStat目录BSTViewer_project/level_matrix/level_1/barcodes_cluster.tsv.gz
: BSTMatrix输出结果 03.Umi2Gene 目录下 xxxAligned.sortedByCoord.out.bam
: 输出路径
运行velocyto:
velocyto run -o ./ -m /path/mouse/mus_repeat.gtf new.bam ../mouse/genes.gtf
-o : 输出路径
-m : 重复序列注释文件(第一步获得)
R转化loom文件为h5ad文件
library(Seurat)
library(SeuratWrappers)
library(SeuratDisk)
loom_data bm bm[[“RNA”]] bm bm bm bm bm
DefaultAssay(bm) <- “RNA”
SaveH5Seurat(bm, filename = “test.h5Seurat”)
Convert(“test.h5Seurat”, dest = “h5ad”)
scvelo RNA速率分析
adata = scv.read(‘/path/test.h5ad’)
scv.pp.filter_and_normalize(adata, min_shared_counts=20, n_top_genes=2000)
scv.pp.moments(adata, n_pcs=30, n_neighbors=30)
scv.tl.velocity(adata)
scv.tl.velocity_graph(adata)
scv.set_figure_params(dpi=300)
scv.pl.velocity_embedding_stream(adata, basis=”umap”, size= 10, alpha=0.6, color=”seurat_clusters”)
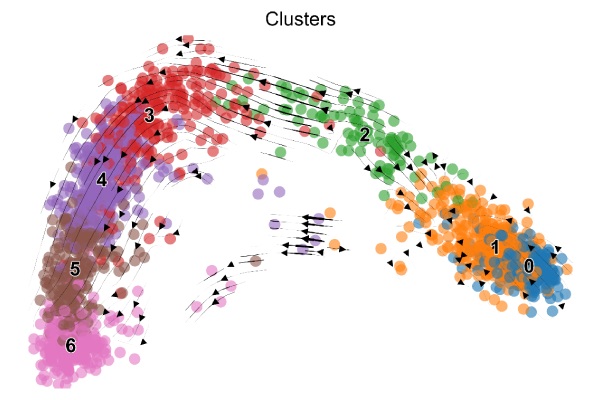
RNA速率分析







 京公网安备 11011302003368号
京公网安备 11011302003368号