在过去视网膜疾病的全基因组关联研究(GWAS)发现常见的变异主要在基因组非编码区域,但由于先导变异区域的局部连锁不平衡(LD),阻碍了特定关联基因和变异的识别,因此阐明与性状相关的遗传变异如何影响调节基因表达网络,并因此影响表型,亟需了解相关组织和细胞类型的3D基因组拓扑结构。
近日,来自美国NIH国家眼科研究所Anand Swaroop团队通过Hi-C技术绘制了人类视网膜细胞染色质三维结构图谱,揭示了人类视网膜中超级增强子(SEs)的3D染色质组织的特性,并将得到的视网膜基因组调控网络与通过GWAS识别的老年性黄斑变性(AMD)和青光眼相关的数量性状基因座(quantitativetraitloci, QTL)和遗传变异相结合,揭示了顺式调控元件(CREs)的靶标,表明了人类视网膜中SEs的3D染色质组织的特性。此次人类视网膜高分辨率基因组结构的研究为组织特异性功能的遗传调控提供了新见解,并能够解剖常见的致盲疾病表型。
Hi-C技术绘制新的老年性黄斑变性和青光眼相关视网膜高分辨率基因组三维结构图谱
High-resolution genome topology of humanretina uncovers super enhancer-promoter interactions at tissue-specific and multifactorial disease loci
发表杂志:Nature Communications
影响因子:17.69
发表日期:2022年10月7日
发表单位:美国NIH国家眼科研究所
研究背景
在不同组织和细胞类型中人类基因组染色质结构增强子-启动子相互作用建立了独特的时空基因表达模式。非编码遗传变异可以通过染色质空间构象从而调控基因表达来影响细胞表型。为了阐明人类视网膜的基因组调控,利用Hi-C技术以高分辨率揭示染色质并与超级增强子(SE)、组蛋白标记、CTCF结合和选择转录因子相互作用,表明与边缘SE的TAD相比,具有中心SE的拓扑相关结构域(TAD)表现出更强的绝缘性,并与视网膜基因的接触增强。全基因组表达数量性状位点(eQTL)分析与拓扑图相结合,揭示了100个eQTL和与视网膜神经变性相关的相应eGene之间的联系,并发现了与年龄相关的黄斑变性和青光眼相关的易感变异的候选基因。对人类视网膜高分辨率基因组结构的研究为组织特异性功能的遗传控制提供了见解,提出了缺失遗传性的模式,Hi-C组学技术为识别视网膜相关疾病表型提供有利工具。
研究设计
材料:一共取材5名患者捐赠视网膜组织(2名女性,3名男性,年龄65-77岁);Hi-C互作取3名男性和1名女性视网膜组织以及HCT116细胞系;CUT&RUN取1名75岁的女性外周视网膜组织;ATAC-seq取2名女性和3名男性视网膜组织。
方法:Hi-C互作(5kb)、ATAC-seq、Cut&Run、ChIP-seq(数据集:GSE13731134)、RNA-seq(数据集)
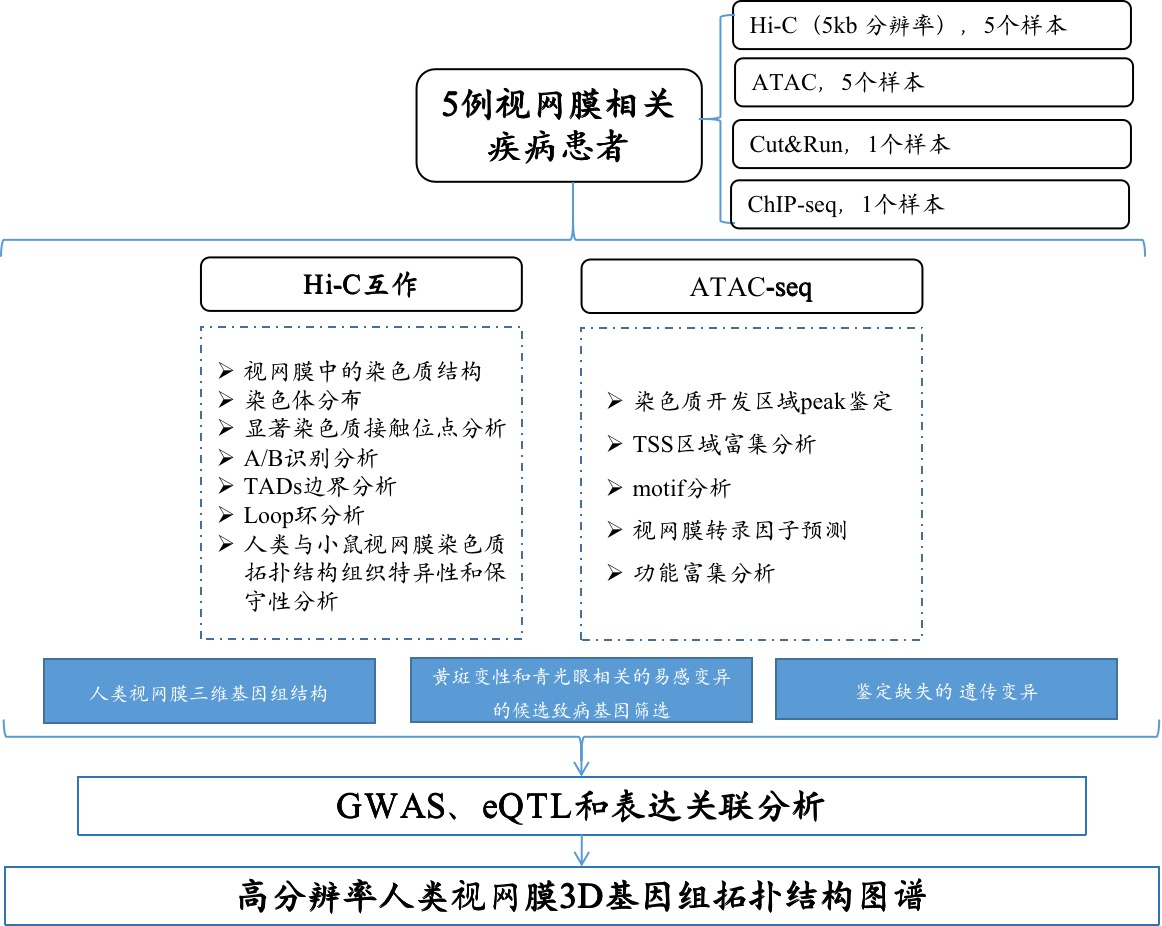
文章亮点
1)本文整合表观调控全面的技术,利用Hi-C互作、ATAC-seq、Cut&Run、ChIP-seq等多组学不仅重构了视网膜基因组拓扑结构,还深入剖析了三维结构对基因调控的网络;
2)视网膜调控基因组拓扑结构与AMD和青光眼相关遗传变异的首次详细整合;
3)高分辨率的基因组结构图像(5kb),深入研究常染色质A/异染色质B区域、TAD边界、Loop形成的变化;
4)利用GWAS、eQTL等疾病性状关联分析建立与视网膜神经变性相关的相应eGene之间的联系。
主要研究内容
1、Hi-C测序以5kb高分辨率识别人类视网膜中的染色质空间构象
利用Hi-C技术检测了4名眼科相关疾病患者的视网膜组织,以超高的分辨率(5kb)的重新绘制了人类视网膜细胞染色质的组织(每个视网膜样本的测序深度约为3亿对reads,HCT116的深度约为5000万对read)。分析表明Hi-C数据在样本间表现出高度的相似性,所有常染色体的分层相关系数(SCC)>0.97。整合所有样本的数据一共获得了11.48亿对reads,其中有效互作reads的比例高达95%,不同染色体之间的互作trans占23.6%。
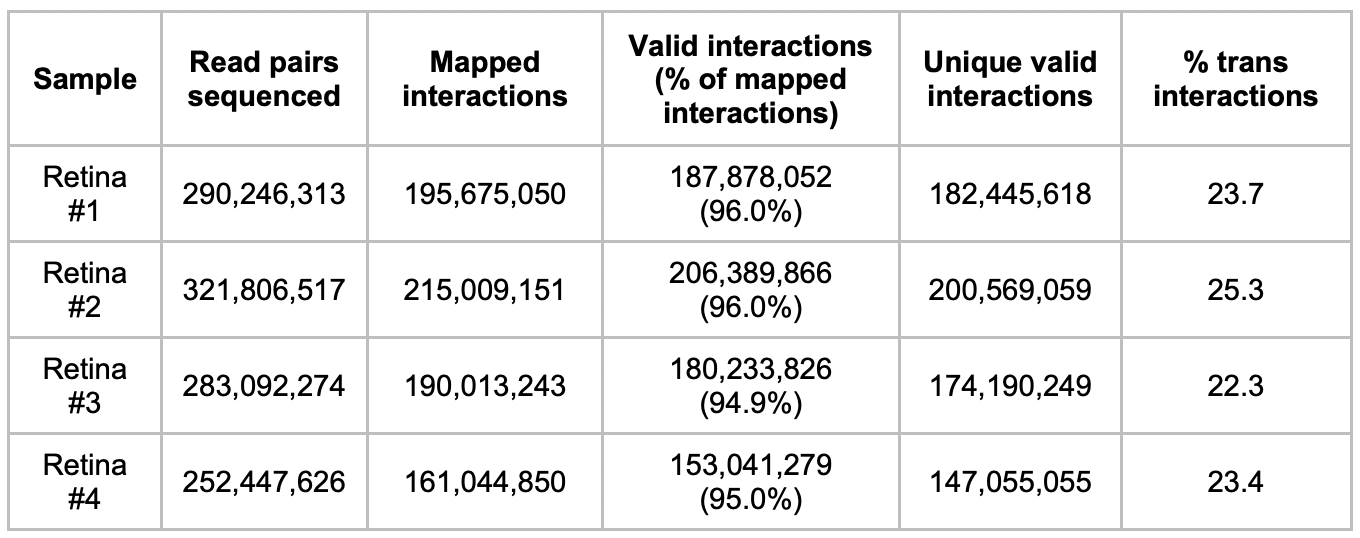
Hi-C测序数据统计(补充表1)
此次研究总共获得了7.04亿个有效染色质互作,这些染色质互作平均分布在染色体之间和染色体之内。
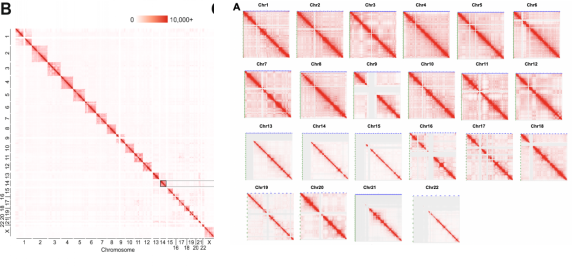
Hi-C检测每个常染色体上染色体内和染色体间的互作
本文Hi-C以3 kb的高分辨率识别出67,841个显著的染色质互作和2948个拓扑关联域(TADs)、以及60000多个loops的形成。如预测一样,视网膜中活跃表达的基因在A Component染色质区域,而大多数低活性的基因出现在B Component中,检测全基因组loop环结构特征发现,在A-A互作、A-B互作、B-B互作形成loops三种模式中,A-A互作形成loops距离小于B-B互作和A-B互作形成的loops,并且A-A形成的loops都是活跃的染色质区域,跨度平均小于1Mb,大多数都是发生在TAD内。
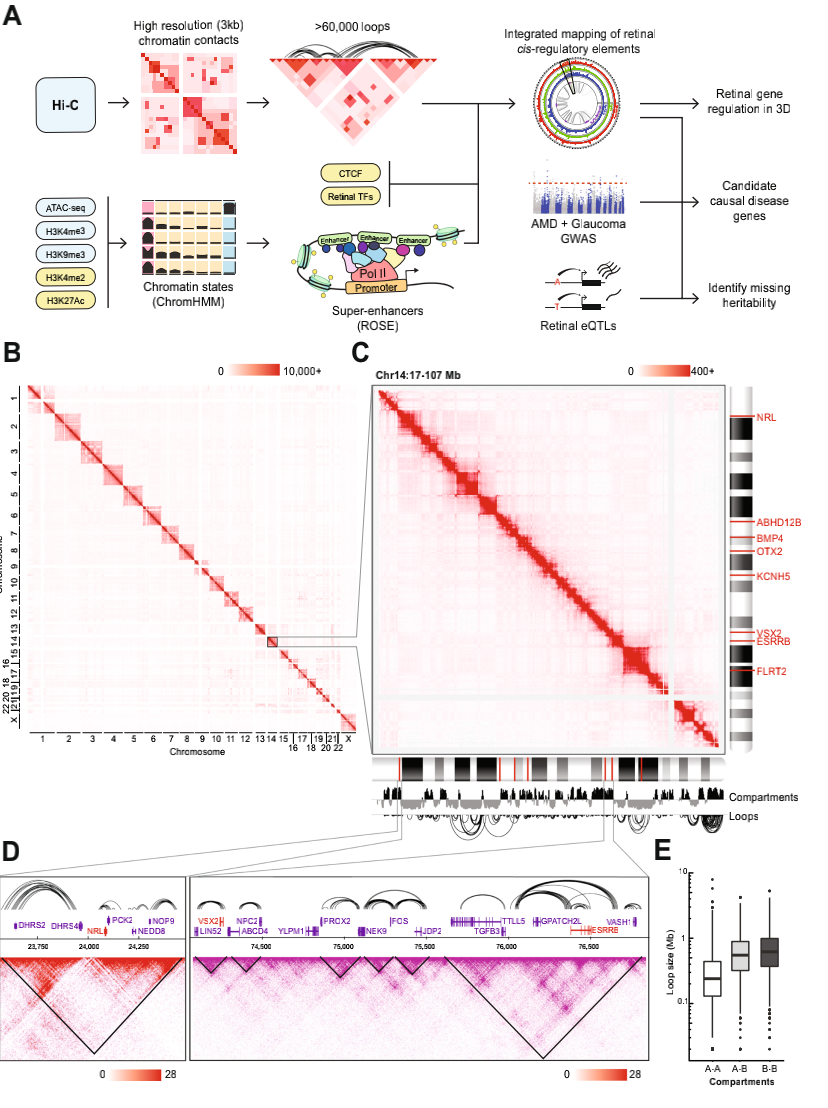
高分辨率Hi-C(5kb)识别人类视网膜染色质结构
2、人视网膜染色质拓扑结构具有组织特异性和物种保守性
为了评估已识别的染色质特征在人视网膜组织特异性和物种保守性,这里比较了人类神经元(ACC)、GM12878淋巴细胞(LCL)、结肠癌细胞(HCT116,clone cancer)Hi-C测序数据,发现视网膜特异性基因,如OTX2、EYS、PDC,只存在于视网膜的A component;由于视网膜和ACC神经元之间的高度相关性,比较发现PAX6基因座在神经元和视网膜中的相互作用与其在两个组织中的表达相似,而编码OTX2和CRX两个关键的视网膜TF,在视网膜中表现出大量的局部相互作用,但在神经元中没有,表明人视网膜组织拓扑关联域具有组织特异性,并在基因调节中发挥着重要作用。
小鼠和人类染色质组织之间的相似性表明了跨物种的守恒,强调了染色质组织模式与视网膜基因调控的相关性。发现小鼠TAD中21.8%的小鼠基因对也位于相应的人类TAD中,超过三分之一(35.7%)的基因对在小鼠体内通过染色质环相互作用,在人类视网膜中也是如此。
在这两个物种中,EGF和ENPEP之间都观察到loops的形成,这些发现表明,视网膜表达的基因表现出保守的染色质拓扑结构,突显了其对调节视网膜基因的重要性,并可能涉及组织特异性顺式调控元件CREs。
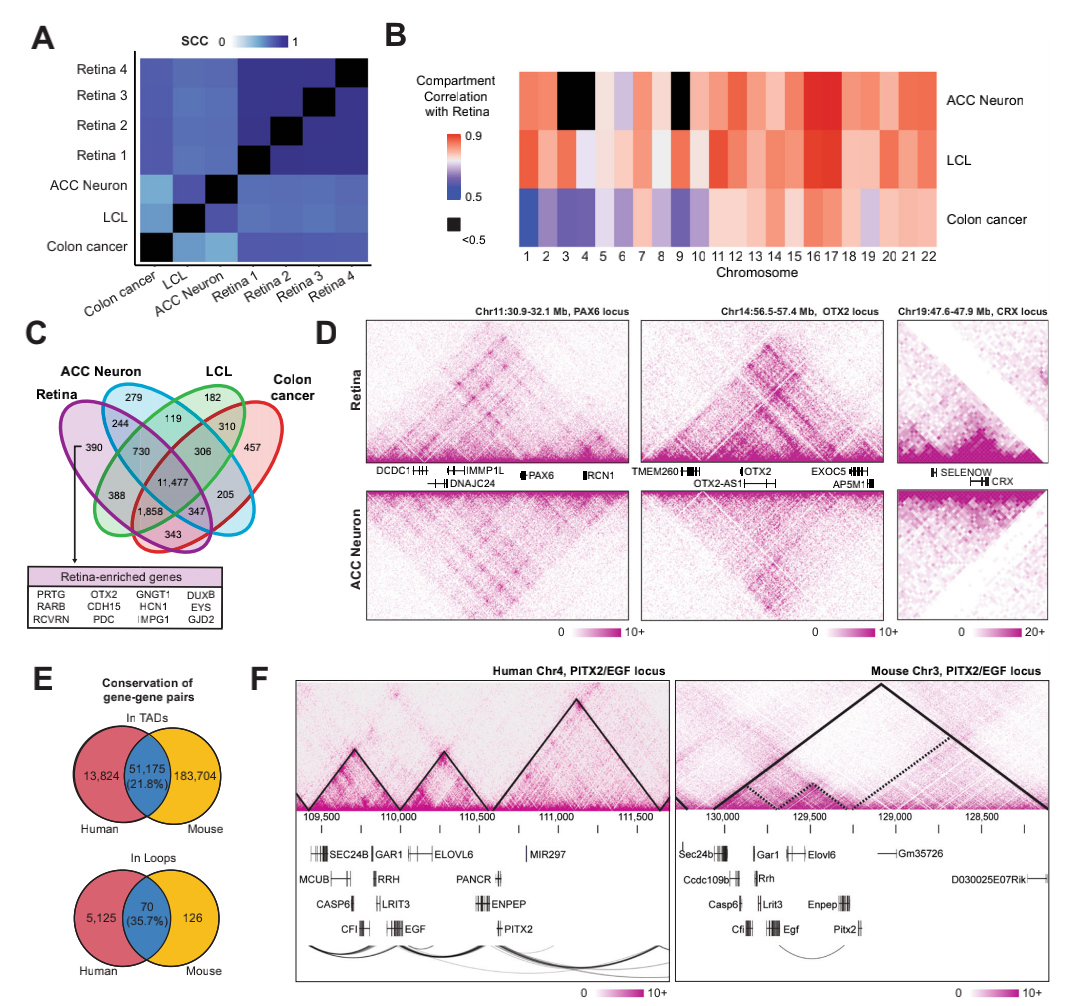
人视网膜染色质相互作用具有组织特异性和物种保守性
3、视网膜中SE与高度表达的组织特异性基因重叠,并富集视网膜TF结合motfis
染色质互作形成的loops促进CRE与其目标基因之间的物理相互作用,并且在控制基因表达方面发挥着至关重要的作用。为了识别视网膜CRE,利用染色质可及性检测(ATAC-seq)确定了染色质开放状态和CUT&Run检测活性(H3K4me3,H3K27Ac,H3K4me234)和抑制性(H3K9me3)组蛋白标记互作的特征序列。通过全基因组染色质开放程度以及开放区域(启动子、增强子、异染色质、低丰度信号)构建了10种染色质状态模型,启动子和增强子区域富集了用于结合CTCF和关键视网膜TF,如CRX、NRL、OTX2、MEF2D、CREB和RORB,另外增强子主要在TSS上游区域富集,而启动子仅在TSS富集。
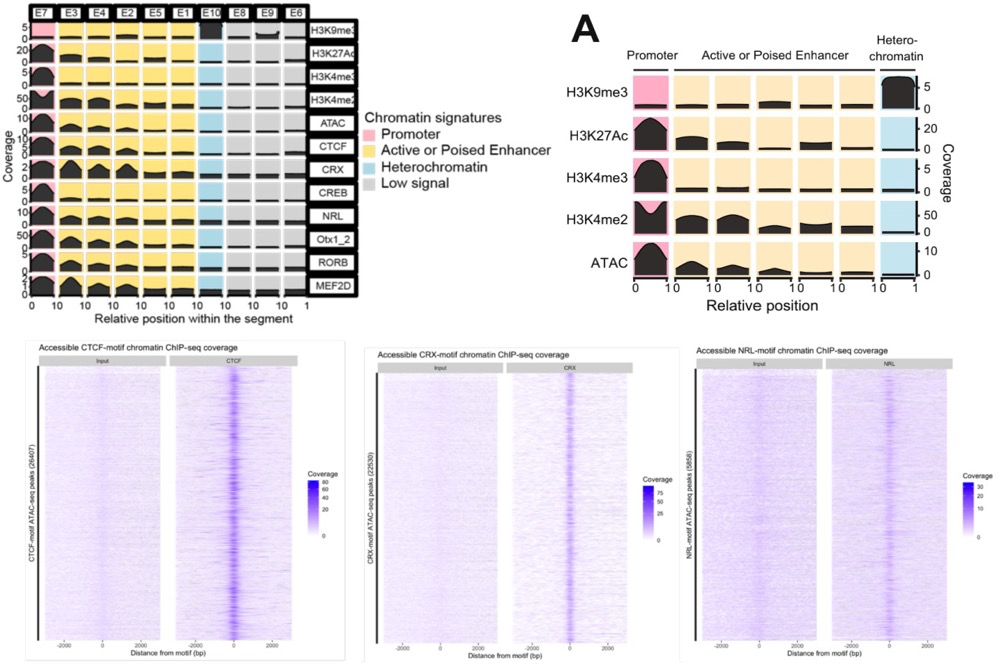
人视网膜全基因组染色质可及性特征
利用H3K27Ac蛋白标记互作在视网膜顺式调控元件启动子和活性或稳定增强子区域鉴定了1325个超级增强子(SEs),发现视网膜表达基因的数量与每个染色体的SE数量之间存在高度相关性。SE与转录组测序数据集结合分析表明,与CRE相比,SE重叠的基因的平均表达量高2.17倍,并且比100组随机SE大小的位点高2.89倍,视网膜中关键的光感受器特异性基因NRL主要在活性组蛋白标记和染色质开放区域的大型SE中富集,与CRE或随机区域相比,TF结合位点主要存在于SE中,其中编码许多具有丰富结合基序TF的基因也与SE重叠,这表明它们在维持视网膜稳态方面的潜在作用。
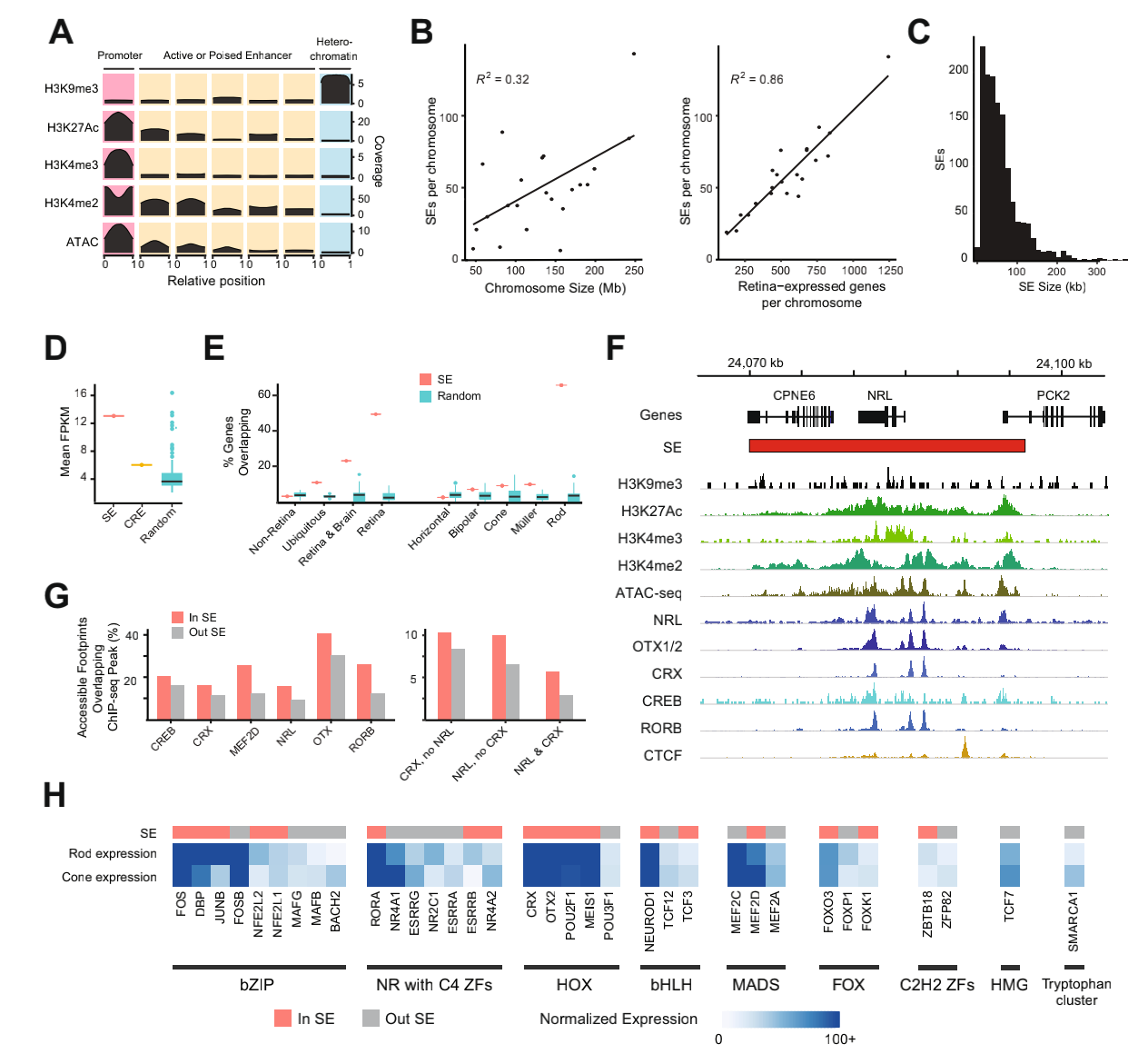
人视网膜超级增强子SE的识别和特征鉴定
4、SE在TAD内部loops富集并参与基因表达调控
以往研究表明SE可能会与其他调节区域互作,形成能够调节多个基因的大型转录单元。视网膜SE在距离较小的loops中富含,并广泛存在与loops内的互作,几乎所有SE参与内部互作的loops都小于1Mb,并且大多数SE主要在TAD中存在交互。
与随机区域相比较,SE在调节相互作用中大量富集,与3,059个独特的CRE、1个独特的TSS和217个独特的SE互作。在视网膜和/或大脑中特异性表达的基因大多与SE重叠,而与SE相互作用但不重叠的基因则是一些常规基因,也有一些SE与许多CRE相互作用,因此可能在协调基因表达模式方面发挥重要作用,这些结果均表明人视网膜中SE的组织特异性和调控独特性。
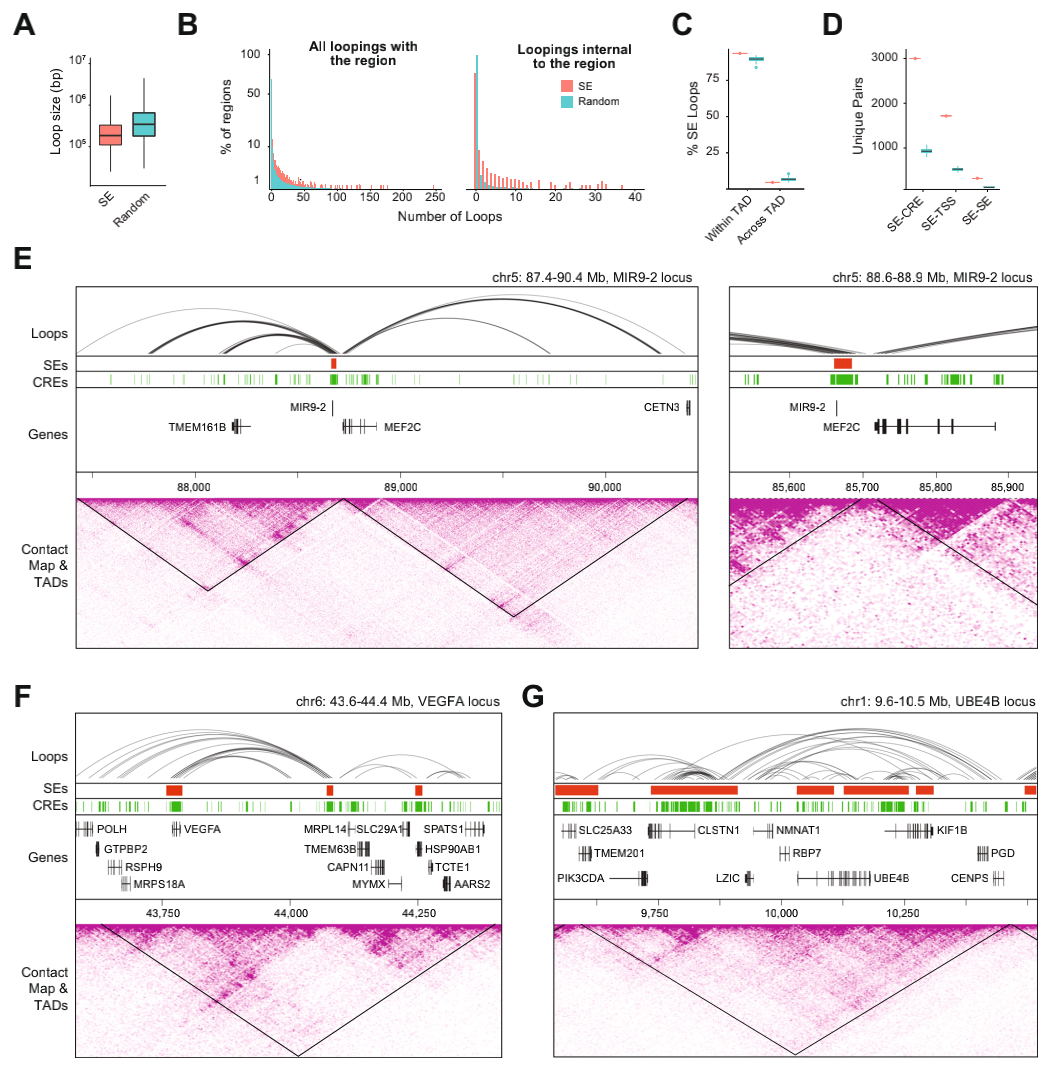
人视网膜SE参与染色质loops形成和互作独特特征
5、SE的TAD内定位与靶基因的边界绝缘和生物学功能有关
进一步研究发现,大多数SE与杆状细胞基因有关,反映了人类视网膜样本中杆状细胞的优势。研究还证明了SE靶基因的生物学功能可能与TAD中的SE定位以及TAD边界绝缘有关。在TADs的边缘,SEs的存在是与较弱的绝缘有关,与其他类型TAD相比,我们发现含SE的TAD中有更多的染色质相互作用。值得注意的是,SE本身大多与位点的相互作用非常接近,表明在局部、TAD边缘有很强的绝缘性,可能是由于CTCF结合的富集和避免附近基因的随机激活,这与之前的研究一致。
SE靶向基因功能研究,即TSS与SE重叠或相互作用的基因,发现SE处在边缘的TAD中都是富含应激反应基因,而视网膜基因主要在SE处于中心的TAD中富集,其中应激反应FOS重叠的SE位于TAD的边缘,该TAD可能会受到超出自身TAD边界的区域变化的影响。
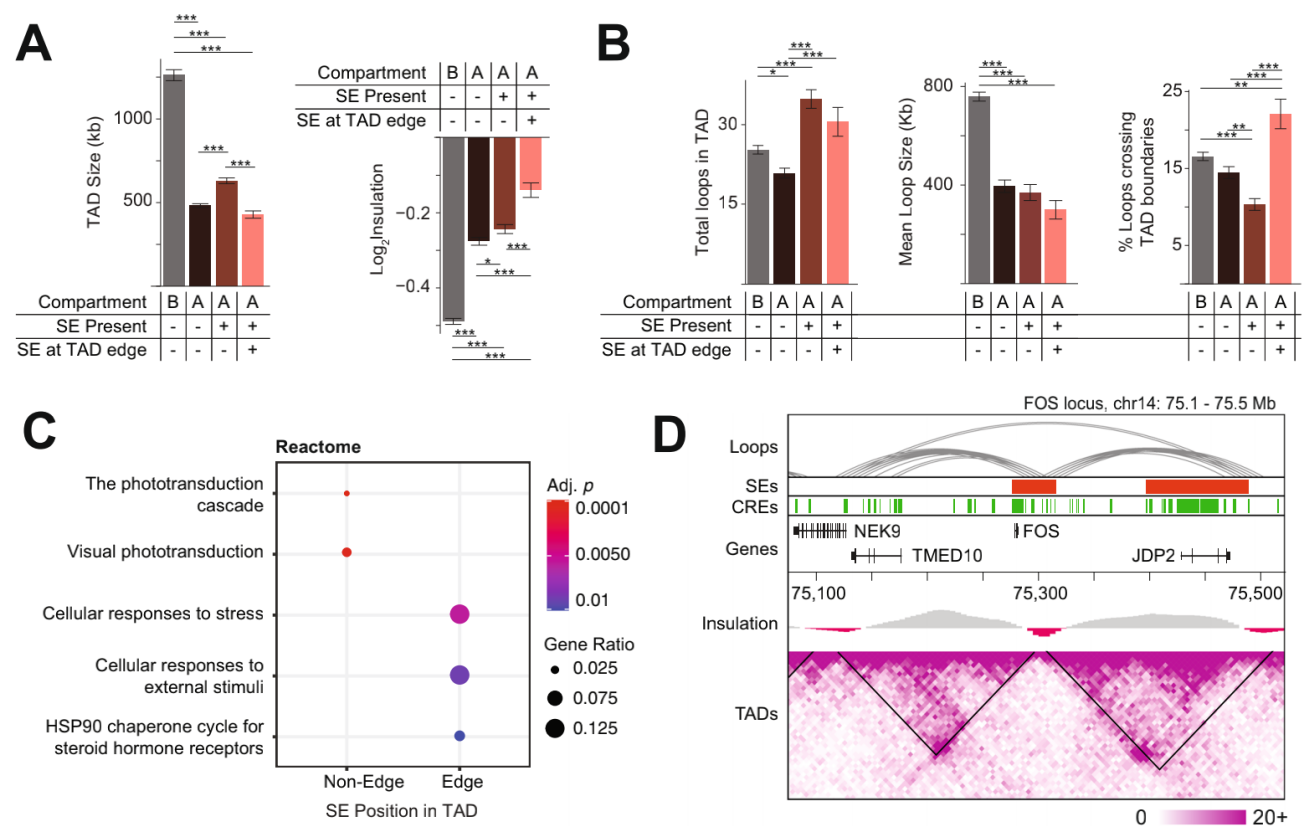
SE的TAD内部定位与TAD的生物特征有关
6、染色质loops、SEs和CREs与视网膜 eQTLs的联合分析
eQTL将特定的遗传变异与目标基因表达的变化联系起来(下文称为eGene),为了识别可能与视网膜基因调节相关的eQTL,结合14,859个视网膜eQTLs,发现大多数视网膜eQTL都存在于活跃的染色质中,并驻留在同一个TAD中,这为解释基因变体对eGene的影响提供了直接机制。此外,评估了变异与其eGene的启动子区域(距离TSS±2.5kb)之间的物理距离,能够区分启动子eQTL、远端eQTL和与启动子相互作用的远端eQTL。分别确定了2,410个CRE(CRE-eQTL)和880个SE(SE-eQTL)特殊的eQTL,其中58.5%的CRE-eQTL和69.3%的SE-eQTL也与相关的eGene相交。总体而言,大多数eQTL在物理上与eGene或其启动子重叠或者通过染色质loops互作接近各自的eGene。
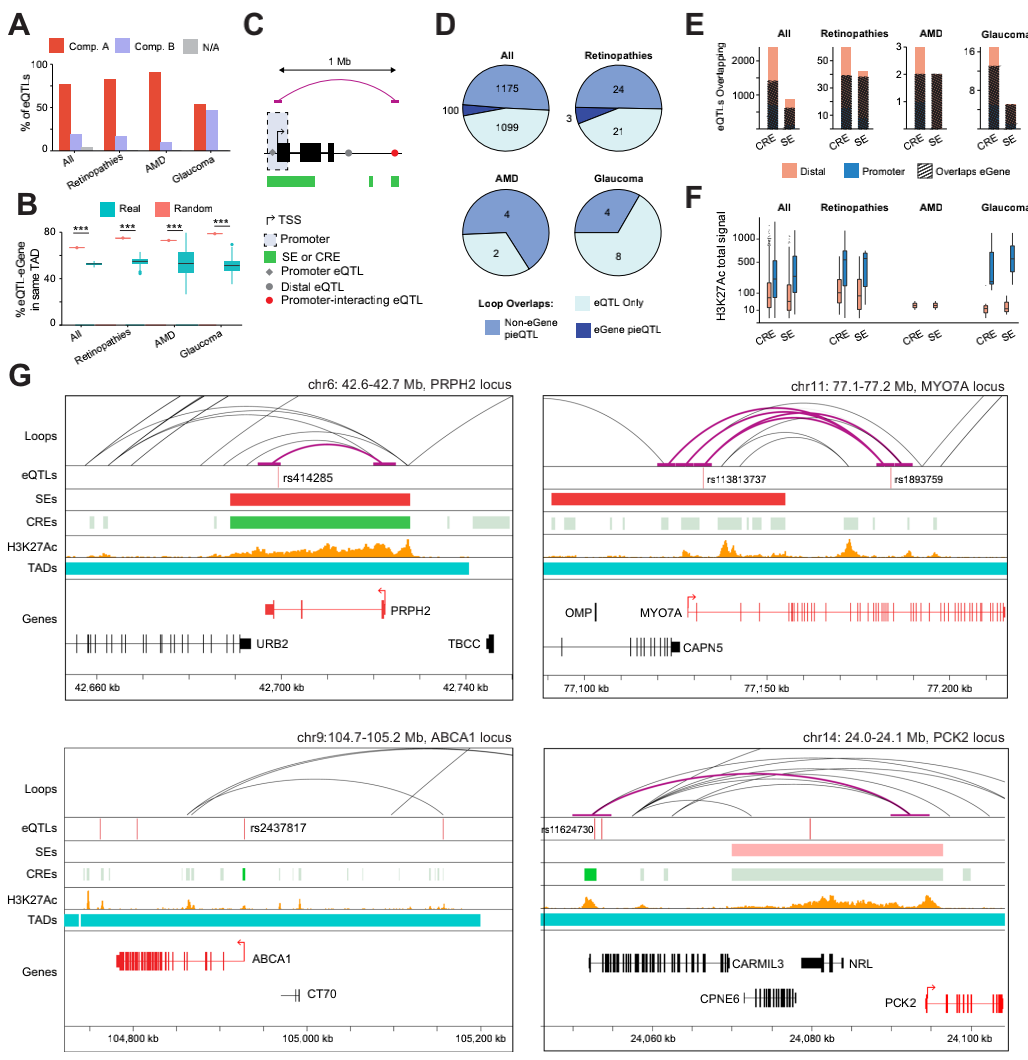
视网膜eQTL和与眼病相关的eQTL的表观遗传变异
7、基因组拓扑结构将目标基因与AMD和青光眼相关的风险变异进行关联
关于青光眼和AMD的GWAS研究已经确定了大量的非编码变异,包括eQTLs分析在内的其他研究提供了进一步见解,但许多相关位点的目标基因和变异仍然难以捉摸。研究人员将染色质互作与从GWAS研究中识别的遗传变异数据进行了整合,并对带有GWAS先导和LD变体的成人视网膜基因组拓扑结构进行了综合分析,发现了几个先前未知的可能与青光眼和AMD有关的基因。例如,研究展示了一个显著的远程相互作用导致目标基因变化的青光眼相关基因TFAP2B/PKHD1。此外,小鼠中TFAP2B表达的中断导致与青光眼一致的强烈病理表型,这些发现指向了与这些疾病相关的特定候选致病基因。研究团队发现了AMD的候选致病基因,在CTRB2/CTRB1位点建立了CFDP1作为AMD相关的候选基因。
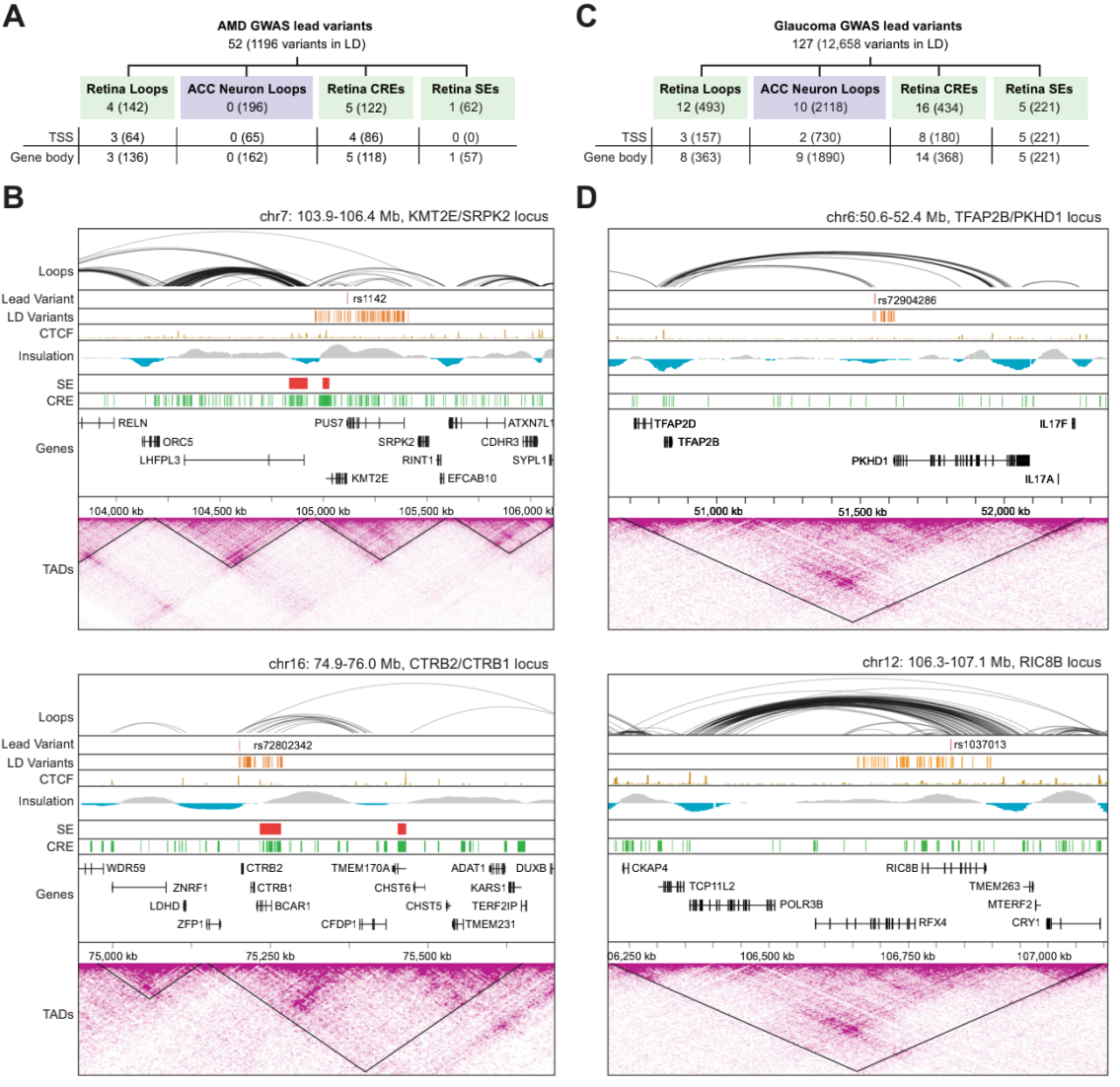
AMD和青光眼变异基因通过视网膜染色质loops与靶基因作用
总结
研究团队通过将高分辨率Hi-C数据与表观遗传图谱和顺式调控元件整合,为人类视网膜研究提供了一个重要的数据资源,促进了基因组调控的研究。研究团队识别了视网膜病变中缺失的遗传性,以及包括AMD和青光眼在内的常见致盲疾病的候选因果基因和变异,该研究为将调控变异与视网膜疾病表型联系起来提供了一个框架。整合的基因组调控图谱还将有助于评估与其他常见视网膜相关疾病(如糖尿病视网膜病变)相关的基因,确定缺失的遗传性,并了解遗传性视网膜和黄斑疾病的基因型-表型相关性。
这项研究的首席研究员Anand Swaroop教授说:“这是视网膜调控基因组拓扑与老年性黄斑变性(AMD)和青光眼相关的遗传变异的首次详细整合,这两种基因变异是视力丧失和失明的两个主要原因。”Swaroop说:“拥有如此高分辨率的基因组结构图像,将继续为组织特异性功能的基因控制提供见解。”
如果您对文中测序技术感兴趣,欢迎点击下方按钮联系我们,我们将免费为您设计文章思路方案
参考文献
Marchal C, Singh N, Batz Z, et al. High-resolution genome topology of human retina uncovers super enhancer promoter interactions at tissue-specific and multifactorial disease loci. Nat Commun. 2022 Oct 7;13(1):5827.









 京公网安备 11011302003368号
京公网安备 11011302003368号