b) GEO Sample (GSM) 样本ID号
c) GEO Series (GSE) study的ID号
d) GEO Dataset (GDS) 数据集的ID号这些数据均可以在ftp(ftp://ftp-trace.ncbi.nih.gov/geo/)进行下载。
 一般我们在文章中看到的都是GSE的ID,那我们如何通过GSE的ID进行数据的下载呢,下面就让小编手把手教您如何进行GEO数据的下载。
一般我们在文章中看到的都是GSE的ID,那我们如何通过GSE的ID进行数据的下载呢,下面就让小编手把手教您如何进行GEO数据的下载。
咱们以下面篇文章为例:
 我们在文末找到作者数据上传地址和GSE的ID
我们在文末找到作者数据上传地址和GSE的ID
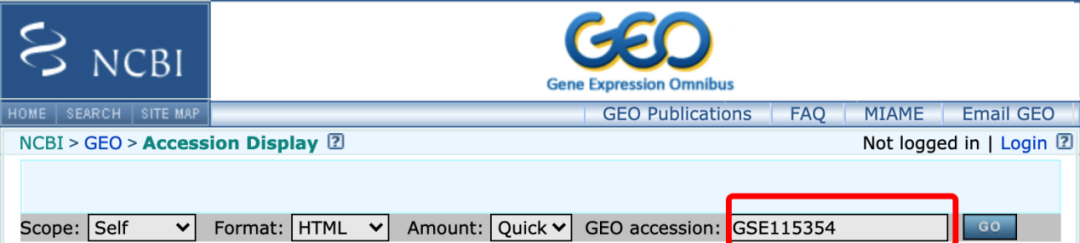
 然后在GEO官网输入GSE115354,
然后在GEO官网输入GSE115354,
首先我们可以看到是关于该study的描述信息,包括文章信息、测序物种、实验类型等等

而我们最关心的东西在页面的下方
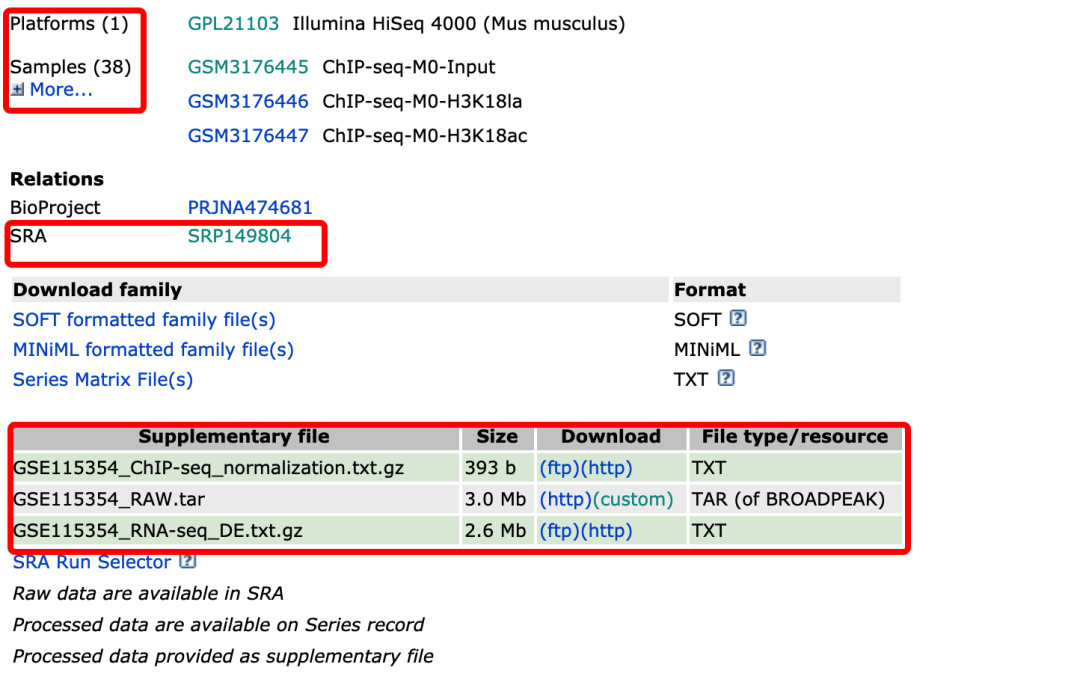
如果我们想下载作者标准化后的数据,可以直接在这个页面中Supplementary file中进行下载,那如果我想下载原始数据怎么办呢,不要着急,您慢慢往下看。
我们点击页面中Sample对应的GSM的ID,每个样本都对一个GSM,我们以第一个为例,点击后进入以下界面
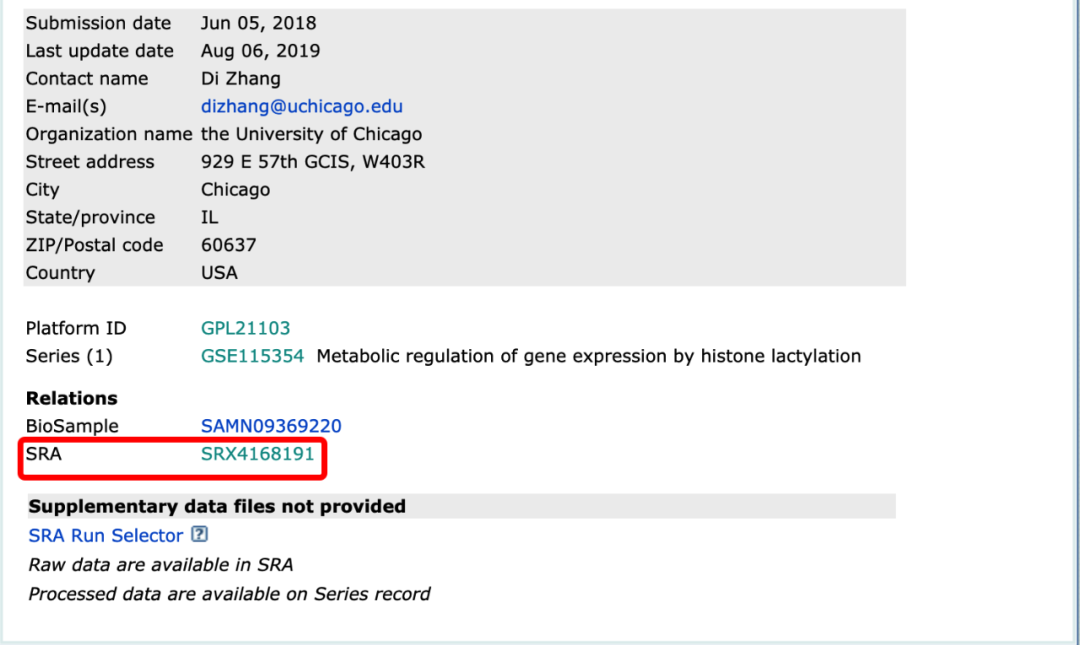 然后点击最下方SRA编号,进入下面的页面
然后点击最下方SRA编号,进入下面的页面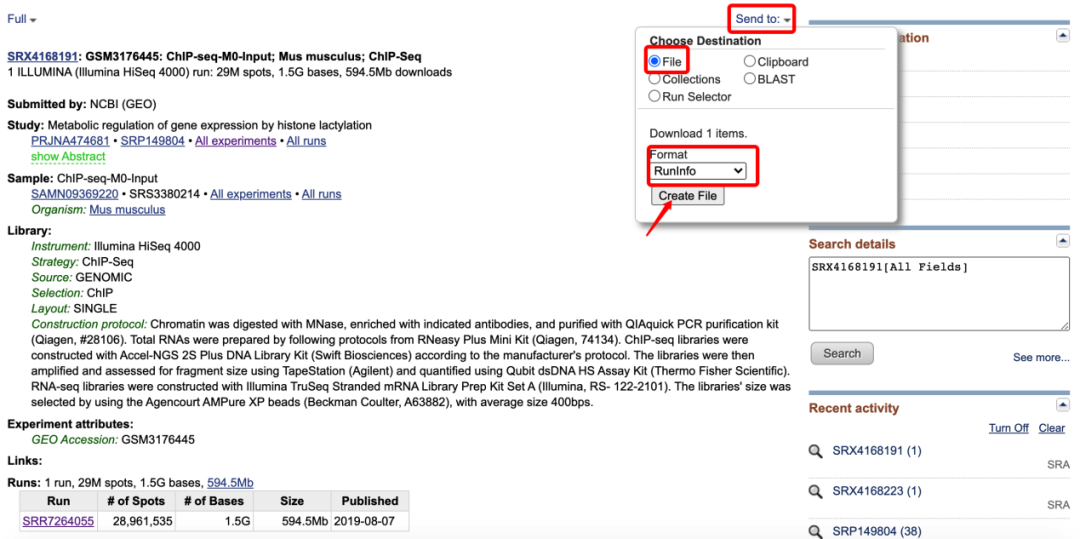 这里面包含了这个样本的基本信息,包括测序平台、文库类型、数据量等基本信息,然后点击页面右上方的Send to按钮,选择File,Format选择RunInfo,然后点击Creat file下载一个csv文件,打开文件,可以看到一个下载链接
这里面包含了这个样本的基本信息,包括测序平台、文库类型、数据量等基本信息,然后点击页面右上方的Send to按钮,选择File,Format选择RunInfo,然后点击Creat file下载一个csv文件,打开文件,可以看到一个下载链接

点击链接就可以直接下载数据了。
下载完成后您可能有些疑问,我们一般测序数据都输pair-end的双端reads,为什么我下载的是一个.sra结尾的数据呢?难道是一个单端数据吗?这个当然不是,SRA为了节省空间,一般上传的数据都是.sra的压缩文件,那我们如何将sra文件转化成常用的双端数据呢?这里就要用到SRA提供的一个工具:fastq-dump,
下载网址:https://trace.ncbi.nlm.nih.gov/Traces/sra/sra.cgi?view=software
官方说明文档:https://trace.ncbi.nlm.nih.gov/Traces/sra/sra.cgi?view=toolkit_doc&f=fastq-dump
当然,这个软件需要在Linux系统下进行操作,命令行如下:
fastq-dump *.sra --split-3
这里的–split-3会把原来双端拆分成两个文件,但是原来单端并不会保存成两个文件。运行完成后就能获得了您想要的原始数据了。如果您还想学习更多实用的高通量测序数据挖掘和分析的知识,欢迎点击下方按钮联系我们。








 京公网安备 11011302003368号
京公网安备 11011302003368号