技术背景
DNA甲基化是一种在原核和真核生物基因组中常见的复制后表观遗传修饰,可以在不改变 DNA分子一级结构的情况下调节基因组的功能,其在基因表达、转座子沉默、染色质相互作用、细胞分化以及生长发育过程中起着重要作用。
其形成过程主要是在DNA甲基转移酶的作用下,将S-腺苷甲硫氨酸分子上的甲基(CH3-)转移到DNA的CpG两个核苷酸的胞嘧啶上进行DNA 甲基化修饰,形成 5-甲基胞嘧啶 (5-mC)、N6-甲基腺嘌呤 (N6-mA) 以及7-甲基鸟嘌呤(7-mG)。
DNA 甲基化造成基因沉默的原因大多都是由于转录受到抑制从而影响了目的基因的表达:(1)启动子区域发生甲基化 ;(2)基因序列中某位点发生甲基化抑制蛋白的表达 ;(3)染色体结构由于受到 DNA 序列的变化从而变得紧密。
因此,阐明DNA甲基化的动力学和遗传学及其分子调控机制,可在基因表达层面上探究动植物的生长发育及抗逆性机理,为优良新品种的培育和遗传改良研究提供更多理论依据。
目前应用广泛的全基因组甲基化检测方式主要是BS-seq:用亚硫酸氢盐处理DNA将胞嘧啶残基(C)转化为尿嘧啶(U),而5-甲基胞嘧啶残基(5mC)不会发生转变。因此可通过测序的手段识别甲基化位点。但是亚硫酸氢盐处理后DNA会发生降解,测序长度受限,使得较大的区域难易被解析。
三代测序平台则可以在不依赖DNA实验处理的情况下,根据信号差来识别甲基化位点信息。其中,PacBio 已经实现通过HiFi测序直接检测 DNA 样本中 CpG 位点的 5mC甲基化。基于HiFi 的 5mC 检测能够在提供既长又准的测序结果的同时,给出准确的单倍型甲基化信息。同时,具有甲基化信息的HiFi数据,所占存储仅增~5%,极大的压缩了可分析的空间,为表观遗传挖掘保驾护航!
技术原理
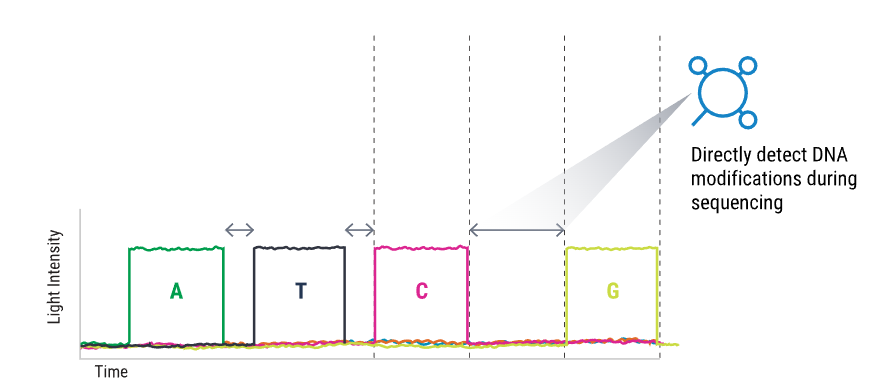
PacBio平台主要通过DNA聚合酶与模板链结合,在链合成的过程中,不同荧光标记的碱基会释放不同的荧光信号,根据光的波长与峰值从而判断碱基类型与顺序。聚合酶合成中每个碱基间都存在时间间隔,当模板碱基有修饰时,两个相邻的脉冲峰之间的距离就会延长。当IPD比例(两个相邻的脉冲峰之间的距离和参考序列距离的比值)明显大于1 时,可推断这个位置存在修饰。
技术优势
- 1)操作便捷:无需亚硫酸氢盐处理,有效避免降解等弊端。
- 2)准确性强:使用HiFi测序进行甲基化检测与亚硫酸氢盐测序结构具有一致性。
- 3)更大范围:与短读长测序相比,可检测到基因组中例如着丝粒等高重复的复杂区域。
- 4)功能强大:可识别等位基因特异性甲基化
- 5)用途广泛:一份数据可同时用于分析SNP、SV、甲基化、基因组组装。
分析方案
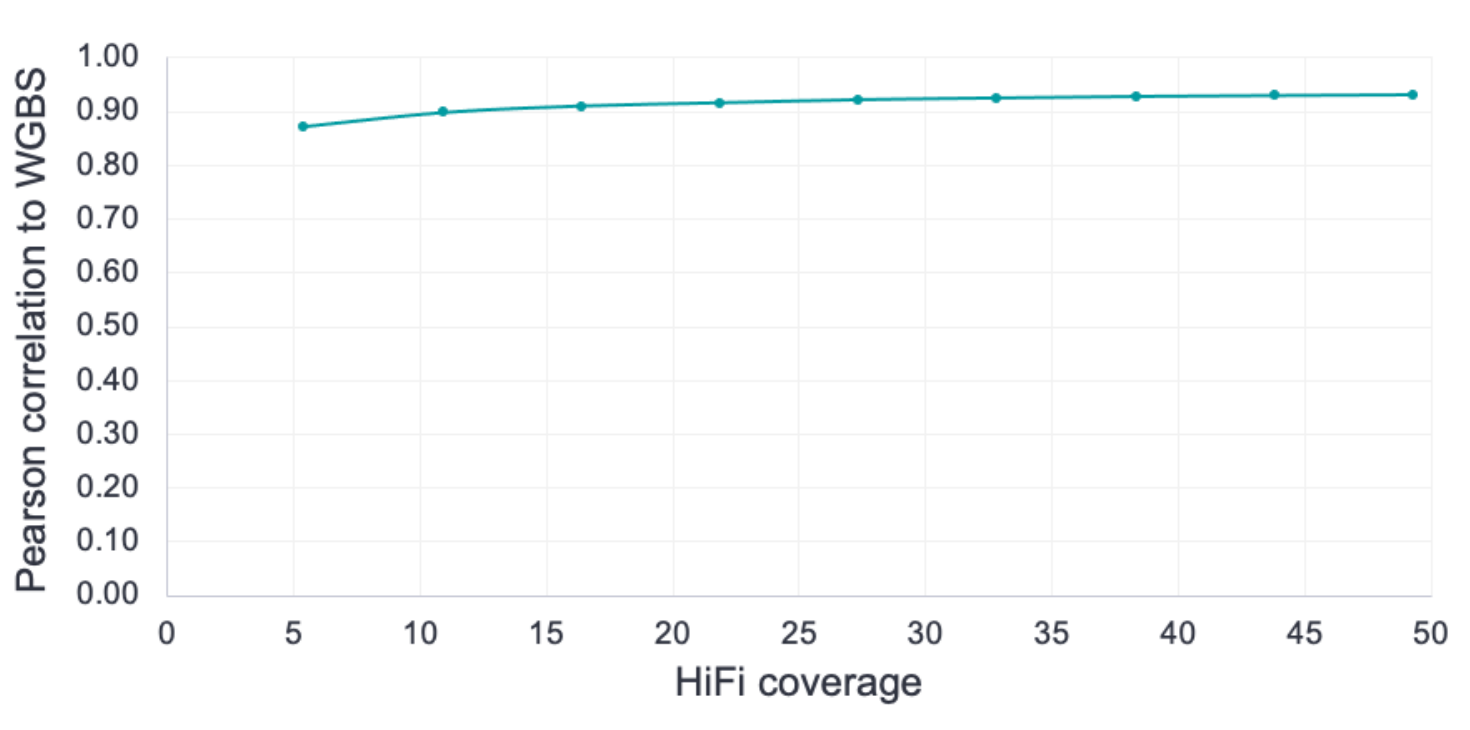
测序深度;≥10x。根据数据检测图可以看到,当深度达到10X,即可达到很好的检出效果。
应用场景
(1)全基因组甲基化
对单个样本进行HiFi测序可检测整个基因组的甲基化模式,如转录起始位点的低甲基化。不同组织部位甲基化差异,构建全基因组的甲基化图谱。
(2)泛基因组甲基化
通过对多个样本进行测序来确定物种品系间的差异甲基化,从而揭示品系间功能差异的调控机制。
 本研究构建了青鳉(Oryzias latipes)近亲繁殖系中12尾材料的基因组,并通过比对构建了青鳉的图型泛基因组。结果显示泛基因组总长度为1.3 Gb,大约是其参考基因组的1.8倍。通过泛基因组展示了更大、更复杂的结构变异-“暗变异”,为进一步研究结构变异(SV)如何影响感兴趣的表型提供了重要的研究基础。作者通过构建基因组时的三代测序数据,进一步对12尾近亲繁殖系的大脑样本中的CpG甲基化进行了差异研究。结果显示,甲基化模式在多代中表现一致,说明甲基化是青鳉鱼的一种遗传性状,能够持续多代,并可能影响表型性状。最后结合RNA等数据,表明galm基因甲基化可能性与肝脏表达之间存在显著相关性。
本研究构建了青鳉(Oryzias latipes)近亲繁殖系中12尾材料的基因组,并通过比对构建了青鳉的图型泛基因组。结果显示泛基因组总长度为1.3 Gb,大约是其参考基因组的1.8倍。通过泛基因组展示了更大、更复杂的结构变异-“暗变异”,为进一步研究结构变异(SV)如何影响感兴趣的表型提供了重要的研究基础。作者通过构建基因组时的三代测序数据,进一步对12尾近亲繁殖系的大脑样本中的CpG甲基化进行了差异研究。结果显示,甲基化模式在多代中表现一致,说明甲基化是青鳉鱼的一种遗传性状,能够持续多代,并可能影响表型性状。最后结合RNA等数据,表明galm基因甲基化可能性与肝脏表达之间存在显著相关性。
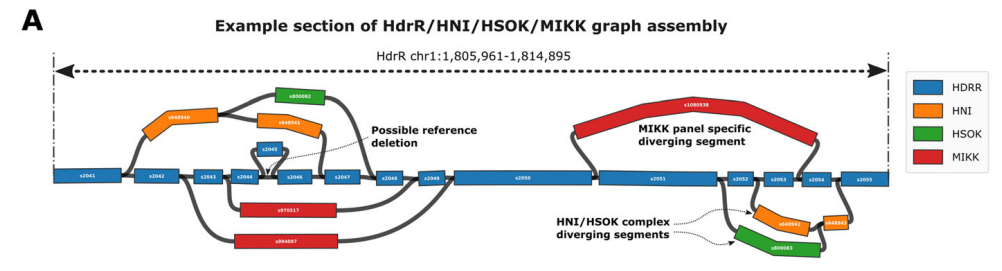
泛基因组结构特征与12尾青鳉甲基化差异特征
(3)单体型甲基化
HiFi测序能够同时分阶段读取母系和父系单倍型,并检测甲基化。这直接揭示了印记的模式,其中染色体的甲基化状态取决于它是从母亲还是父亲遗传的。
日本田中鱼(Oryzias latipes)作为脊椎动物模式生物有着悠久的历史。我们利用其异常高的近亲繁殖耐受性建立了Medaka近亲繁殖Kiyosu Karlsruhe(MIKK)小组:来自单个野生种群的近等基因脊椎动物系的集合[3]。
在与这篇文章一起发表的配套文章中,我们根据Illumina短读序列与最近、完全组装的参考基因组(日本南部medaka自交系HdrR)的比对,提供了80个个体MIKK面板系[4]的详细遗传特征。
尽管这使我们能够发现与HdrR相关的MIKK面板中的许多遗传变异,但该方法不可避免地隐藏了某些变异,包括更大、更复杂的结构变异-“暗变异”-这可能会对每个品系产生功能性后果。在这里,我们描述了我们如何使用牛津纳米孔技术(ONT)长读测序来揭示12个MIKK面板系中的一些暗变异,从而使我们对其基因组变异进行更完整的评估,并为进一步研究结构变异(SV)如何影响感兴趣的表型铺平了道路。
DNA甲基化分析:
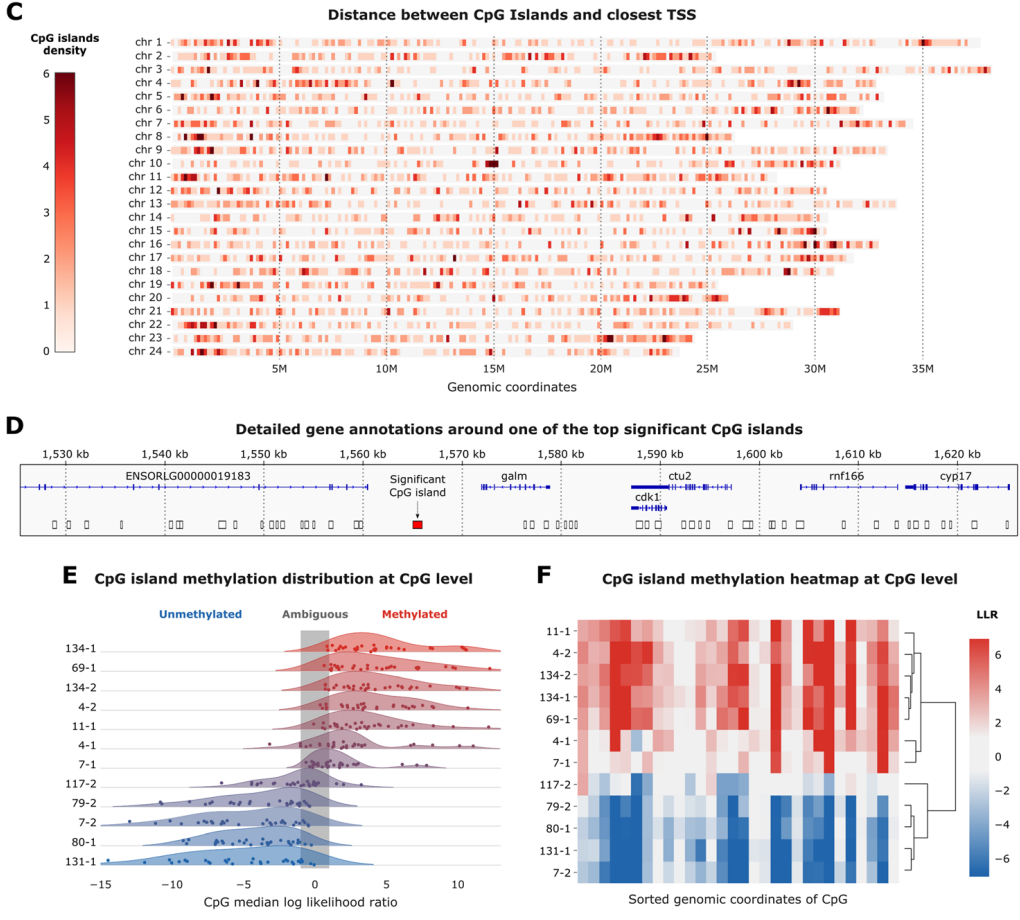
- MIKK面板样本中所有显著CpG岛差异甲基化的热图。
- CpG岛按基因组位置和X轴排序。样本通过Y轴上的分层聚类排序,并进行颜色编码,以便用相同的颜色表示兄弟线。
- 颜色方案根据来自的中值对数似然比的值−3(蓝色)到3(红色)之间的值不明确−1和1为白色。
- B红色显著差异甲基化CpG(n=4249)和蓝色所有非显著岛(n=8727)的最接近基因TSS的距离分布。
- C 24条HdrR染色体基因组窗口中显著CpG岛的数量,从0(白色)到6(深红色)。
- D示例100kb区域包含红色的显著CpG岛(chr15:1565040-1565987),而非白色的非显著CpG岛。
- E面板D中突出显示的CpG岛的CpG水平对数似然比核密度图。通过降低中值llr,在Y轴上对样本进行排序。单个CpG值由点表示。
- 图D中突出显示的CpG岛的对数似然比热图,按样本分层聚类。X轴上是按基因组位置排序的单个CpG
如果您对我们提供的服务感兴趣,欢迎联系我们。







 京公网安备 11011302003368号
京公网安备 11011302003368号