
发表期刊:Front. Cell Dev. Biol.
影响因子:6.684
发表时间:2021.09
发表单位:甘肃省人民医院
全文链接:https://international.biocloud.net/zh/article/detail/33681226
研究背景
肺腺癌(LUAD)是一种常见的高死亡率肺癌,microRNAs(miRNAs)在其调节中起着重要作用。mRNAs可能受miRNAs调控,参与LUAD肿瘤的发生和发展。然而,参与LUAD的miRNA-mRNA调控网络尚未完全阐明。本研究研究了与LUAD相关的miRNA-mRNA调控网络,探索了关键miRNAs和mRNA在该网络中的潜在功能。这些发现有助于识别新的进展临床环境中LUAD患者的诊断诊断标志物和治疗靶点。
研究材料
TCGA数据集:LUAD和正常肺组织的miRNA数据以及mRNA数据
测序数据:LUAD患者(n=6)和对照组(n=4)的血浆样本分别构建sRNA文库,使用Illumina测序仪进行测序。(由北京百迈客生物科技有限公司提供测序及技术支持)
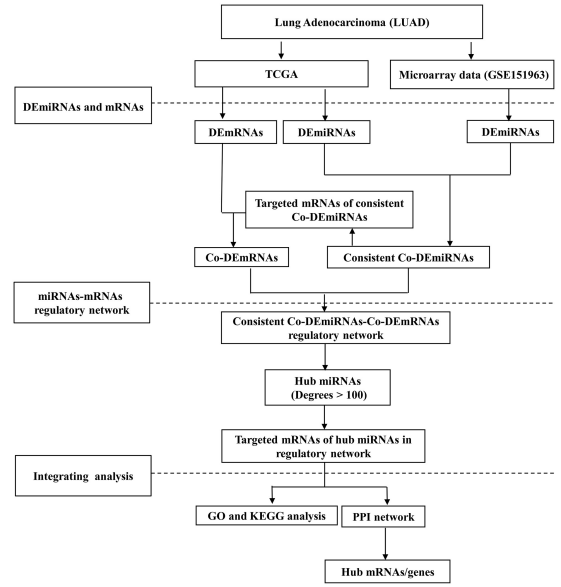
研究流程
分析方法
通过TCGA数据库和测序数据分别得到差异表达的miRNAs和mRNA。然后,通过两个数据集之间的交叉分析,获得共同差异表达miRNA。再通过靶向的mRNA和TCGA中的差异mRNA的重叠确定共有差异mRNA。利用cytoscape构建了miRNA-mRNA调控网络。前5个miRNA在网络中被确定为枢纽miRNA。通过GO分析和KEGG通路分析,揭示了与枢纽miRNA靶向基因相关的功能和信号通路。利用STRING数据库和CytoHubba对蛋白相互作用(PPI)网络中的关键mRNA进行了鉴定。使用基因表达谱交互分析(GEPIA)进行生存分析。
主要结果
通过小RNA数据的标准分析,作者共得到了3166个miRNA,用于后续的数据分析。从测序数据中LUAD和对照组的血浆中鉴定出319个差异miRNAs(145个上调和174个下调)。此外,利用TCGA鉴定出4206个差异mRNAs(1105个上调,3101个下调)和197个差异miRNAs(126个上调,71个下调)。此外,197个差异miRNAs中有147个(95个上调,52个下调)有亚型表达。
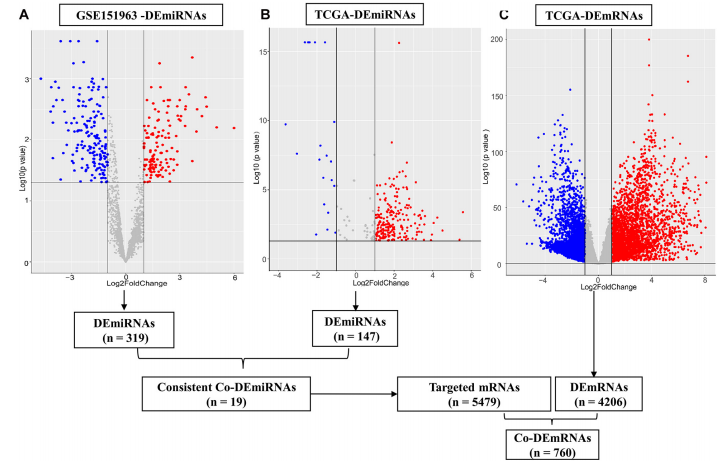
(图2)miRNA和mRNA的差异表达
通过在TCGA和测序数据之间的交叉分析,得到30个共有的差异miRNAs,包括19个上下调一致的差异miRNAs和11个上下调不一致的差异miRNAs。从19个一致的共有差异miRNA中总共鉴定出5479个靶mRNA。接下来,通过对TCGA和测序数据中的差异mRNAs进行交叉分析,鉴定出760个共有差异mRNAs(152个上调,608个下调)。miRNA-mRNA调控网络由19个共有差异miRNAs和760个共有差异mRNAs组成(图3)。总的来说,19个一致的共有差异miRNAs与760个共有差异mRNAs确定了985对靶向关系。此外,关联度最高的前5个miRNAs被确定为网络中的枢纽miRNAs,包括miR-539-5p、miR-656-3p、let-7b-5p、miR-2110和miR-92b-3p。最后,677个共有差异mRNAs由来自5个枢纽miRNAs的靶向mRNA组成。
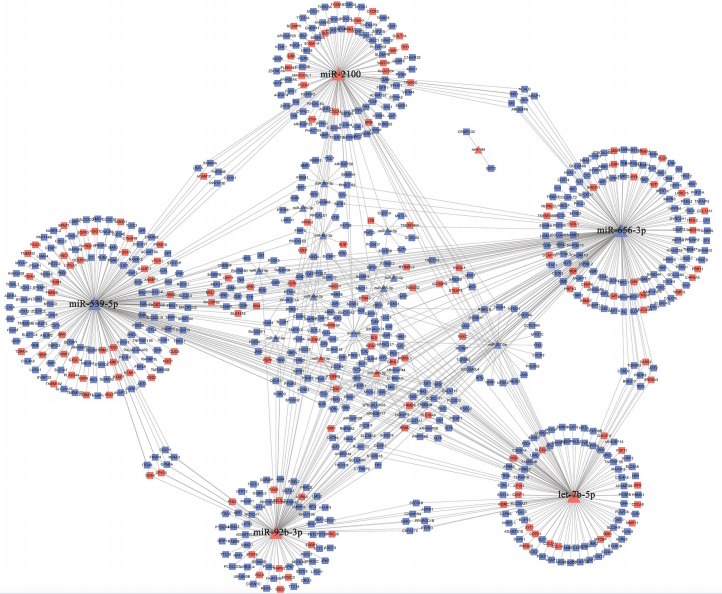
(图3)miRNA-mRNA调控网络
三、关键miRNAs靶向mRNA的GO和KEGG分析
作者使用了677个共有差异mRNAs,通过GO和KEGG通路富集分析来鉴定关键miRNAs的潜在功能。作者在GO分析中获得了836个结果,其中图4A显示了来自生物过程、细胞成分和分子功能的前10个结果。一些结果与肿瘤的发生和进展有关,包括调节细胞对生长因子刺激的反应,含胶原蛋白的细胞外基质和基底膜等。此外,从KEGG通路分析中注释了48条信号通路,前10个结果如图4B所示。这些通路大多数与肿瘤的发生和发展相关,包括焦点粘连、癌症中的蛋白多糖、细胞外基质-受体相互作用和Wnt信号通路。其中粘着斑通路富集最为显著。
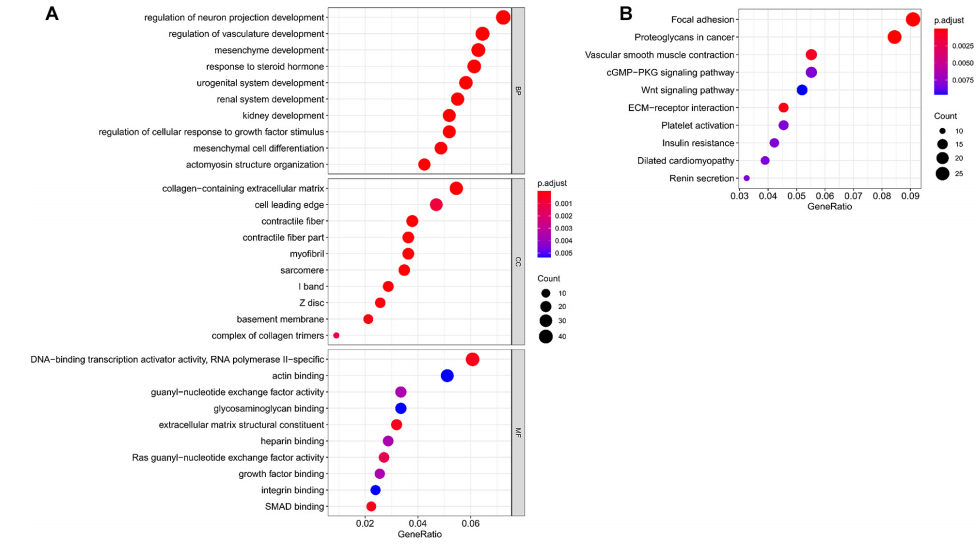
(图4)关键 miRNA靶向mRNA的功能富集分析
四、关键基因的鉴定
PPI网络从677个共有差异mRNA中识别出597个mRNA,其中有2246条边相互关联(图5)。使用cytoHubba插件MCC排名确定了前10个关键mRNA,包括神经源性位点notch同源蛋白1(NOTCH1)、基质金属蛋白酶2(MMP2)、胰岛素样生长因子1(IGF1)、激酶插入结构域受体(KDR)、分泌磷蛋白1(SPP1)、血管内皮生长因子受体1(FLT1)、肝细胞生长因子(HGF)、血管生成素1受体(TEK),血管生成素-1基因(ANGPT1)和血小板源性生长因子(图6)。通过生存分析的计算,作者使用GEPIA进一步验证了这些关键的mRNA。在这里,只有两个(SPP1和HGF)被鉴定为关键基因,并与生存相关(图7)。生存分析显示,在LUAD中,SPP1的表达与生存呈负相关,而HGF与生存呈正相关(图7)。此外,SPP1和HGF均在黏附信号通路中富集。
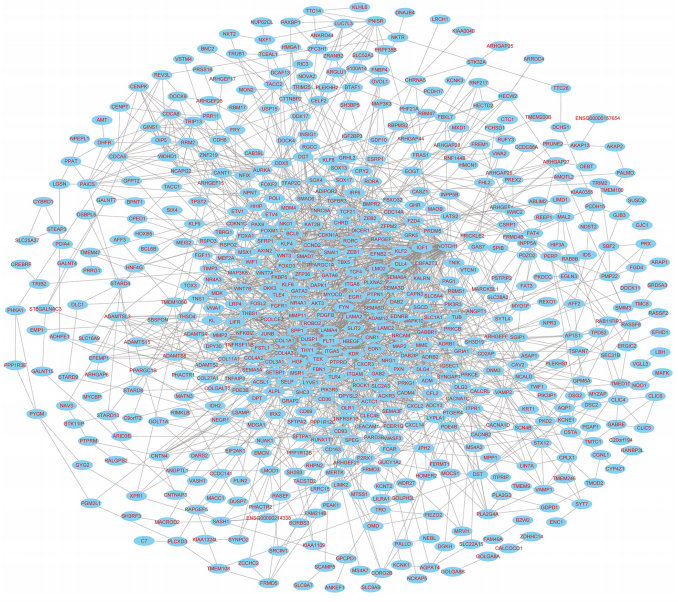
(图5)关键miRNAs靶向mRNA的PPI网络图
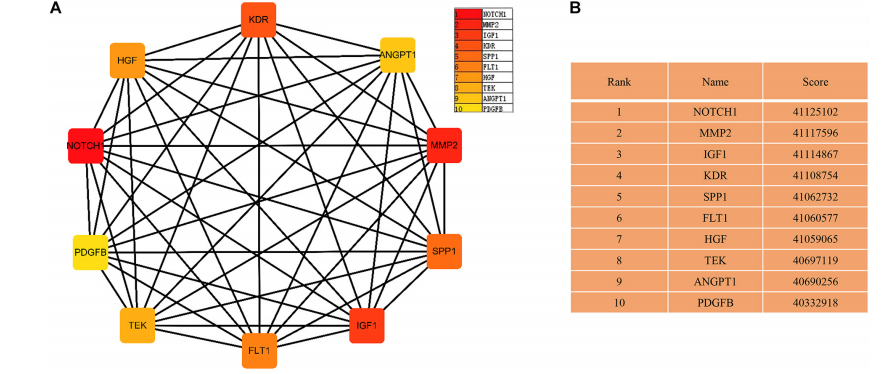
(图6)关键mRNA的网络
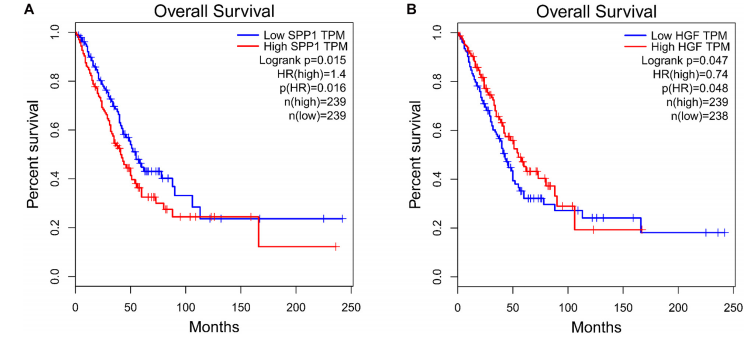
(图7)关键基因的生存分析
总 结
作者利用测序数据和TCGA数据集构建了LUAD相关的miRNA-mRNA调控网络,鉴定了5个关键miRNAs和两个关键mRNA。miR-539-5p,miR-656-3p、let-7b-5p、miR-2110和miR-92b-3p在LUAD肿瘤的发生和进展中发挥重要作用。两个关键mRNA,SPP1和HGF,与LUAD患者的生存相关。这些发现为临床环境中的预测、预后和治疗靶点提供了新的分子标记物,并阐明了LUAD调控的新机制。








 京公网安备 11011302003368号
京公网安备 11011302003368号