当前蛋白质组学已成为21世纪生物医学、植物学、微生物学等研究领域最活跃的热点之一,其理论和方法在植物学科中的应用方兴未艾,已渗透到植物遗传学、植物生理学、植物发育生物学、植物生态学、植物共生生物学和植物病理学等诸多领域,为农学、林学、畜牧学和水产学等相关农业生物科学领域的研究注入了新的活力。在农业生产领域,蛋白质组学已经在作物遗传育种,作物逆境胁迫应答,作物品质改良和农作物-微生物相互作用等领域的研究中得到了广泛应用。
蛋白质组学在植物研究中的应用案例一
中文标题:比较非标记label free定量蛋白质组学揭示了N-连接糖链通过调节蛋白丰度对拟南芥耐盐性起到的关键作用
研究对象:拟南芥幼苗
发表期刊:Frontiers in Plant Science
运用技术:非标记label free定量蛋白质组学
研究背景
作为固着生物,陆地植物经常遭受各种压力和不利环境,例如极端温度、干旱和高盐度。在农业生产中,高盐分影响自然界作物的地理分布,造成大量的产量损失。许多研究表明,多种基因和/或代谢途径在植物的非生物胁迫反应中发挥协同作用,很难通过调节单一有效基因或蛋白质来显着提高植物对外界胁迫的抵抗力。
在植物中,N-糖基化参与许多生物过程,包括类固醇激素受体的 ER 质量控制、配子体识别, 亚细胞转运, 植物先天免疫 和气孔发育等,主要是通过调节底物蛋白的稳定性、功能或亚定位实现调控过程。此外研究发现复杂的N-聚糖生物合成失败会导致拟南芥幼苗对盐敏感,但对其潜在的分子机制仍知之甚少。本文利用纳升流液相色谱串联质谱 (nanoLC-MS/MS) 技术进行了无标记蛋白质组学分析,比较分析了野生型 (WT) 和两个抗盐胁迫的突变体的蛋白质组差异。
研究流程
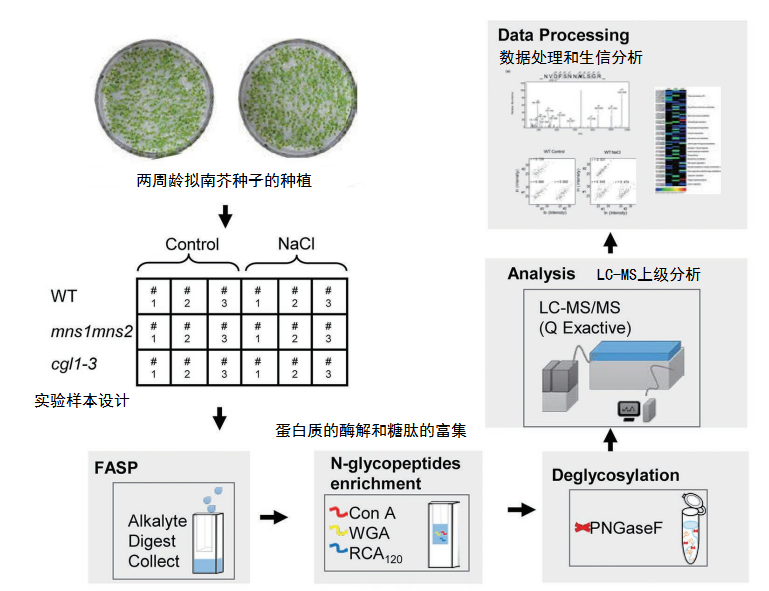
研究结果
从632个多肽中共鉴定出727个糖基化位点,对应于371个糖蛋白。大多数 N-糖基化位点表现出典型的NXS/T基序序列。鉴定出的 N-糖蛋白的数量因样品而异。在对照组中,在mns1 mns2中鉴定出311个糖蛋白,在cgl1-3突变体中鉴定出300个糖蛋白,而在WT中仅鉴定出203个。在WT中,减少的N-糖蛋白可能是因为携带复杂N-聚糖的糖蛋白的富集程度低于突变体中具有高甘露糖型 N-聚糖的糖蛋白。然而,在高盐度存在的情况下,WT中检测到的糖蛋白数量略有增加(从203到244),但在mns1mns2(从311到297)和cgl1-3(从300到263;图1)中出现减少。一种可能性是响应盐胁迫的糖蛋白的丰度受到附着的N-聚糖的调节,N-聚糖的欠成熟使盐胁迫条件下相关的糖蛋白不稳定,这与之前的研究一致,即N-聚糖的成熟对植物耐受盐胁迫至关重要。
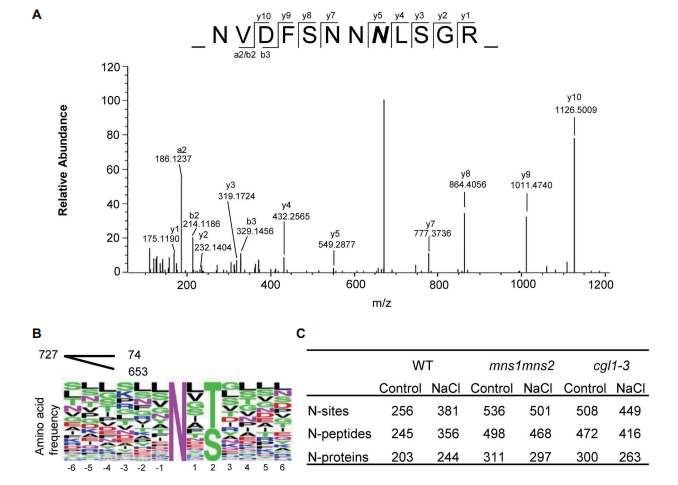
图1 鉴定到的糖肽和蛋白质概览
文中进一步分析了盐胁迫下DAG在GO注释和KEGG代谢途径中的生物学功能。在两个突变体中观察到更多减少的糖蛋白,尤其是在cgl1-3中(图2 A–C)。值得注意的是,与 WT 相比, mns1 mns2和cgl1-3突变体中的水解酶均得到富集(图2 D–F)。在盐胁迫下,WT和两个参与内质网聚糖链转移的突变体中 STT3A(OST亚基)、紫色酸性磷酸酶 2(PAP2)和类胚蛋白10(GLP10)的丰度发生了变化,这些蛋白分别参与了根系生长和碳代谢(图2 D)。STT3A在 WT中增加,但在mns1 mns2和cgl1-3中减少;PAP2 在 WT 和mns1mns2中增加,但在cgl1-3中减少,并且GLP10在所有三个样本中都增加。进一步的分子功能分析发现,与WT相比,水解酶活性在mns1 mns2和cgl1-3中富集(图2 E,G)。WT中鉴定的蛋白质的生物学过程多与应激反应相关,而来自突变体的大多数DAG与碳水化合物代谢过程有关(图3 G)。这些结果表明,mns1 mns2和cgl1-3突变体中的盐响应糖蛋白不能正确响应压力,这可能是由于缺乏正确的N-聚糖修饰,导致它们的亚细胞定位异常。
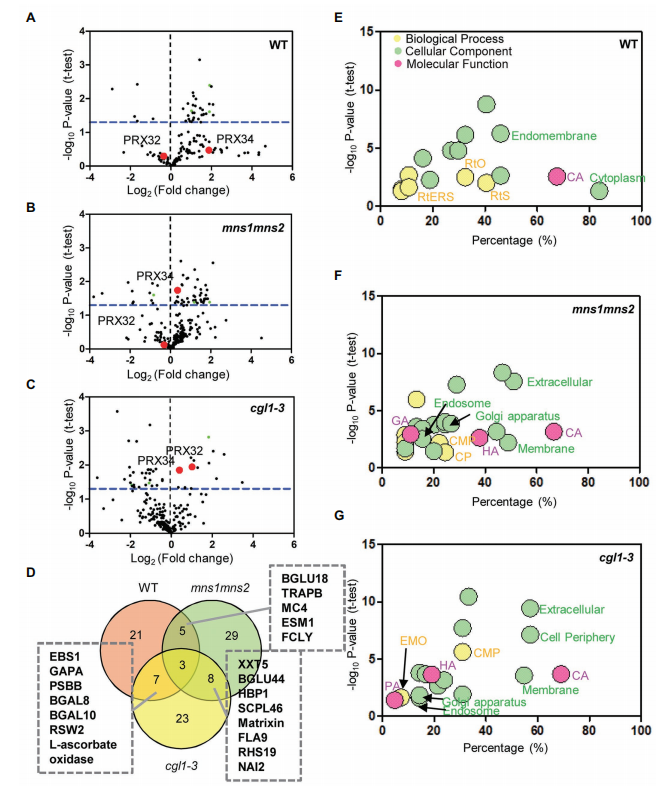
图2 盐胁迫下不同丰度差异糖蛋白的GO注释
研究结论
许多证据表明,植物对盐胁迫的适应性反应需要相关蛋白上加工成熟的N-聚糖参与。然而,关于在这个过程中起作用的盐响应糖蛋白知之甚少。本研究鉴定了野生型 (WT)拟南芥中的盐响应糖蛋白和两个N-聚糖成熟缺陷的突变体mns1 mns2和cgl1。通过液相色谱串联质谱 (LC-MS/MS) 进行定量分析,共鉴定出97种丰度变化 > 1.5 的蛋白质对抗盐胁迫。表明N-聚糖对于调节应激反应蛋白水平至关重要,并且列出了几种负责拟南芥耐盐胁迫的新型糖蛋白。
蛋白质组学在植物研究中的应用案例二
中文标题:通过非标记label free定量蛋白质组学揭示菊芋(Helianthus tuberosus L.)块茎蛋白质组
研究对象:菊芋块茎
期刊:Molecules
运用技术:非标记label free定量蛋白质组学研究背景Helianthus tuberosus L.,俗称菊芋(简称JA),是向日葵科菊科(菊科)的一年生草本植物。这种植物以其对环境压力的抵抗力而闻名,并且在不同的土壤(干旱-盐碱)和温度(冷-热)条件下茁壮成长,这使它在世界大部分地区都有种植。除此之外,该植物还因其在原料、功能性食品、生物质、生物医学和化学工业中的重要性而闻名。该研究小组一直致力于将菊芋块茎作为日常生活中的传统/替代药物进行栽培和利用,并研究降低人体受试者的血糖水平、HbA1c等。了解块茎的蛋白质成分可能会揭示其愈合特性,尤其是与糖尿病有关的特性。本文使用三种商业加工的JA块茎产品(干粉和干片)进行了非标记label free定量蛋白质组学研究。
研究技术流程
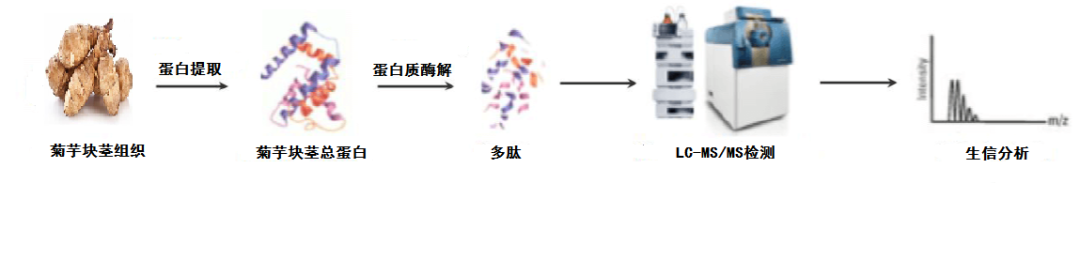 研究结果
研究结果
无标记定量蛋白质组学方法用于分析三个块茎样品之间的差异蛋白质丰度。去除污染物和低置信度蛋白后,共鉴定出2967个高置信度蛋白组(图1 A)。由于使用了三种不同的块茎样品,在MS运行后预计会出现多个缺失值;因此,在三个重复中的两个(2102个蛋白质组)中可重复鉴定的蛋白质组被选择并考虑进行进一步分析(图1 A)。
维恩图分析显示1702个蛋白质组通常在所有三个块茎样品中鉴定,而89、36和54个蛋白质组分别仅在块茎 1、2和3中鉴定。此外,块茎 1 和块茎 2 共有 112 个蛋白质组,表明它们的蛋白质组与块茎对 (2,3) 和 (3,1) 相比更密切相关(图1 B)。为了检查同一样品一式三份中无标记蛋白质定量的重现性,使用 Perseus软件生成多散点图(图1 C)。相同样本的散点图显示出典型的非均匀分布,即在较低强度时较宽,在较高强度时指向较高。同一样品的不同重复的皮尔逊相关系数大于0.944,表明同一样品的三次重复之间具有高度相关性。直方图显示了缺失值插补前后的蛋白质计数 (图1 D)。
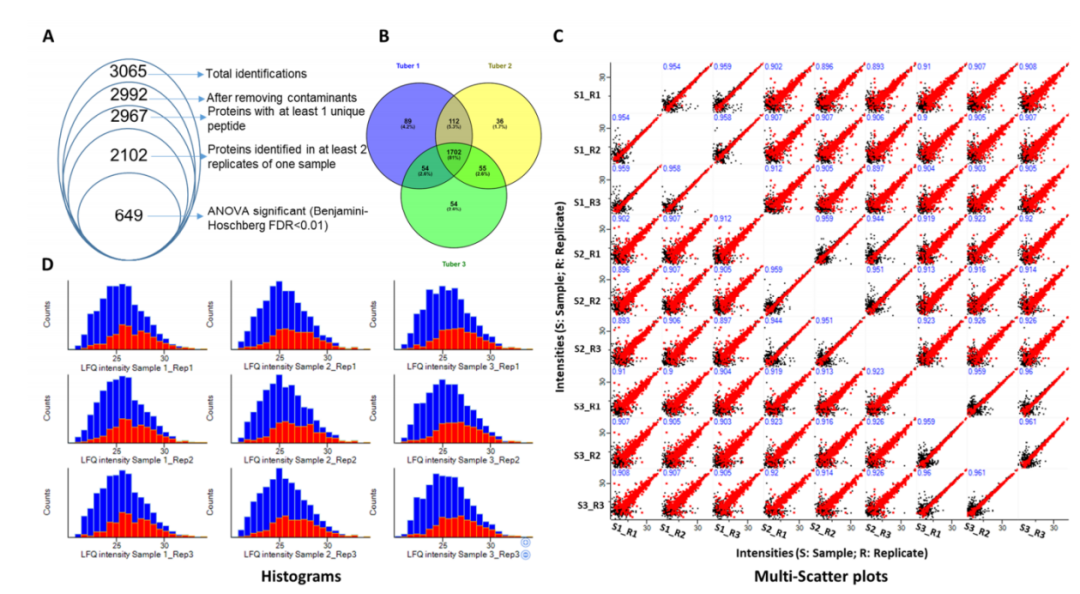
图1 三种(1, 2, 3)不同JA块茎的蛋白质组学差异概览
层次聚类分析将差异蛋白分成四个簇,每个簇都有不同的表达谱(图2 A)。簇1包含61种蛋白质,在块茎3中主要下调,而簇1的891种蛋白质主要在块茎2中上调。簇3显示块茎3中丰度增加的379种蛋白质,而簇4中的771种蛋白质在块茎 1 中显示最大丰度。显示前48种差异调节蛋白质丰度模式的热图位于 (图2 B)。在这48个DEs中鉴定了多种具有独特功能的蛋白质。共有19、24和34种DE蛋白受到高度调节,而块茎1-3中分别有27、22和14种蛋白质降低。结果表明,块茎 1 和块 3 的蛋白质含量非常不同,而块茎2的结果则参差不齐。此外,偏最小二乘判别分析 (PLS-DA) 将成分 1 中的块茎1和3分开,占总方差的32.7%,而成分2中的块茎2与块茎1和3分开,占总方差的 25.5%总方差的百分比 (图2 C)。最后确定了有助于PLS-DA图分离的前15种蛋白质(图2 D)。
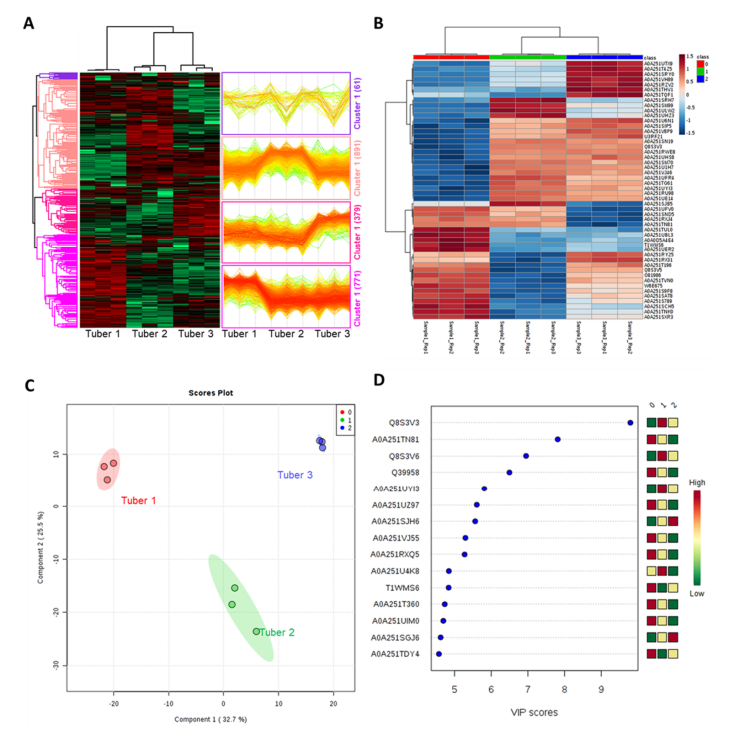
图2 JA块茎蛋白组的统计学分析
统计分析后,作者进行基于基因本体(GO)的功能分类,在2102种蛋白质中,PANTHER 19.0 针对Helianthus annus数据库检测到2044种蛋白质。接下来,进行 PANTHER 通路分析以对蛋白质通路进行分类,发现了大多数蛋白质归因于泛素蛋白体和核苷酸(嘌呤)生物合成途径,其次是TCA循环、糖酵解、丙酮酸代谢、细胞周期和嘧啶核糖核苷酸途径。
本文还确定了几种直接或间接参与各种疾病相关机制的蛋白质。三种蛋白质命中被确定与阿尔茨海默病相关,并发现 11 个蛋白质属于亨廷顿病通路。除此以外,作者还确定了众所周知的具有治疗意义的蛋白质,当作为膳食蛋白质来源或通过口服补充剂食用时,这些主要用于治疗糖尿病和癌症等慢性疾病。还鉴定了一些抗微生物蛋白(图3)。
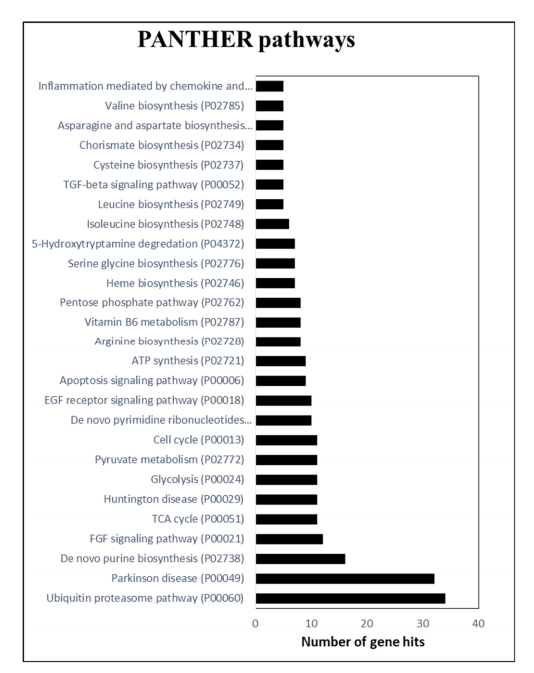
图3 使用PANTHER工具对块茎蛋白质组进行GO通路分析
研究结论
本文使用基于高通量无标记LC-MS/MS的组学技术对来自两种不同菊芋加工技术,包括干粉(块茎 1 和 2)和干片(块茎 3)的块茎蛋白质组进行了研究。鉴定出的蛋白质及其分类表明了支持了先前的研究数据(尚未发表)的功能:数据表明菊芋对血糖水平有降低作用,以及与人类糖化血红蛋白 (Hb-A1c) 的关系,预防老年痴呆以及治疗亨廷顿病等功效。
参考文献
1. Liu C, Niu G, Li X, Zhang H, Chen H, Hou D, Lan P, Hong Z. Comparative Label-Free Quantitative Proteomics Analysis Reveals the Essential Roles of N-Glycans in Salt Tolerance by Modulating Protein Abundance in Arabidopsis. Front Plant Sci. 2021 Jul 2;12:646425. doi: 10.3389/fpls.2021.646425.
2. Bakku RK, Gupta R, Min CW, Kim ST, Takahashi G, Shibato J, Shioda S, Takenoya F, Agrawal GK, Rakwal R. Unravelling the Helianthus tuberosus L. (Jerusalem Artichoke, Kiku-Imo) Tuber Proteome by Label-Free Quantitative Proteomics. Molecules. 2022 Feb 7;27(3):1111. doi: 10.3390/molecules27031111.









 京公网安备 11011302003368号
京公网安备 11011302003368号