如何进行细胞轨迹分析?
目前进行细胞轨迹分析的方法和软件非常之多,大致可以概括为两种方法,一种是以monocle软件为代表的拟时序分析(pseudotime analysis),另一种则是以velocyto /scVelo为主的RNA速度分析(RNA velocity)。
1、拟时序分析
2019年Nature biotechnology的一篇文章A comparison of single-cell trajectory inference methods [1] 就对45种拟时序分析的方法进行了评估,这些软件在性能、可扩展性、稳定性和易用性上存在差异,当然也各有其优缺点。软件推断的细胞分化轨迹主要有7种类型,包括环状(cycle)、线性(linear)、分叉(bifurcation)、多分叉(Multifurcation)、树型(Tree)、多种轨迹并存的连接图(Connected graph),及多种不相连轨迹并存的断开图(disconnected graph)。(图1)
![图1 轨迹分析算法评估流程及7种细胞分化轨迹类型[1]](http://www.biomarker.com.cn/wp-content/uploads/2022/02/640.png)
图1 轨迹分析算法评估流程及7种细胞分化轨迹类型[1]
2、RNA速度分析
目前绝大多数软件利用的是细胞的基因表达信息,2018年发表在Nature上的一篇文献RNA velocity of single cells[3],提出了一个新的概念,即RNA速度(RNA velocity ),利用的是reads到表达的信息,通过分析新转录的、未发生剪接的前体mRNA,与成熟的、发生剪接的mRNA(reads是否比对到内含子)丰度之间的比值,来表征细胞定向的动态信息。scVelo[4]软件可以推断包括转录、剪接和降解在内的”基因特异性速度(gene-specific rates)”,并还原细胞过程中的”潜在时间(latent-time)”,而潜在时间近似为细胞分化的真实时间,同时可以识别调控机制,并系统性的推定驱动基因。scVelo分析流程及代码贴在文章末尾,感兴趣的老师不妨实操看看。
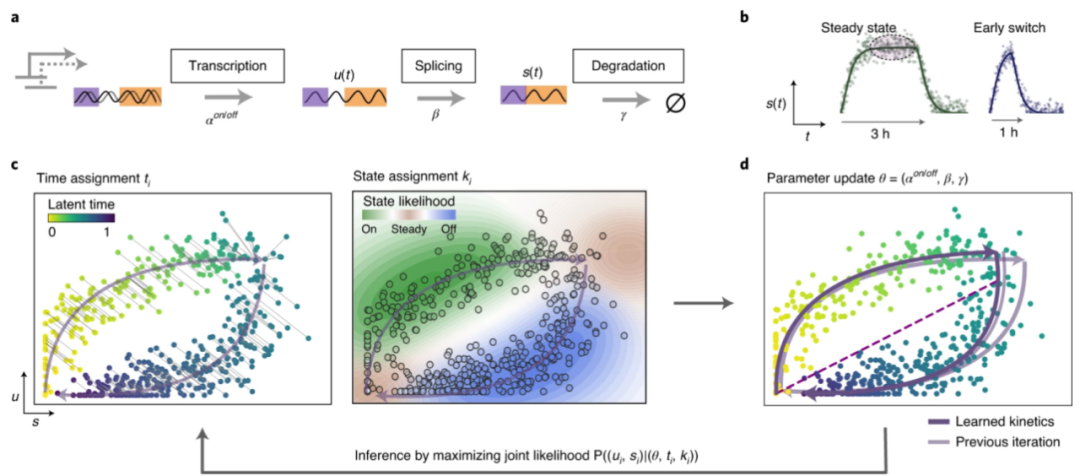
图2 scVelo软件分析原理示意图[4]
相较于拟时序分析来说,RNA velocity分析的是细胞在某一时刻的潜在分化方向,可以直接从具有时间向量的结果读取到分化方向,无需基于生物学背景指定分化起点和终点;同时RNA velocity的数据可以与Seurat降维数据兼容,也可以嵌入monocle构建细胞轨迹,这对于联合两种分析策略十分便利,利用RNA velocity分析揭示所有未知的细胞分化关系,然后筛选关注的细胞分化过程进行拟时序分析,挖掘分化过程中的调控机制。
如何应用细胞轨迹分析?
1、细胞分化过程重现
基于单细胞测序分析数据,对其他技术手段获取的已知细胞分化关系进行验证。
2020年Nature Communication发表的Single-cell RNA-seq reveals that glioblastoma recapitulates a normal neurodevelopmental hierarchy[5],利用RNA速度表征了胶质母细胞瘤的分化过程,证实了胶质母细胞瘤层次结构顶点是肿瘤祖细胞,星形细胞、间充质、少突胶质细胞和神经元癌细胞比肿瘤祖细胞的分化程度更高,并且在每个患者中均可见特定细胞分化方向(图3)。
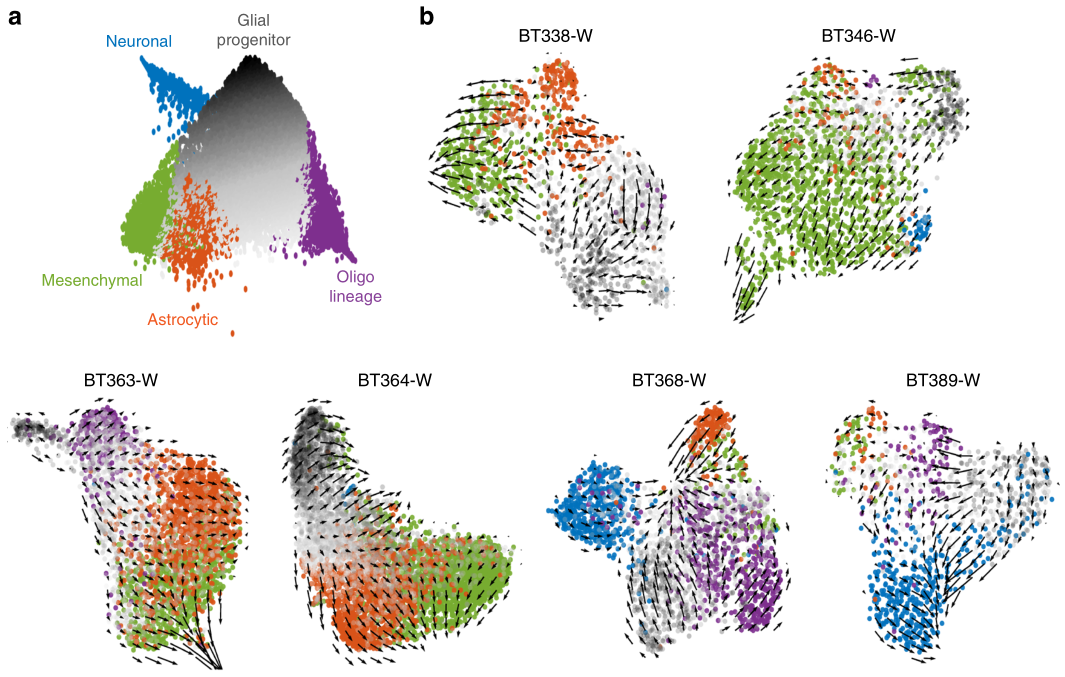
图 3 胶质母细胞瘤RNA速度分析[5]
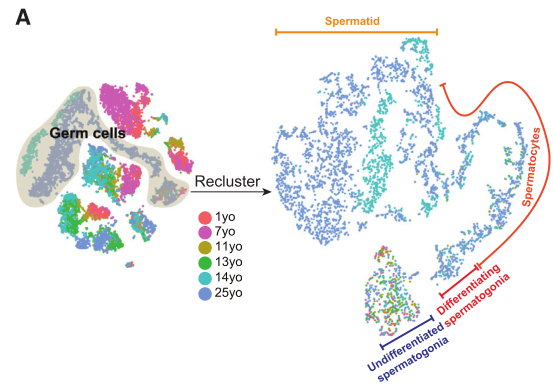
图4 人睾丸生殖细胞亚群及分化轨迹鉴定[6]
2、潜在的细胞分化路径挖掘
除了鉴定已知的细胞分化过程,通过细胞轨迹分析也能推断未被报道的、可能的未知细胞分化路径,如神经干细胞发育、肿瘤/疾病微环境中免疫细胞分化过程,甚至挖掘到一些稀少的、常规技术手段很难获得的中间状态细胞。
2021年Nature Communication发表的Single-cell sequencing of immune cells from anticitrullinated peptide antibody positive and negative rheumatoid arthritis[7],研究首先通过比较两种亚型类风湿性关节炎(RA)患者与健康对照外周血免疫细胞图谱差异,关注到了B细胞类型丰度变化,发现ACPA-RA组IGHG4+ plasma B细胞明显升高,HLA-DRB5+ plasma B细胞明显降低(图5,左图);随后对B细胞进行拟时序分析探究B细胞亚型分化关系,发现在健康对照和RA患者之间B细胞亚型存在不同的分化路径:在健康对照中naïve B细胞有两条分化路径,1条是分化为HLA-DRB5+plasma B细胞再分化为plasma B细胞,另1条分化为Mem-unsw B细胞到Mem-sw B细胞;而RA患者中,naïve B细胞直接分化为IGHG4+ plasma B细胞(图5,右图)。
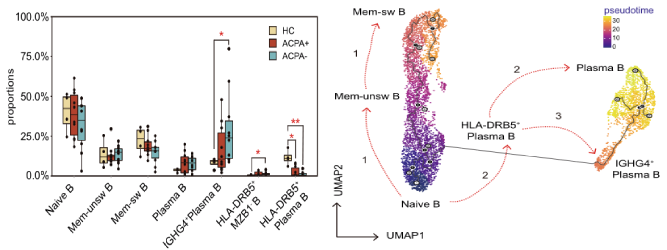
图5 类风湿关节炎患者外周血B细胞分析[7]
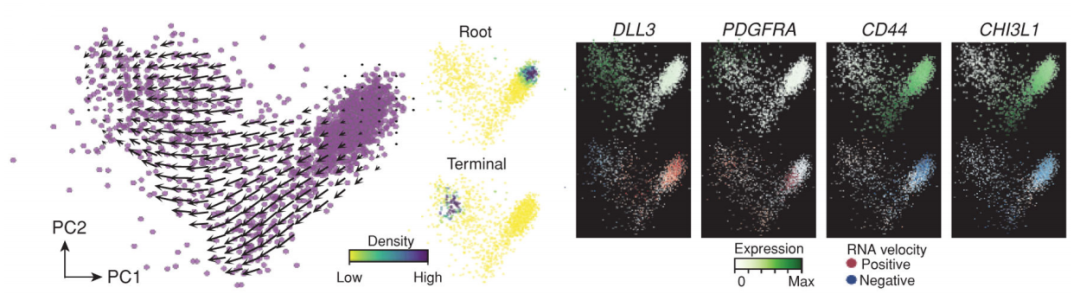
图6 胶质母细胞瘤干细胞样细胞分化轨迹[8]
3、细胞分化过程调控机制解析
通过细胞轨迹分析,分析处于重要分化节点的关键细胞亚群,寻找随着分化时间呈现特定表达模式的关键基因,解析驱动细胞亚群分化的调控机制。
2021年 Nature发表的Single-cell transcriptomic characterization of a gastrulating human embryo[9],为了探索原肠胚形成过程中外胚层的变化,利用RNA速度分析了外胚层(Epiblast)、外胚层(羊膜/胚胎)(Ectoderm (amniotic/embryonic))、新生中胚层(Nascent mesoderm)、原条(Primitive Streak),由Epiblast起,发生两个分支,一侧由Primitive Streak发育到Nascent mesoderm,一侧发育到ectoderm(图7,左图);随后利用拟时分析推断Epiblast分化过程中的基因表达变化,检测到ectoderm marker genes(DLX5、TFAP2A 和GATA3)表达上调,而早期神经诱导marker genes(SOX1、SOX3 和PAX6)和分化的神经元marker genes(TUBB3、OLIG2和NEUROD1)表达极低甚至检测不到,这表明CS7中神经分化尚未开始(图7,右图)。
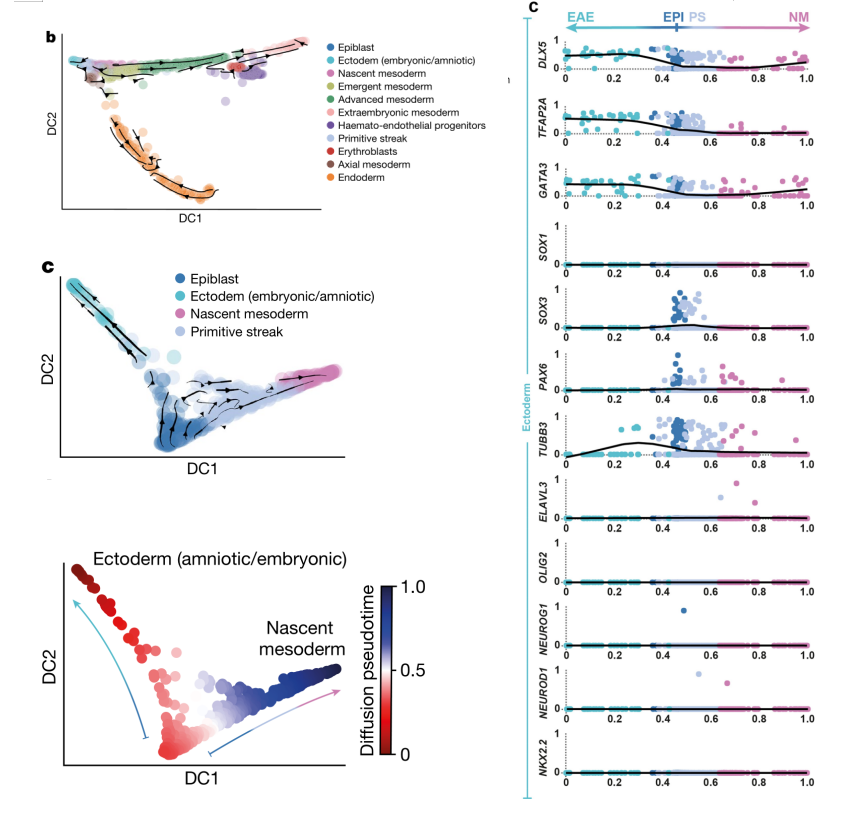
图7 RNA速度分析解析原肠胚分化轨迹[9]
如何进行RNA速度分析?
1、软件安装:
pip install -U scvelo #需要3.6以上版本python
pip install velocyto
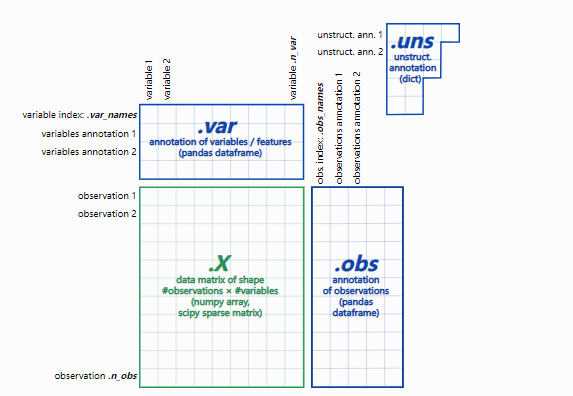
2、adata 数据结构:
– adata.X:数据矩阵
– adata.obs:细胞的注释
– adata.var:基因的注释
– adata.nus:非结构化注释,例如图
– adata.layers:附加层数据,如spliced/unspliced矩阵
3、加载需要的库及一些配置
import seaborn as sns
import scvelo as scv
import pandas as pd
import scanpy as sc
import numpy as np
import loompy
scv.settings.verbosity = 3 # show errors(0), warnings(1), info(2), hints(3)
scv.settings.presenter_view = True # set max width size for presenter view
scv.set_figure_params(‘scvelo’) # for beautified visualization
4、计算获取unspliced/spliced矩阵
## 运行run10x
/software/python3.9.6/bin/velocyto run10x \
# 相应的repeatmasker的重复序列注释文件(可从USCU中获取)
# http://genome.ucsc.edu/cgi-bin/hgTables
-m RMSK
cellranger_results # cellranger的输出结果路径
sample_name # 样本名
seurat_GTF # 用于cellranger定量的基因组注释文件
## 输出结果,在cellranger的结果中会创建单个样本的velocyto文件夹,其中的loom文件即为最终结果
data/SC/Normal1-sc/velocyto:
Normal1-sc.loom
data/SC/Normal2-sc/velocyto:
Normal2-sc.loom
data/SC/Normal3-sc/velocyto:
Normal3-sc.loom
data/SC/Normal4-sc/velocyto:
Normal4-sc.loom
5 、loom文件的读取及合并
# 读取单个样本loom文件
Normal_loom = sc.read(‘./Normal1.loom’, sparse=True, cache=True)
# 合并同组样本的loom文件
import loompy
Normal_loom_list = [Normal1-sc.loom,
Normal2-sc.loom,
Normal3-sc.loom,
Normal4-sc.loom]
loompy.combine(Normal_loom_list, output_file = ‘Normal_combined.loom’)
# 读取
Normal_loom = sc.read(‘./Normal_combined.loom’, sparse=True, cache=True)
6 导入Seurat对象的降维聚类结果
使用scVelo进行分析时需要对数据进行降维聚类,若上游分析的降维聚类注释结果基于Seurat,可以直接将Seurat的降维聚类结果添加到adata对象中,下面方法仅供参考。此外,如果上游分析基于scanpy,则可以直接调用scanpy的降维聚类相关的方法。注:此处并没有进行注释,在实际操作中将 seurat_cluster替换为注释后的信息即可,导入seurat之后的所有调用的方法中的groupby参数设置为 seurat_cluster。
library(Seurat)
library(tidyverse)
seurat_obj@meta.data %>% rownames_to_column(var = ‘barcodes’) %>%
write.table(‘seurat_meta_df.txt’, sep = ‘\t’, quote = F)
seurat_obj@reductions$umap@cell.embeddings %>% rownames_to_column(var = ‘barcodes’) %>%
write.table(‘seurat_umap.txt’, sep = ‘\t’, quote = F)
def using_seurat_meta_umap(loom_file, seurat_meta_df, seurat_umap):
# 读入seurat的meta.data以及umap降维结果
seurat_meta_data_raw = pd.read_csv(seurat_meta_df)
seurat_umap_raw = pd.read_csv(seurat_umap)
# loom文件原始的index顺序
anndata_index = loom_file.obs.index.values
# 合并
meta_data = loom_file.obs.reset_index(). \
merge(seurat_meta_data_raw,
left_on=”CellID”,
right_on=”barcodes”, how=’left’). \
set_index(‘CellID’).reindex(anndata_index)
meta_data[‘seurat_clusters’] = meta_data[‘seurat_clusters’]. \
apply(lambda x: “%d” % int(x) if ~np.isnan(x) else x).astype(‘category’)
loom_file.obs = meta_data
loom_file = loom_file[loom_file.obs.dropna().index, :]
loom_file.obsm[‘umap’] = seurat_umap_raw.set_index(‘barcodes’). \
reindex(loom_file.obs.index).to_numpy().astype(‘float64’)
return loom_file
Normal_loom = using_seurat_meta_umap(Normal_loom,
‘seurat_meta_df.txt’,
‘seurat_umap.txt’)
# 查看是否替换成功,与seurat结果进行比较
sc.pl.umap(Normal_loom, color=’seurat_clusters’)
分析结果展示
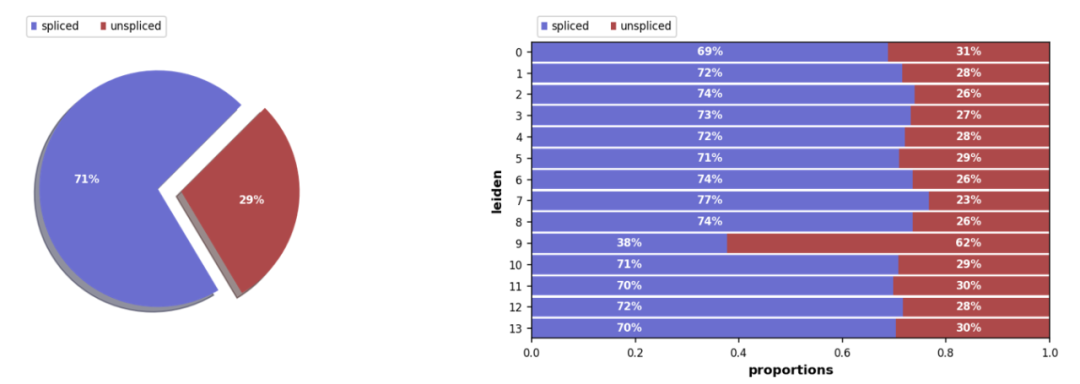
拼接/未拼接计数的比例,通常有 10%-25% 的未拼接分子包含内含子序列。
scv.pl.proportions(Normal_loom, groupby=’seurat_clusters’)
RNA-速度:速度是基因表达空间中的向量,代表单个细胞运动的方向和速度。速度是通过模拟剪接动力学的转录动力学获得的,对于每个基因,拟合了成熟(未剪接)和成熟(剪接)mRNA 计数的稳态比率,这构成了恒定的转录状态。正速度表明基因被上调,这种情况发生在该基因未剪接 mRNA 丰度高于预期的稳定状态的细胞中。相反,负速度表明基因被下调。
# 预处理
scv.pp.filter_genes(Normal_loom, min_shared_counts=20)
scv.pp.normalize_per_cell(Normal_loom)
scv.pp.filter_genes_dispersion(Normal_loom, n_top_genes=2000)
scv.pp.log1p(Normal_loom)
scv.pp.filter_and_normalize(Normal_loom,
min_shared_counts=20,
n_top_genes=2000)
# 运行scVelo动态模型,如果算力不够可以考虑静态或者随机方法
scv.tl.recover_dynamics(Normal_loom, n_jobs=30)
scv.tl.velocity(Normal_loom, mode=’dynamical’)
scv.tl.velocity_graph(Normal_loom, n_jobs=n_jobs)
# 保存计算结果
Normal_loom.write(‘Normal_dy_model.h5ad’, compression=’gzip’)
scv.pl.velocity_embedding_stream(Normal_loom,
basis=’umap’,
color=’seurat_clusters’)
scv.pl.velocity_embedding(Normal_loom,
arrow_length=3,
arrow_size=2,
show=False,
color=’seurat_clusters’)
潜伏时间:动力学模型描述了细胞过程的潜伏时间(这个潜伏时间代表细胞的内部时钟,并近似于细胞在分化时经历的真实时间,仅基于其转录动力学。)
scv.tl.latent_time(Normal_loom)
scv.pl.scatter(Normal_loom, color=’latent_time’,
color_map=’gnuplot’, size=80, show=False)
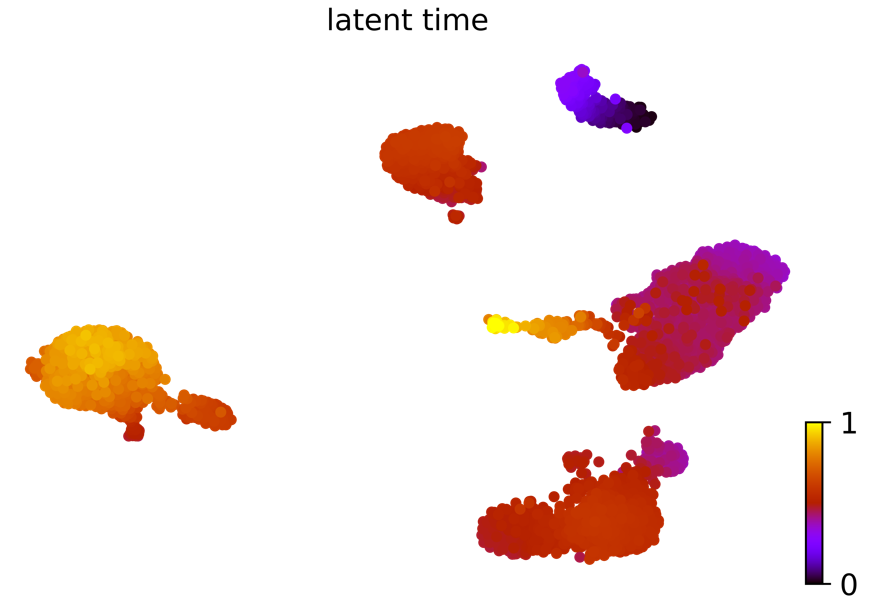
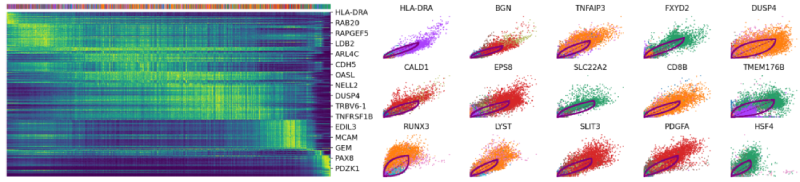
驱动基因:显示出明显的动态行为,并通过动态模型中的高可能性特征系统地检测到。
top_genes = Normal_loom.var[‘fit_likelihood’]. \
sort_values(ascending=False).index[:300]
scv.pl.heatmap(Normal_loom,
var_names=top_genes, sortby=’latent_time’,
col_color=’seurat_clusters’, n_convolve=100)
scv.pl.scatter(Normal_loom, basis=top_genes[:15],
ncols=5, frameon=False,
color=’seurat_clusters’)
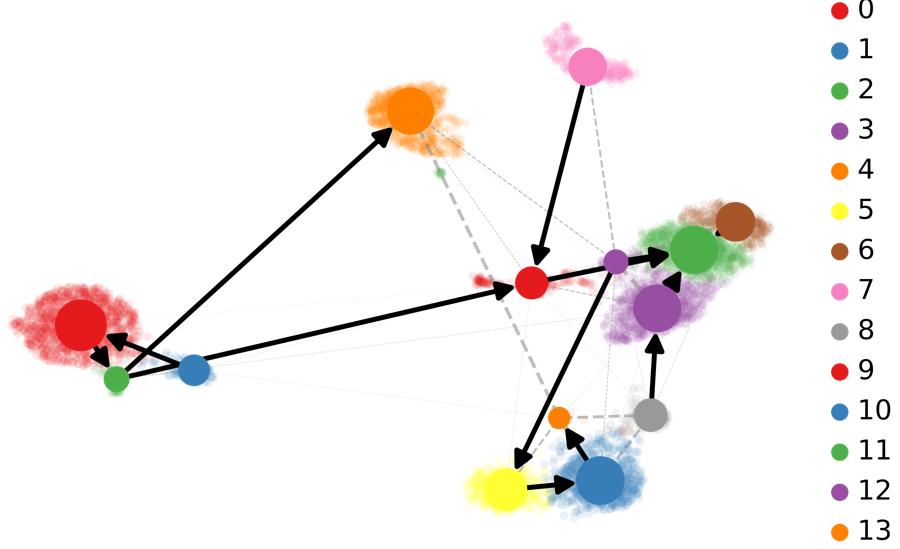
基于RNA-速度的PAGA分析:PAGA提供了轨迹推断构图的方法,其中加权边对应于两个集群之间的连接。在这里,PAGA通过速度推断的方向性进行了扩展。
scv.tl.paga(Normal_loom, groups=’seurat_clusters’)
scv.pl.paga(Normal_loom, basis=’umap’,
size=50, alpha=.1,
min_edge_width=2, node_size_scale=1.5)
 参考文献:
参考文献:






 京公网安备 11011302003368号
京公网安备 11011302003368号