单细胞转录组测序是在单细胞水平进行高通量基因表达谱检测的技术,可以对复杂细胞群深入分析,表征单个细胞的表达谱,避免单个细胞的异质性生物学信息被大量细胞的均质化覆盖。通过单细胞转录组,可鉴定细胞群、细胞亚群、筛选细胞群特有marker基因,并完成对细胞分化轨迹的追踪,从而解析细胞异质性和发育轨迹。但是单纯通过单细胞测序得到的细胞分群,是不能将恶性与非恶性细胞区分开的。因此单细胞CNV分析应运而生,可以辅助区分肿瘤组织中的恶性与非恶性细胞。
什么是infer CNV

InferCNV是TrinityCTAT工具包的一个组成部分,可以用于肿瘤scRNA-seq数据中鉴定大规模染色体拷贝数变异(copy number variation,CNV),例如整个染色体或大片段染色体的扩增或缺失。它的原理是:用一组“正常”细胞(如癌旁组织来源的细胞)作为对照,分析肿瘤基因组上各个位置的基因表达量强度变化,通过热图的形式展示每条染色体上的基因的相对表达量,那么相较于正常细胞,肿瘤基因组会表现出过表达或者低表达。
Infer CNV的输入数据如下:
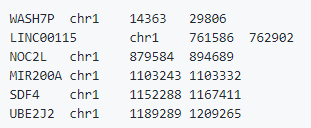
(1) scRNA-seq表达原始矩阵:该矩阵为基因(行)与细胞(列)矩阵,是以制表符分隔的文件。
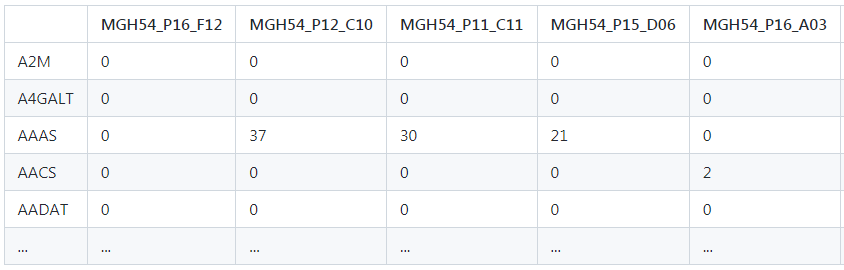
(2) 注释文件:用于定义肿瘤和正常细胞。共两列,第一列是cluster,第二列表示已知细胞类型,以制表符分隔。
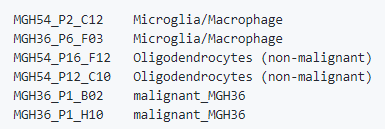
(3) 基因或染色体位置文件:提供每个基因的染色体位置。文件共4列,分别是基因名称、染色体和基因跨度,文件以制表符分隔。
代码实操
1.安装infercnv
2.Docker安装
3.sudo docker pull trinityctat/infercnv:latest\
4.R安装
5.安装infercnv之前需要安装软件JAGS(Just Another Gibbs Sampler)
1.##安装infercnv
2.if (!requireNamespace(“BiocManager”, quietly = TRUE))
3. install.packages(“BiocManager”)
4.BiocManager::install(version = “devel”)
5.BiocManager::install(“infercnv”)
6.library(infercnv)
7.##将制作好的文件作为输入
8.#raw_counts_matrix:scRNA-seq表达原始矩阵
9.#annotations_file:注释文件
10.#ref_group_names:基因或染色体位置文件
11.infercnv_obj = CreateInfercnvObject(raw_counts_matrix=system.file(“extdata”, “oligodendroglioma_expression_downsampled.counts.matrix.gz”, package = “infercnv”),
12. annotations_file=system.file(“extdata”, “oligodendroglioma_annotations_downsampled.txt”, package = “infercnv”),
13. delim=”\t”,
14. gene_order_file=system.file(“extdata”, “gencode_downsampled.EXAMPLE_ONLY_DONT_REUSE.txt”, package = “infercnv”),
15. ref_group_names=c(“Microglia/Macrophage”,”Oligodendrocytes (non-malignant)”))
16.
17.##将infercnv_obj对象作为输入
18.infercnv_obj = infercnv::run(infercnv_obj,
19. cutoff=1, # cutoff=1 works well for Smart-seq2, and cutoff=0.1 works well for 10x Genomics
20. out_dir=tempfile(),
21. cluster_by_groups=TRUE,
22. denoise=TRUE,
23. HMM=TRUE)
24.##输出文件在当前文件夹
https://sourceforge.net/projects/mcmc-jags/files/JAGS/
https://github.com/broadinstitute/inferCNV/wiki/Installing-infercnv
以上来自InferCNV官方:https://github.com/broadinstitute/inferCNV/wiki
infer CNV的应用
1、胶质母细胞瘤中的恶性细胞分析
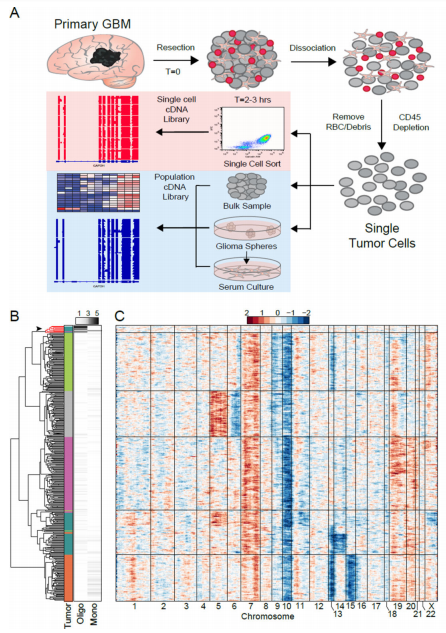
2014年发表在science(IF=47.728)上的《Single-cell RNA-seq highlights intratumoral heterogeneity in primary glioblastoma》[1],通过对基因相对表达水平来推断每个细胞的大规模拷贝数变异(CNV)。该研究选择正常人的脑组织bulk RNA-seq的数据作为对照,单细胞样本聚为7组,无监督分析确定了9个异常细胞,它们具有成熟少突胶质细胞基因表达增加和胶质母细胞瘤基因下调的特点,剩余的420个细胞中没有一个显示出与非恶性细胞或免疫细胞类型的转录程序相似的特征。虽然非恶性细胞是肿瘤微环境的关键组成部分,但该研究通过解离和CD45+分选后,已经很大程度上排除了非肿瘤细胞。该研究通过infer CNV有效地将脑胶质母细胞瘤中的正常细胞与恶性细胞区分开来。
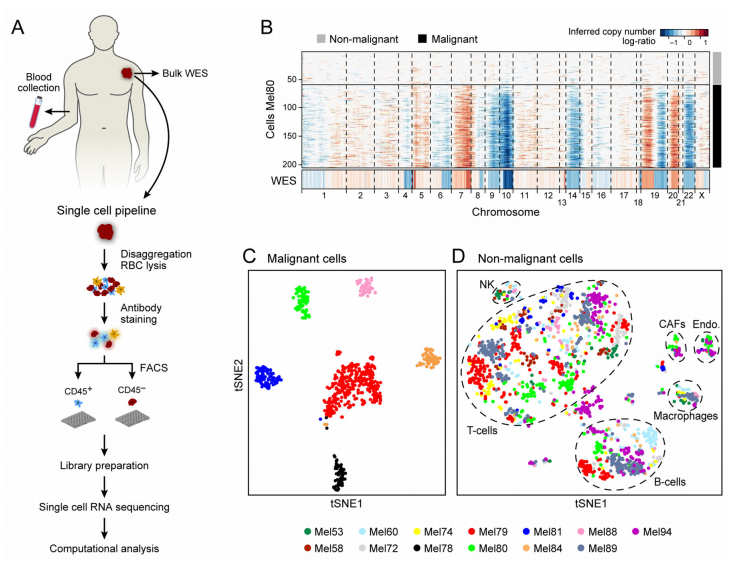
2、转移性黑色素瘤的恶性细胞分析
2016年发表在science(IF=47.728)上的《Dissecting the multicellular ecosystem of metastatic melanoma by single-cell RNA-seq》[2],使用多步骤方法根据遗传和转录状态来区分黑色素瘤中的不同细胞类型。首先通过对各染色体上100个基因片段的表达进行平均,从表达谱中推断出大规模拷贝数变异(CNV)。其次,根据细胞的表达谱对细胞进行分群,结合CNV分析,发现恶性细胞形成一个单独的簇,表明高度的肿瘤间异质性。相比之下,按细胞类型聚集的非恶性细胞分别被注释为T细胞、B细胞、巨噬细胞、内皮细胞、癌症相关成纤维细胞(CAF)和NK细胞。针对恶性细胞,该研究后期进行细胞周期分析,筛选处于高分类状态的细胞,查看它们的分子表达情况;对于非恶性细胞,该研究主要讨论了其与黑色素瘤微环境的相互作用。
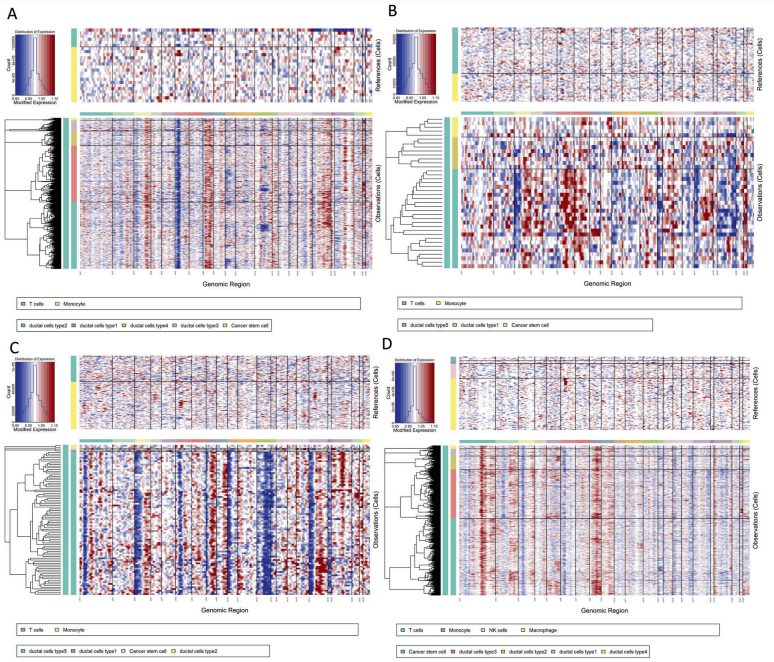
3、转移性胰腺导管腺癌的肿瘤内异质性分析
2021年发表在Cell Death Discovery(IF=5.241)上的《Single-cell RNA transcriptome reveals the intra-tumoral heterogeneity and regulators underlying tumor progression in metastatic pancreatic ductal adenocarcinoma》[3],基于每个样本的inferCNV计算和识别大规模染色体拷贝数变异(CNV),得到CNV热图,结果发现与对照样品相比,5种导管细胞亚型经历了显著的CNV事件,其中1型导管细胞的CNV水平显著高于其他类型的导管细胞。这些结果表明导管细胞的5种亚型都是转移性病变中的恶性细胞。针对筛选到的恶性细胞,着重研究其转录异质性,结果发现,恶性导管细胞和癌症干细胞具有不同的基因表达谱,癌症干细胞上调表达干性特征基因,包括ALDH1A1、PROM1和NES,而导管谱系细胞cluster上调表达导管marker,如MMP7、TSPAN8和SOX9。
 参考文章:
参考文章:
1. Anoop P. Patel, Itay Tirosh, et al. Single-cell RNA-seq highlights intratumoral heterogeneity in primary glioblastoma. Science. 2014 Jun 20: 1396-1401.
2. Tirosh I et al. Dissecting the multicellular ecosystem of metastatic melanoma by single-cell RNA-seq. Science. 2016 Apr 8;352(6282):189-96.
3. Qianhui Xu et al. Single-cell RNA transcriptome reveals the intra-tumoral heterogeneity and regulators underlying tumor progression in metastatic pancreatic ductal adenocarcinoma. Cell Death Discov. 2021 Nov 3;7(1):331.
百迈客单细胞转录组产品介绍
百迈客单细胞转录组测序产品基于10x Genomics系统利用微流体系统和油滴包裹技术,在单细胞水平进行高通量基因表达谱检测,对复杂细胞群深入分析,表征单个细胞的表达谱,避免单个细胞的异质性生物学信息被大量细胞的均质化覆盖。通过单细胞转录组,可鉴定细胞群、细胞亚群、筛选细胞群特有marker基因,并完成对细胞分化轨迹的追踪,从而解析细胞异质性和发育轨迹。
如果您对我们的服务感兴趣,欢迎点击下方按钮联系我们,我们将免费为您设计文章思路方案。









 京公网安备 11011302003368号
京公网安备 11011302003368号