GWAS研究背景
在数量遗传学领域,GWAS通常用于鉴定与特定数量性状相关的DNA分子标记。通过鉴定关联群体中基因型数据和关注性状的表型数据,然后利用合适的模型/软件对数据进行统计分析,从而确定性状-标记关联位点。研究表明这些位点对诊断和预测数百种人类疾病(甚至对新生儿)非常有用。同样,在牲畜/作物改良研究中,这些位点也通常用于动植物的标记辅助选择(MAS)育种。所以,GWAS研究在近20年一直是非常火的研究领域(图1)。
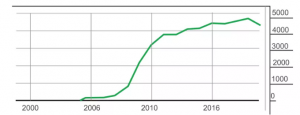 图1 2000年到2019年12月,“GWAS”一词在PubMed文章中每年出现的次数
图1 2000年到2019年12月,“GWAS”一词在PubMed文章中每年出现的次数
GWAS标记类型
在用于GWAS的标记中,最初使用的是SSR标记,后来SNP标记成为常用标记。但是,研究人员逐渐意识到基于SNP标记的GWAS分析不足以解释的群体中全部的遗传变异。并且,随着测序技术的发展,在人以及诸多动植物中发现了大规模的SVs [包括拷贝数变异(CNV)/存在缺失变异(PAV)和易位/倒置(translocations/inversions)]与疾病、驯化及重要农艺性状有关。本期推文,我们一起来看一下SVs在动植物群体研究中的进展。
SV和SV-GWAS在动物中的应用
近年来,动物遗传变异研究中,在牛、猪、狗、小鼠等物种中鉴定出了大量的SVs。例如,在62头牛组成的群体中,鉴定得到了6,426个SVs,部分SVs位于乳品性状的QTL内;利用全基因组测序数据构建了猪的SVs变异图谱,在分布全球的55个猪种的305个个体中鉴定得到了33,698个SVs,基于检测的SVs进行选择清除分析,在梅山猪中鉴定到64个新的受选择区域,其中的一些位点与繁殖性状相关;2021年发表在《Communication Biology》上的研究中,作者在27个不同的狗中鉴定了14,953,199个SNPs、6,958,645个indels和217,951个SVs(图2)。
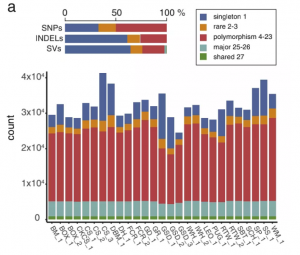 图2 27个狗中SNPs、indels和SVs的分布
图2 27个狗中SNPs、indels和SVs的分布
另外,拷贝数变异(CNV)作为基因组结构变异的重要来源,是造成哺乳动物性状差异的重要遗传变异类型,可作为SNP的代替分析标记用于GWAS研究,已用于多个包含牛、羊、猪和山羊的GWAS研究中。
(1)例如,在416个奶山羊中鉴定得到了7,208个CNVs,GWAS分析发现31个CNVs所在区域与产奶量等性状显著相关,并且发现在显著相关的CNVs中,注释的基因大多数在产奶相关的生物过程和健康相关性状中发挥着重要作用。在另一个研究中,利用新开发的GWAScore方法整合已发表的GWAS数据来鉴定潜在的候选位点,并对3个不同品种(CKA,n=10、HU,n=10、STHS,n=9)山羊进行全基因组CNVs检测(图3),通过将CNVs与GWAScore和QTL位点相整合,发现197个候选基因与CNVs存在重叠,进一步鉴定得到28个与生长性状相关的CNVs。
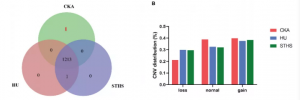 图3 3个不同品种山羊CNVs分析
图3 3个不同品种山羊CNVs分析
在牛的CNV变异研究中,利用已发表的1,116个牛中的鉴定的25,030个CNVs,基于不同性状(功能、健康、繁殖等)的CNVs进行GWAS,并评估其对养殖和育种实践的影响,结果显示,共有56个CNVs与8个表型性状中的一个或多个显著相关,其中12号染色体上的3个CNVs与产脂性状和乳腺性状的显著关联,这些CNVs位于23个不同的基因上,该研究是首次对如此大的瑞士奶牛样本进行基因组分析,将CNVs与繁殖、乳腺和健康性状联系起来。同样,基于在528个荷斯坦奶牛中鉴定的197个CNVs(图4),对473头牛10个重要生产性状进行GWAS分析,共检测到57个CNVs与至少一个性状显著相关(图5),对这些CNVs与候选基因的潜在重叠进行分析,预测得到一些表型相关的候选基因,该研究结果对荷斯坦奶牛的基因组鉴定了CNV,并提供了影响饲料效率和采食相关性状变异的候选基因。
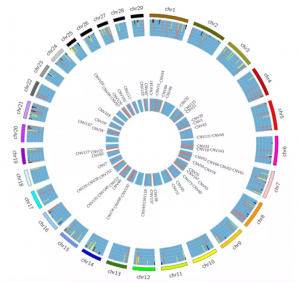
图4 荷斯坦奶牛CNVs在基因组上的分布
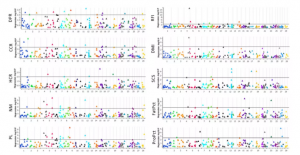
图5 10个重要性状的CNV-GWAS关联结果
SV和SV-GWAS在植物中的应用
在作物和拟南芥中也发现了大量的SVs,并将其用于GWAS研究。
(1)在水稻中,已经报道数篇SV进行GWAS的案例,其中2018年发表在《Nature》上的3000份水稻重测序研究中,对测序深度在20×以上的材料进行SV分析,确定了93K的SVs,其中有582个大于500kb的SVs,平均每个染色体有12,178个。籼稻和粳稻表现出了非常强烈的SV差异,每个籼稻与日本晴基因组有14K的SVs,是粳稻的3.5倍。SV系统发育树(基于453份材料)与SNP树相似(图6);在另一项研究中,基于8个GWAS研究队列,绘制了348个水稻QTNs(包括PAVs和CNVs)的图谱,还开发了基因组导航系统RiceNavi,用于QTN金字塔构建和育种路线优化,并应用于中国广泛栽培的籼稻品种黄花沾的改良。
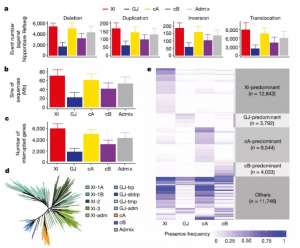 图6 453个高深度测序材料的SVs分析
图6 453个高深度测序材料的SVs分析
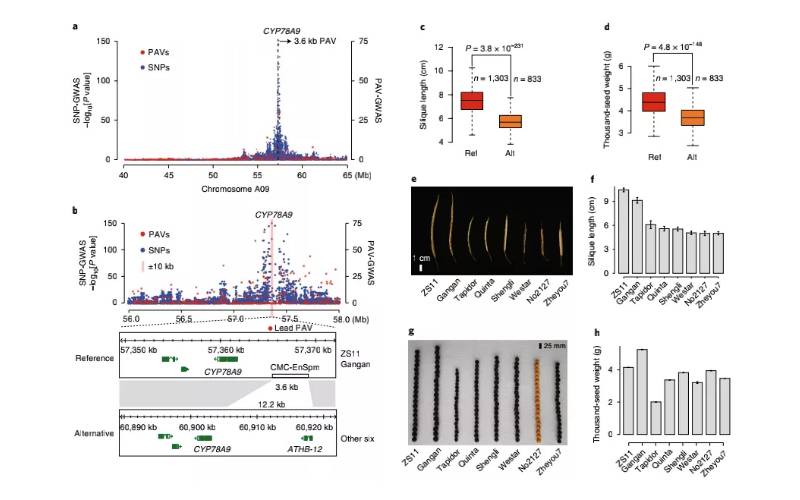
(2)在除水稻以外的作物中也有使用SVs进行GWAS的报道。例如,在玉米中,521个自交系玉米基因组序列中的SVs被用于绘制玉米SV图谱;在小麦中发现了影响染色体3B上基因和转座元件的SV;在最近的一项木瓜研究中(使用栽培木瓜和野生木瓜),共鉴定出8083个SVs,其中包括5260个缺失,552个串联重复和2271个插入,在该研究中,还发现CNVs与生长和对环境压力的反应有关;在油菜中,基于鉴定得到的数百万SVs和PAVs用于PAV-GWAS,直接鉴定到了荚果长度、种子重量和开花时间相关的结构变异,这些是之前基于单核苷酸变异SNP进行的GWAS所没能鉴定出来的,充分说明了PAV-GWAS可以互补于SNP-GWAS用来鉴定特定性状相关的位点(图7);另外,其他植物SVs鉴定例子包括:沟酸浆属的耐盐性、葡萄的浆果颜色和番茄的驯化性状等研究。
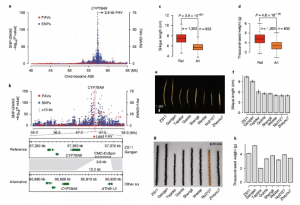 图7 油菜SNP-GWAS与PAV-GWAS
图7 油菜SNP-GWAS与PAV-GWAS
(3)在模式植物拟南芥(A thaliana)中,也有与许多重要性状相关的SVs。其中一个研究中,SVs与编码锌指转录因子的CONSTANS (CO)基因启动子相关。在另一项研究中,利用SVs(包括PAVs、重复、倒位)对来自非洲、欧洲、亚洲和北美的1301份自然种群进行GWAS研究(图8),与春化(冷)反应、开花和抗旱/耐热相关的3个基因之间也存在较强的相关性。
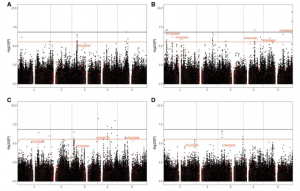 图8 基于PAV(红点)和SNP(黑点)的GWAS
图8 基于PAV(红点)和SNP(黑点)的GWAS
小结和展望
开发新的GWAS方法一直是一个活跃的研究领域,将SVs标记用于GWAS也是近期主要进展之一,基于上述的介绍,不难发现SVs已在人类、牲畜和植物的多项研究中被用于鉴定性状表型关联标记。随着测序技术和软件的不断发展,鉴定全基因组范围内的结构变异变得唾手可得,可满足基于动植物群体的SV-GWAS研究,在SNP-GWAS的基础上,实现对复杂数量性状更加深入的解析!
参考文献
[1] Gupta PK. GWAS for genetics of complex quantitative traits: Genome to pangenome and SNPs to SVs and k-mers. Bioessays. 2021 Nov;43(11):e2100109.
[2] Gupta PK. Quantitative genetics: pan-genomes, SVs, and k-mers for GWAS. Trends Genet. 2021 Oct;37(10):868-871.







 京公网安备 11011302003368号
京公网安备 11011302003368号