2021年5月,华中农业大学棉花遗传改良团队研究成果“Comparative genome analyses highlight transposon-mediated genome expansion and the evolutionary architecture of 3D genomic folding in cotton”发表于国际学术期刊Molecular Biology and Evolution(IF:16.241)。华中农业大学王茂军教授和李健英博士为论文共同第一作者,张献龙教授、中国农业科学院棉花研究所王坤波研究员和爱荷华州立大学Jonathan F. Wendel教授为共同通讯作者,百迈客也有幸参与其中基因组相关工作。
为了更好的分享热门科研进展与研究经历,助力科研解读基因,百迈客特邀李健英博士,于11月12日以直播的形式,为我们分享棉花转座子与基因组三维结构进化的奥秘(扫描图中二维码即可免费报名参与)。
文章详细解读
研究背景
转座因子(Transposable elements,TEs)在真核生物基因组结构的形成中起着重要作用。TE扩增和消除影响物种的表型变异、基因转录、基因组进化和群体多样性。最近的研究表明TEs也可能影响3D基因组结构,而在植物中,差异TE积累引起的基因组大小的显著变化对染色质高级结构进化的调节作用仍然未知。
棉属是棉族(Gossypium)里最大的属,含有大约52个种,共9种基因组类型,包括二倍体基因组A-G、K(2n = 2x= 26)和四倍体AD1-AD7(2n = 4x= 52)。其中最大的二倍体基因组属于K组(~2.6 Gb)与四倍体棉花基因组(~2.4 Gb)相似,约为最小二倍体棉种的三倍(D基因组;~840Mb)。因此,棉花是基因组大小进化与转座子扩增及染色质高级结构形成进化研究的重要资源。
材料与方法
材料:圆叶棉G. rotundifolium (K2) 、亚洲棉G. arboreum (A2)、雷蒙德氏棉G. raimondii (D5)新鲜叶片
测序:
基因组:K2、A2和D5分别 108×, 118×, 132×illumina以及124×, 131×, 167× Nanopore
Hi-C挂载:6碱基酶HindⅢ;K2、A2和D5分辨率分别为20kb、20kb、10kb
Hi-C互作:4碱基酶DpnⅡ;分辨率20 Kb, 50 Kb, 100 Kb.
研究结果
1. 圆叶棉、亚洲棉、雷蒙德氏棉基因组组装注释
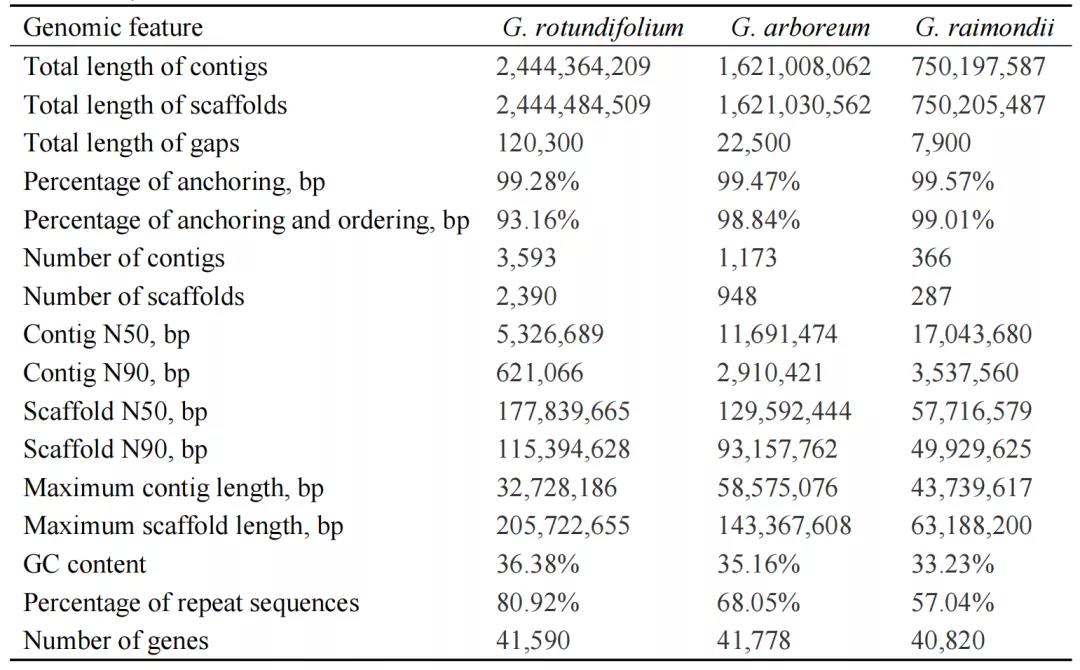
作者利用Nanopore测序技术组装了圆叶棉(K2)基因组,组装大小为2.44Gb(contigN50 = 5.33 Mb);并提升了亚洲棉(A2)和雷蒙德氏棉(D5)的基因组,组装大小分别为1.62 Gb (contigN50 = 11.69 Mb)和0.75 Gb(contigN50 =17.04 Mb)(表1)。Hi-C挂载率均超过99%,BUSCO结果分别为 92.5%, 93.9%,及95.4%。众多指标表明,本次组装的三个二倍体棉花品种基因组为参考级基因组水平。
表1 三种棉组装结果
通过从头预测、同源预测与转录预测结合的方式,在K2、A2和D5分别注释到41,590, 41,778与40,820个基因。与许多植物一样,三种棉中重复序列非常丰富,分别占到了每个基因组的57-81%,并且含量随基因组的增加而扩张(K2=1978 Mb、A2=1103 Mb和D5=428 Mb)(图1)。重复序列注释表明,棉种特异的反转录转座子(LTR)扩增是造成D5、K2和A2这三个棉种基因组大小呈现三倍差异的原因, 尤其是Gypsy和DIRS类型。
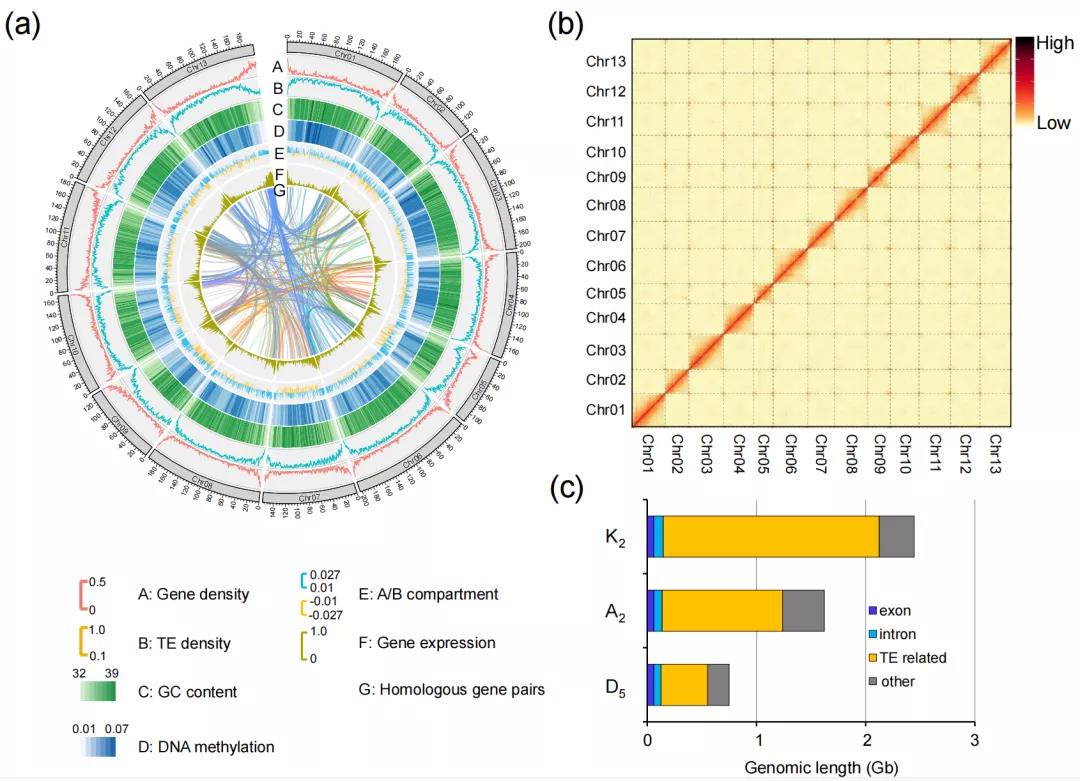
图1 圆叶棉基因组特征
在K2中,LTR逆转录转座子的插入时间峰值出现在4.5-5 MYA,A2在0.6-1 MYA有一个较新的扩增峰值,表明K2基因组中转座子插入最为古老,并在5 MYA左右获得了大量LTR,而A2基因组有更多新的转座子。系统发育树显示Clade III中Gypsy-like的逆转录转座子的巨大Gorge3扩张(图2)。
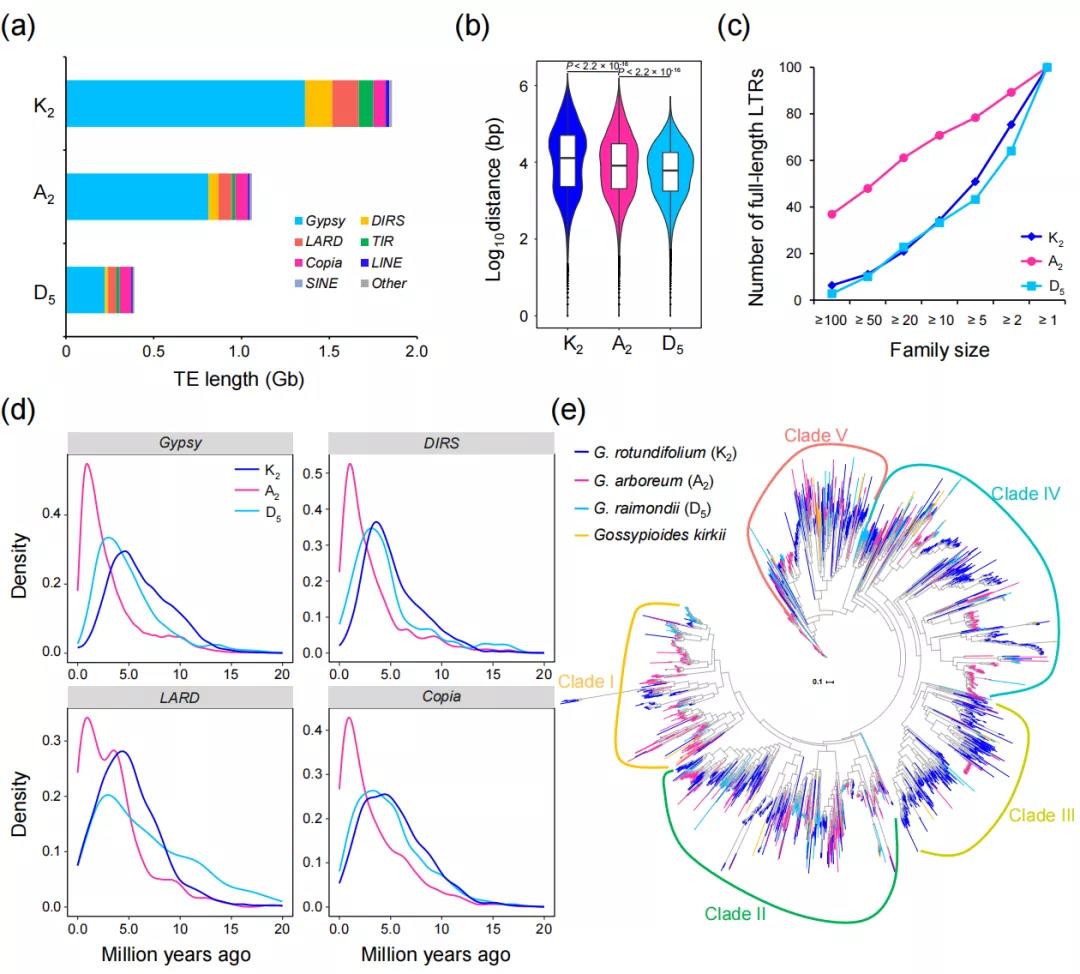
图2 三种棉中TE进化特征
2. 比较基因组学和进化
比较基因组分析表明,A2和K2基因组在Chr01与Chr02染色体间存在一个大的易位;K2和D5基因组在Chr13与Chr05染色体间存在一个大的易位。Ks分析与前期研究一致,结果表明,三个棉种在57-71百万年前存在一次共同的全基因组复制事件(图3),并在5.1-5.4 百万年前发生物种分化,K2、A2和D5基因组与它们最近的外类群约在8.5-10 百万年前发生了分化。由于这三个基因组来自同一祖先,作者探讨了物种形成后共线基因丢失和增加的程度。分析表明结果表明,K2和D5/A2之间的比较显示 K2缺失基因数量最多(D5和A2有5868个共线,而K2中没有).使用OrthoMCL进行的基因家族分析,发现三个棉中每个基因组大约有15%特异的基因家族。
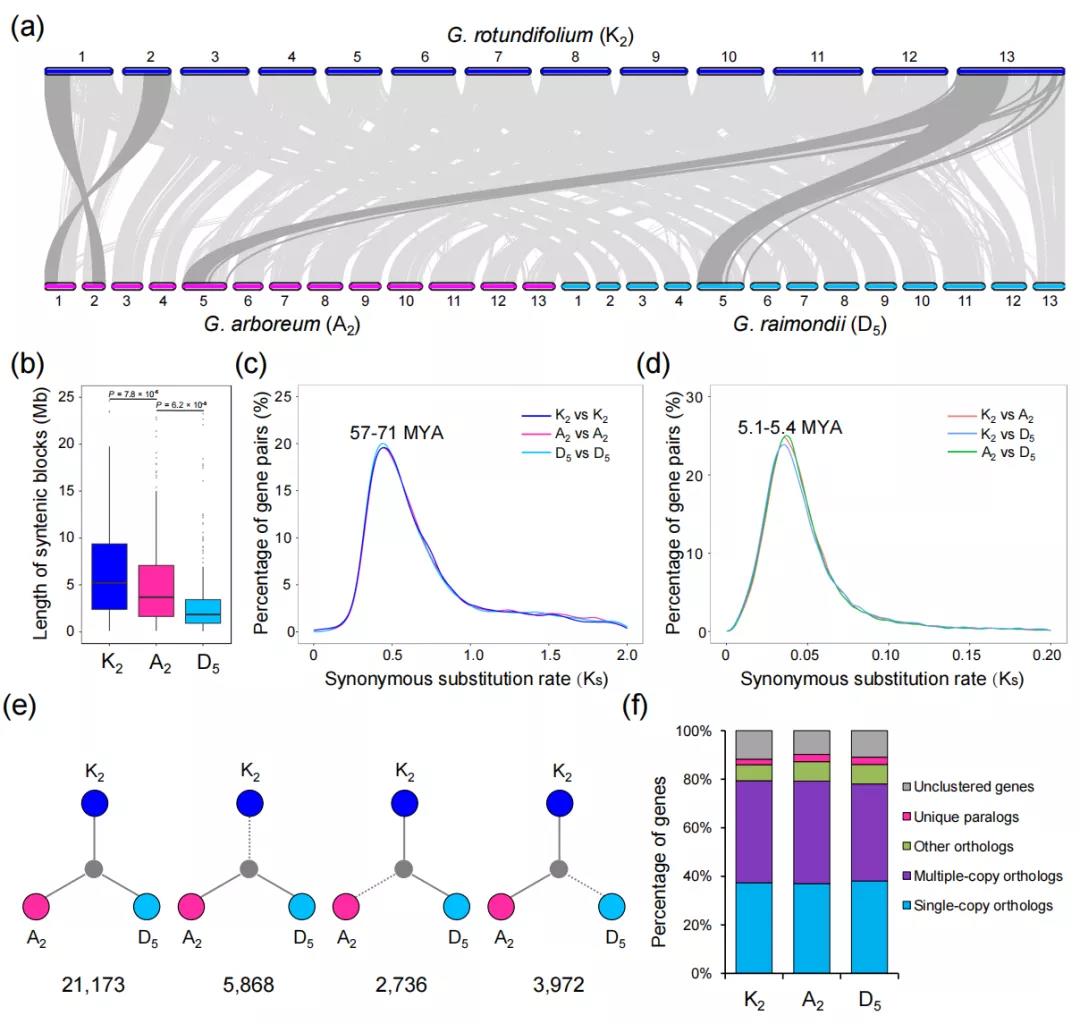
图3 基因组共线性与进化
3. A/B compartment演化
植物染色质被划分为“活性”(A)或“非活性”(B)区域,通常分别对应于常染色质和异染色质。HiC染色质互作分析发现,与基因组大小的差异一致,与A2或D5相比,K2基因组的活跃区域(约占基因组的44%)较少,而非活跃区域(55%)较多。较大的K2和A2基因组在A/B compartment之间表现出更多的插入,与这些较大基因组中发现的富含TE的区域相对应。A2/K2与D5相比多了约7000个基因,这与活跃的转座子扩增相关。对于较大的基因组,一个compartment中TEs的比例也稍大,表明这些较大基因组(K2和A2)所显示的A/B compartment变化模式导致更扩散的边界,并在A compartment中包含更多的基因和TEs。
K2与A2及与D5相比更多的倾向于A向B的转化。K2和A2中有更多的基因处于A compartment,D5中有更多的基因处于B compartment。功能富集分析结果表明,A-to-B基因在离子结合和转录因子活性途径中富集,而B-to-A基因参与基本活性,如泛素转移酶活性、果胶酸裂解酶活性和ATP结合。表达分析显示,表达的TEs可能参与了A-to-B区的转换,这与基因转录有关。
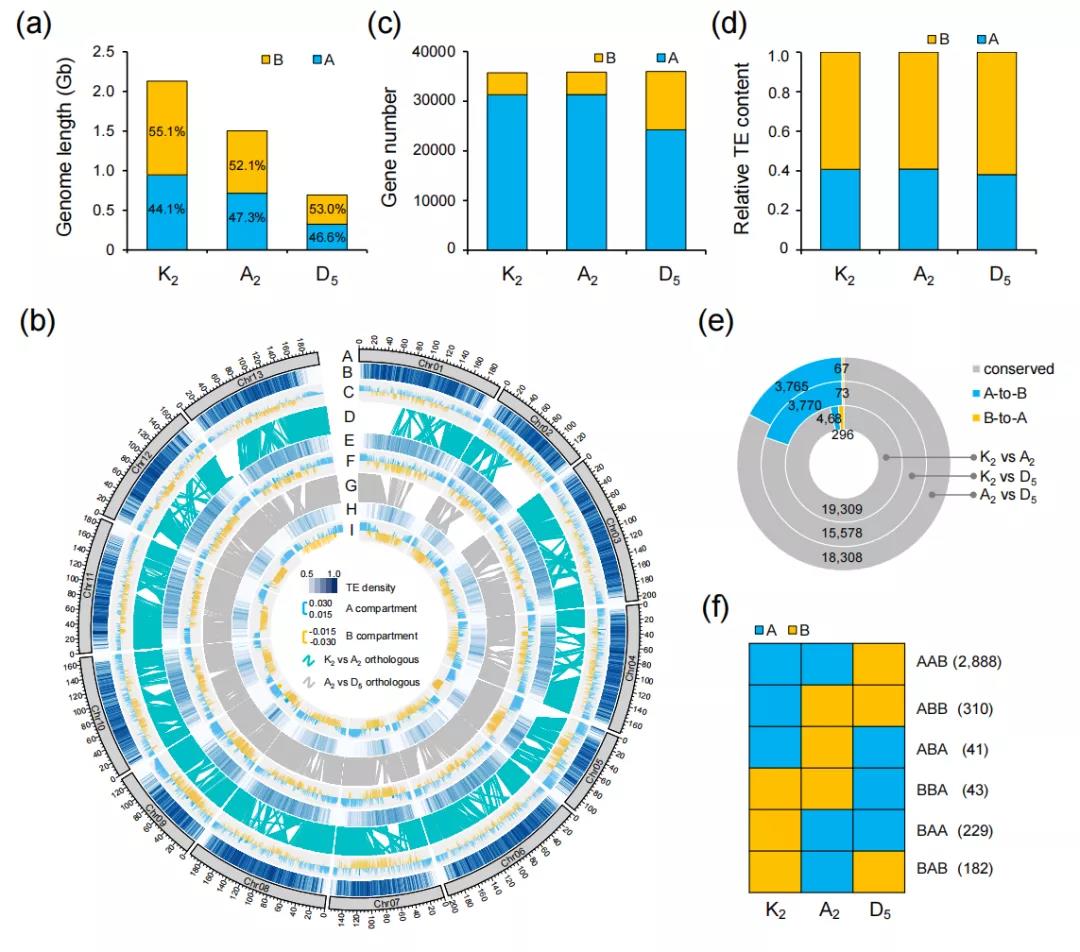 图4 三种棉中A/B compartment 特征
图4 三种棉中A/B compartment 特征
4. TAD结构演化及转座子扩增对TAD结构影响研究
拓扑结构域(TAD)是位于A/B compartment内的较小域,表现出频繁的域内相互作用,与位于域外的位点的相互作用频率较低。由于TAD边界的转换可以指示结构重组,作者比较了共线区中的TAD边界,以探索三个基因组中的TAD保护和转换。研究发现,在三个基因组中,K2中有406个TAD边界是保守的,并且随着TAD总数的增加,谱系特异性边界的数量增加。K2基因组在TAD边界的基因数最少,而D5的基因数最多(图5)。大约60%的拓扑结构域(TAD)在三个基因组中发生了重新组织,K2基因组中有更多特异的TAD。TAD边界处的motif分析显示69个特定于K2的motif,但A2和D5中分别只有8个和4个特定motif。
基于边界TE覆盖度,边界TE表达以及TE插入时间分析,发现K2不保守的TAD边界存在特异的和较新的转座子(物种分化后爆发的TE)插入。这些结果表明最近在K2和A2基因组中表达的TEs的扩增可能有助于在三个物种分化后形成谱系特异性TAD边界。基于这些结果,作者提出了三个棉种分化过程中,基因组扩张-转座子扩增介导的A/B compartment转换和TAD重组的进化模型(图6)。
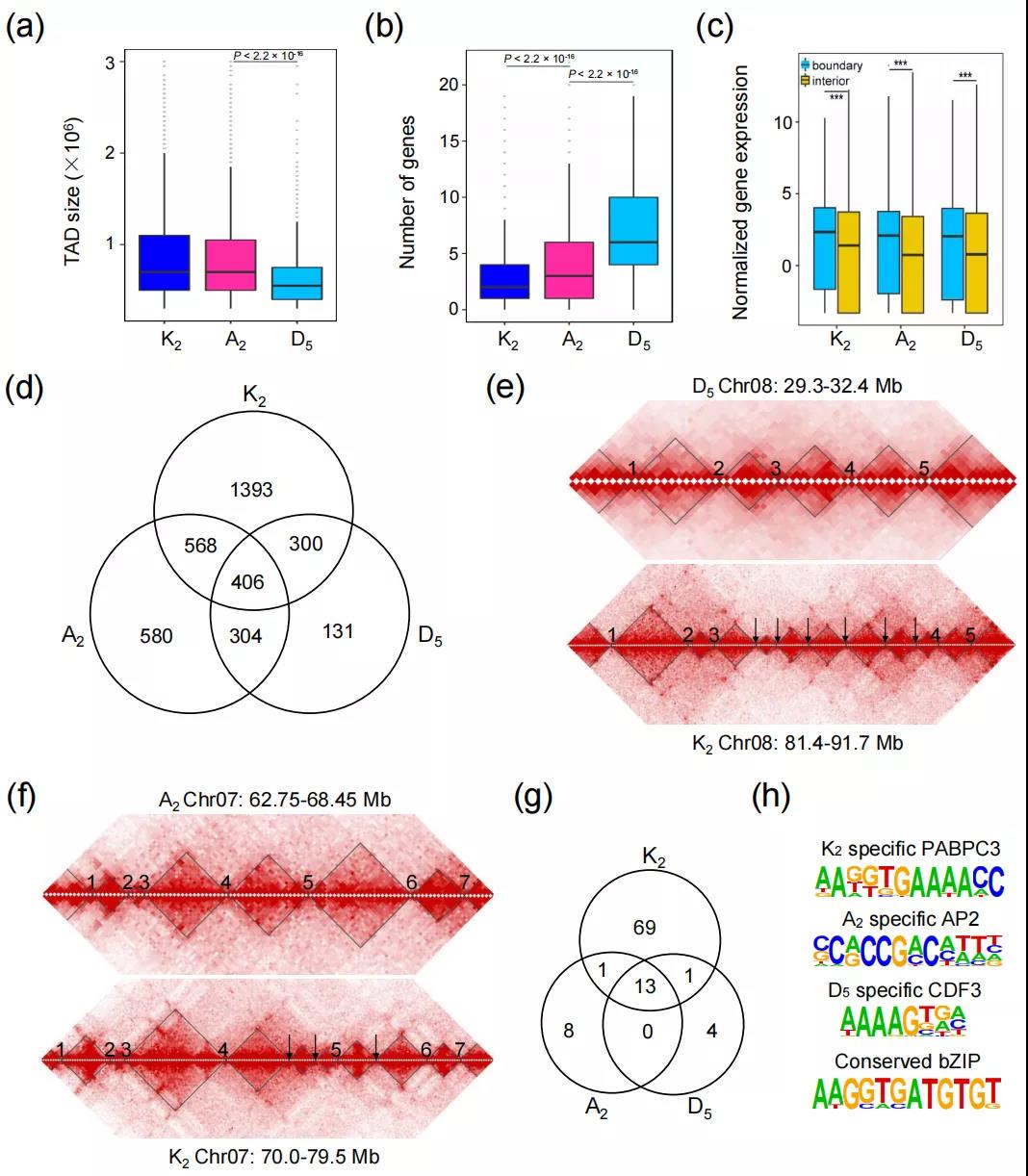
图5 特异和保守的TADs
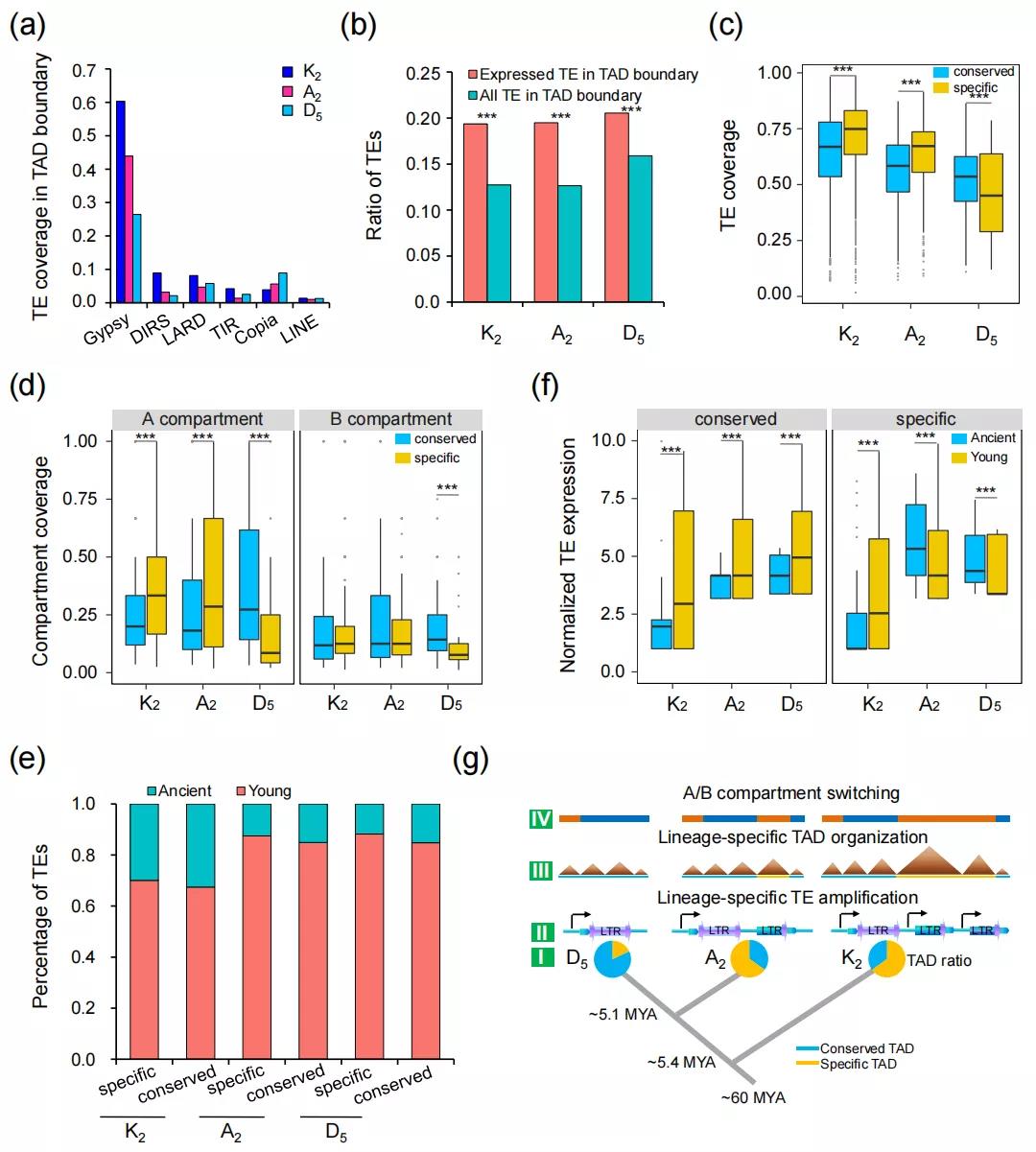
图 6 TE扩增对TAD边界重组的影响
小结
本次研究首次公布了棉属中二倍体圆叶棉基因组,并对亚洲棉和雷蒙德氏棉基因组进行了升级,解析了转座子活动驱动的基因组大小进化特征,从转座子扩增和染色质空间结构角度为棉花物种进化提供新的见解,为植物中转座子活动介导的转录调控进化研究提供参考。
原文链接:
https://academic.oup.com/mbe/article/38/9/3621/6262643









 京公网安备 11011302003368号
京公网安备 11011302003368号