核心种质是种质资源的一个核心子集,以最少数量的遗传资源最大限度地保存整个资源群体的遗传多样性。对种质资源进行核心种质鉴定不仅可以作为种质资源群体研究和利用的切入点,提高整个种质库的管理和利用水平,还能有重点地进行优异种质的研究,结合GWAS分析、遗传进化分析、QTL定位、特有标记开发等方法进一步进行基因的挖掘与克隆,提高种质资源的利用效率。本期精选发表的核心种质构建与应用文章,包含水稻、大麦、高粱、黄瓜、辣椒多种物种,包含GWAS、遗传进化等应用方向,提供核心种质鉴定应用的研究思路。
(1)大麦种质资源遗传多样性[1]
Genebank genomics highlights the diversity of a global barley collection

基因库储存来自世界各地的农作物品种、地方品种和野生近缘种的样本,以保护我们的农业遗产,并开发用于未来作物改良。位于Gatersleben的IPK研究所的德国联邦异地基因库是拥有世界上最全面的栽培植物收集品的地方之一,包括22,000多个大麦种子样本。本研究对种质库中22,626份库存大麦种质资源进行单株基因分型,得到了171,263个SNP位点,通过PCA和ADMIXTURE群体结构分析全球驯化大麦的种群结构,并检测到这些材料之间存在33%的重复比例。该研究大样本量的SNP标记为全基因组关联分析提供了强大的基础。进一步检测了大麦基因库的已报道和新的潜在形态位点,找到了大麦和水稻中无倒刺芒趋同选择的证据,同时在此基础上构建了基因型数据和目录性状数据的整合平台,并依托种质资源目录性状数据实现了质量性状新基因发掘。本研究不仅为各类库存农作物种质资源的高效保藏和多样性种质的国际交流提供了借鉴,也为开展基于基因型的大样本核心种质构建、精准鉴定和深度研究提供了思路。
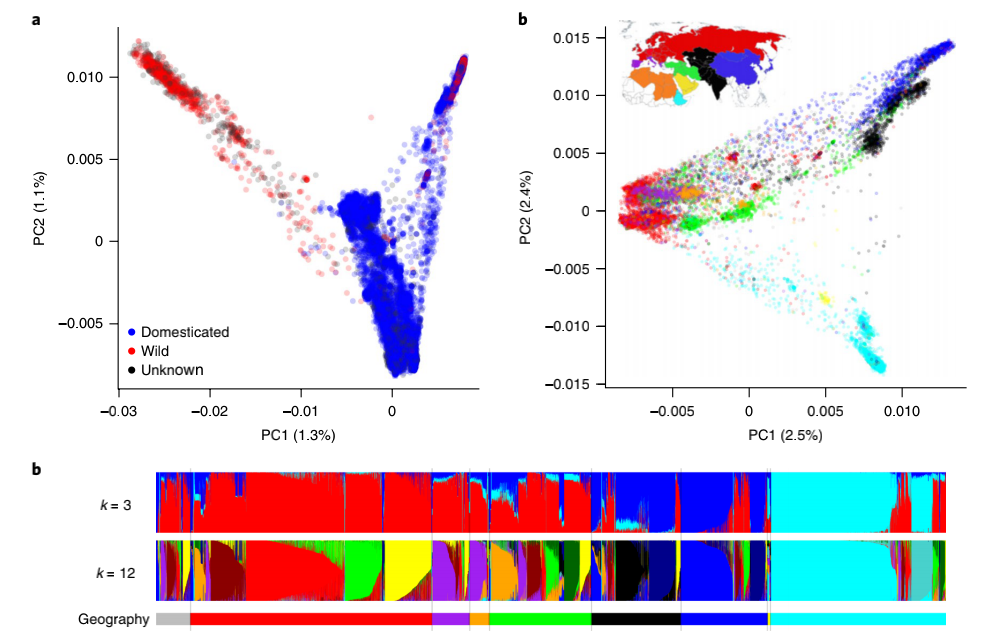
全球Genebank中大麦遗传多样性
(2)辣椒种质全球范围扩张史[2]
Global range expansion history of pepper (Capsicum spp.) revealed by over 10,000 genebank accessions

基因库收集和保存大量植物和详细的护照信息,目的是保护遗传多样性以进行保护和育种。此类集合的遗传表征有可能阐明重要作物的遗传历史,使用标记-性状关联来鉴定感兴趣性状的位点,寻找正在进行选择的位点,并通过识别分类错配和重复为基因库管理作出贡献。本研究通过GBS测序对来自全球基因库的10,038个辣椒 (Capsicum spp.) 种质进行基因分型,并调查了这种标志性主食的近期历史。基于SNP数据计算了identity-by-state (IBS) 比例,在基因库内和基因库之间检测到多达 1,618 个重复种质。进一步深入分析了常见食用辣椒的遗传多样性以调查其历史,发现在全球广泛地区收集的辣椒种类有很大的重叠。GWAS分析和选择清除分析发现辣椒性状,如刺激性,这些性状在全球分布不均匀,表明人类偏好对驯化辣椒的遗传结构产生了主要影响。
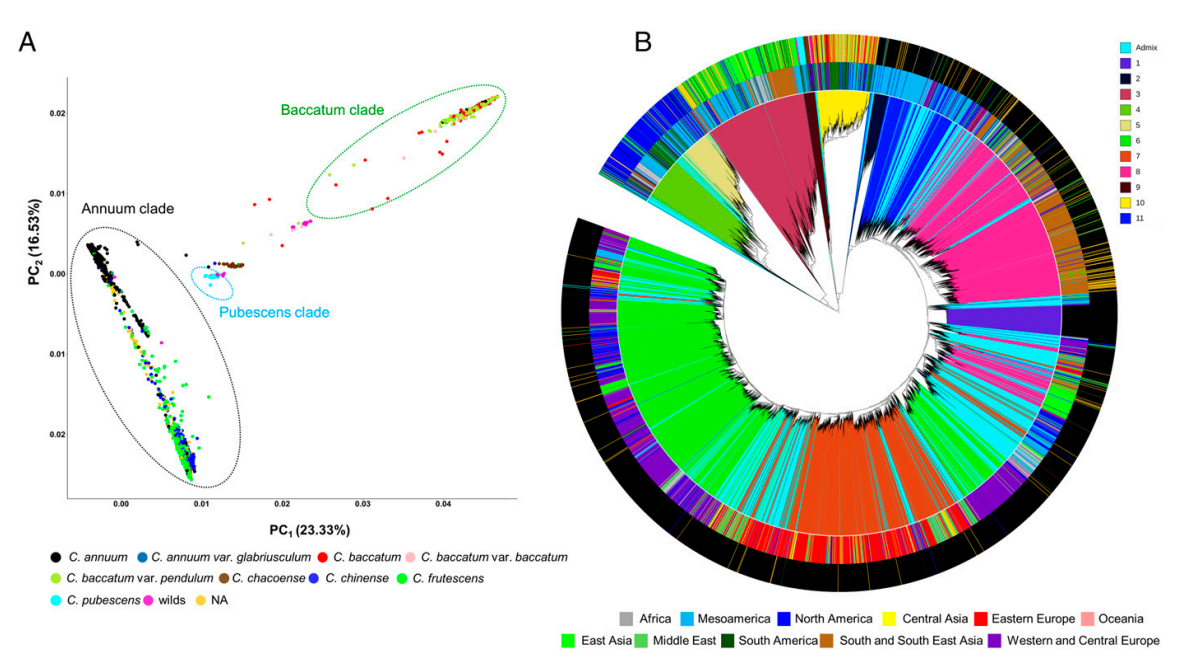
全球Genebanks中的辣椒多样性
(3)3004份水稻核心种质开发[3]
Designing a Mini-Core Collection Effectively Representing 3004 Diverse Rice Accessions

遗传多样性为植物育种和遗传研究奠定了基础。3K水稻基因组 (3KRG) 项目对3000多个水稻基因组进行了测序。本研究新增了四个印度水稻品种以创建一个包含3004个水稻品种的群体。但是如此多的种质资源难以保存和评估。构建核心和微核心种质是遗传资源管理的有效方法。因此,本研究开发了一个包含 520 个种质的微核心种质集,它捕获了大部分SNP并代表了原始种质资源群体中的所有表型和地理区域。微核心种质集使用不同的统计分析进行了验证,并包含来自所有主要水稻的代表,包括粳稻、籼稻、aus/boro 和印度香米等。微核心种质集的全基因组关联分析有效地复现了原始群体中确定的标记-性状关联。单倍型分析验证了微核心种质集的实用性。本研究中开发的水稻微核心种质集对于农艺性状评估和通过标记辅助分子育种进行的水稻改良具有重要价值。
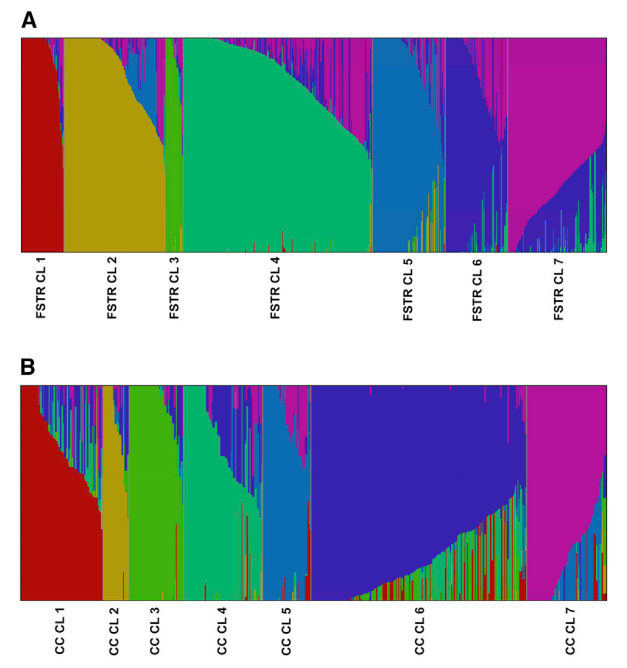
水稻原始种质资源和微核心种质集的种群结构分析
(4)黄瓜遗传多样性、种群结构、GWAS和核心种质开发[4]
The USDA cucumber (Cucumis sativus L.) collection: genetic diversity, population structure, genome-wide association studies, and core collection development

种质资源收集与保存是保护自然遗传多样性的重要资源,同时也是提供优异性状来源的重要资源。本研究使用GBS技术对美国农业部种质资源库保存的1234个黄瓜种质 (Cucumis sativus L.)进行基因型鉴定。得到了超过23 K的SNP位点,利用这些SNP标记对黄瓜种质的遗传多样性、种群结构、系统发育关系、连锁不平衡和种群分化进行分析,发现黄瓜主要分为三个亚群,分别是黄瓜起源地南亚亚群、东亚亚群和其他地区的品种形成的亚群,并且不同亚群之间的连锁不平衡指数(LD)、核苷酸多样性(π)以及群体分化指数(Fst)都存在明显的差异。还通过全基因组关联研究 (GWAS) 确定了与 13 个性状相关的SNP位点。最后,开发了一个包含 395 种材料的核心种质集,代表了96%的遗传变异。这些核心种质资源对黄瓜优异基因资源的利用和黄瓜基因组的深度解析提供了数据支持。
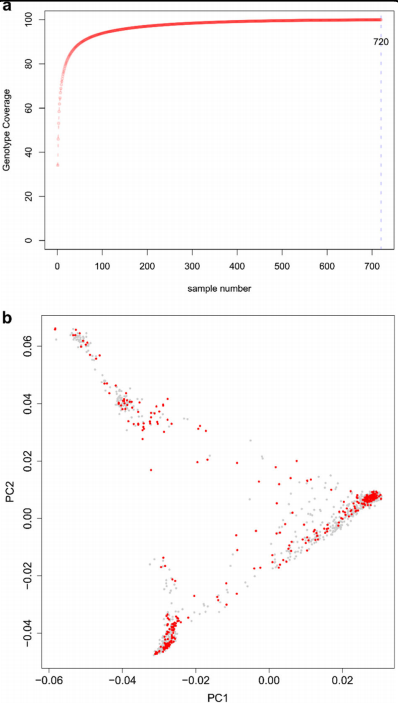
黄瓜核心种质开发与评估
(5)埃塞俄比亚高粱种质鉴定[5]
A comprehensive phenotypic and genomic characterization of Ethiopian sorghum germplasm defines core collection and reveals rich genetic potential in adaptive traits

了解作物的种群遗传结构和多样性对于设计植物育种的选择策略至关重要。本研究对大约2010份埃塞俄比亚高粱种质在多个地点进行了不同性状的表型鉴定,并且对其中1628个种质,主要是地方品种、一些改良品种和近交系,进行了基因分型。表型数据显示,埃塞俄比亚高粱种质中重要性状与不同的农业气候地区相关,遗传多样性高,且存在罕见的自然变异。随后的基因型分析确定了每个高粱品种的最佳亚群数、不同的聚类群和祖先。为提高种质资源的利用率,在聚类分析的基础上,通过基因型的后验分组和数量性状的分层随机采样,筛选出387个核心种质集。进一步对筛选出的核心种质集进行了评估。此外,通过基因组-环境关联分析,确定了与非生物因子适应相关的候选基因,这对开发不同环境的适应潜力具有重要意义。本研究描述了高粱收集的多样性和关系、已开发核心的代表性,并为与非生物胁迫耐受性相关的候选基因提供了新的见解。
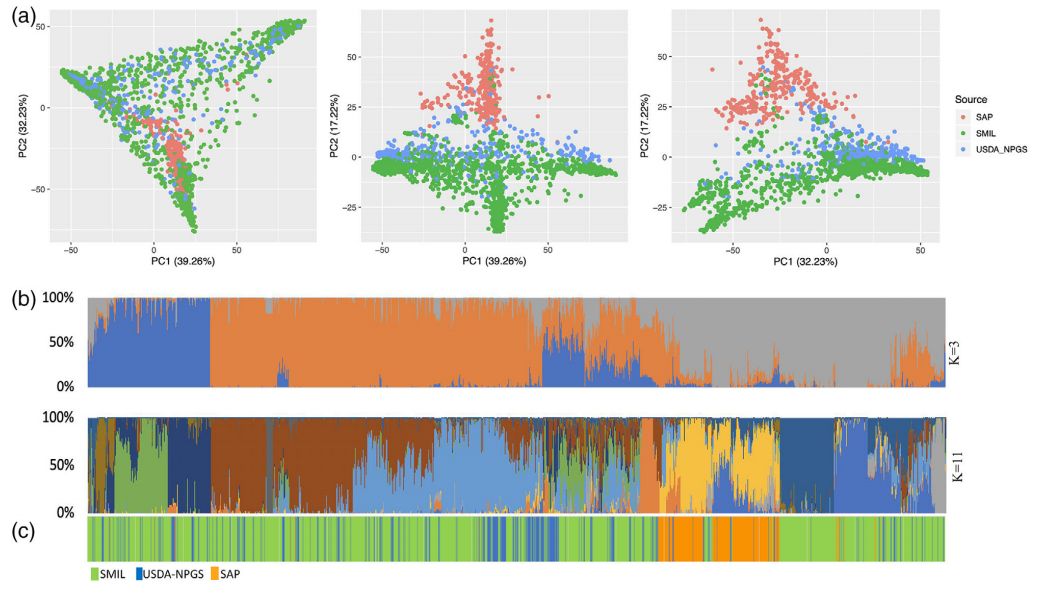
高粱种质遗传多样性
百迈客助力种质资源精准鉴定
基于以上文献,我们不难发现,随着高通量测序技术的不断发展,基于基因型鉴定来筛选核心种质,利用高分辨率的分子标记类型,是提高遗传材料内及材料间遗传相似度和杂合度的鉴别的有效手段,更加有利于核心种质的构建。百迈客致力于将好的技术提供给科研人员,为推动种质资源精准鉴定,可提供大样本量种质资源全基因组重测序与SLAF简化基因组测序服务,进一步可进行核心种质鉴定、DNA指纹图谱构建等分析,详情可联系当地销售经理/技术支持。
参考文献
[1] Milner SG, Jost M, Taketa S, et al. Genebank genomics highlights the diversity of a global barley collection. Nat Genet. 2019;51(2):319-326.
[2] Tripodi P, Rabanus-Wallace MT, Barchi L, et al. Global range expansion history of pepper (Capsicum spp.) revealed by over 10,000 genebank accessions. Proc Natl Acad Sci U S A. 2021;118(34):e2104315118.
[3] Kumar A, Kumar S, Singh KBM, et al. Designing a Mini-Core Collection Effectively Representing 3004 Diverse Rice Accessions. Plant Commun. 2020;1(5):100049.
[4] Wang X, Bao K, Reddy UK, et al. The USDA cucumber (Cucumis sativus L.) collection: genetic diversity, population structure, genome-wide association studies, and core collection development. Hortic Res. 2018;5:64.
[5] Girma G, Nida H, Tirfessa A, et al. A comprehensive phenotypic and genomic characterization of Ethiopian sorghum germplasm defines core collection and reveals rich genetic potential in adaptive traits. Plant Genome. 2020;13(3):e20055.






 京公网安备 11011302003368号
京公网安备 11011302003368号