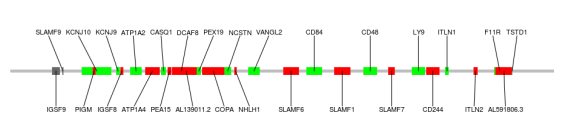
PPT、PS等好多工具可以帮助你实现这样的图,但是如果批量画呢?可能手都废了吧,还是上代码吧。
首先用你能用的办法将目标区域(1:160000000-161000000)内的基因、目标区域上游的两个基因、目标区域下游的一个基因的位置都提取出来,然后为每个基因赋一个颜色。中间灰色的bar两端的突出可以从第一个基因的开始位置向上游扩100K、最后一个基因的结束位置向下游扩100K。整理成作图用的输入文件,如下:
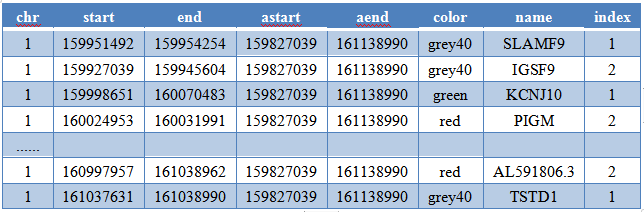
第1-3列是作图基因的位置,第7列是基因的名字。
第4-5列是整个灰色bar的位置(经过上下游扩完以后的)。
第6列是基因的颜色,目标区域上下游的基因用灰色表示,目标区域内的基因用你想要的颜色表示。
为了方便调整基因的symbol不重叠,增加第8列,1和2表示基因名字在图的上部和下部。
用ggplot2作图很方便,每个bar的可以通过画矩形框填充不同的颜色实现。
将输入文件读进来
library(ggplot2)
library()
data<-read.table(file=”Region.xls”,header=T,sep="\t",stringsAsFactors=F)
计算基因最中间的位置方便标基因label
data$middle<-(data$end - data$start)/2+data$start
color<data$color
geom_rect函数先画灰色的bar,再画基因,用geom_text_repel函数标基因的label
p<-ggplot(data)+geom_rect(aes(xmin=astart,xmax=aend,ymin=-0.01,ymax=0.01),colour="grey",fill="grey",size=0.1)+geom_rect(aes(xmin=start, xmax=end,ymin=-0.03,ymax=0.03),colour="grey", fill=color,size=0.1)+geom_text_repel(aes(x=middle,y=rep(0,length(color)),label=name),min.segment.length = 1,nudge_y = ifelse(data$index==1,0.3,-0.3))
p<-p+ylim(c(-1,1))
p<-p+theme_classic() +theme(axis.line = element_blank(),axis.text = element_blank(),axis.ticks = element_blank(),axis.title = element_blank())
print(p)







 京公网安备 11011302003368号
京公网安备 11011302003368号