随着分子生物学的兴起和向各方面的渗透,生物科学的各分支学科也经历着兴衰更替的变化。从目前的发展状况来看,在基因组研究中,利用常规研究技术及单一组学研究方法在冲击文章的过程中,对于技术的新颖性及物种生物学问题挖掘的深入性还是有所欠缺的,因此在组学整合的前提下,可尝试引用*新分子生物学研究技术,以为冲击高分文章“添砖加瓦”。如下小编就通过一些文章案例为大家进一步介绍下在破译某物种基因组密码的基础上,怎样利用新兴生物学研究技术,如基因编辑,单细胞测序技术及表观遗传学研究等对某物种生物学性状进行深度挖掘。
01 基因编辑(CRISPR/Cas9)
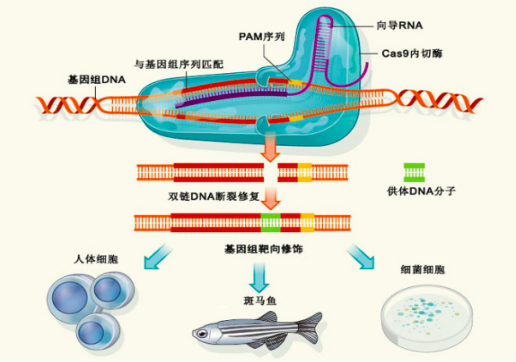
基因编辑(gene editing),又称基因组编辑(genome editing)或基因组工程(genome engineering),是一种新兴的相对√确的对某生物体基因组特定目标基因进行修饰的一种基因工程技术。其中CRISPR-Cas9基因编辑技术,则是对靶向基因进行特定DNA修饰的技术,也是用于基因编辑中前沿的方法。以CRISPR-Cas9基础的基因编辑技术在一系列基因治疗的应用领域都展现出极大的应用前景,而在动植物研究中也初露锋芒。
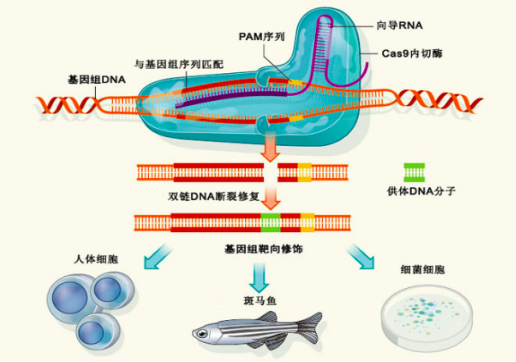
CRISPR-Cas9基因编辑技术原理图
技术应用:基因组De novo(PacBio CCS+ONT+ALLHiC)+ CRISPR-Cas9基因编辑

文中在对同源四倍体紫花苜蓿(Medicago sativa L.)基因研究中,首先利用了70 GB,~22x PacBio CCS数据进行基因组组装,组装获得紫花苜蓿基因组大小3154 Mb,Contig N50=459 kb, 然后利用ALLHiC进行同源染色体组群的划分,最后通过Hi-C互作热图、遗传图谱共线性、ONT数据回比、BUSCO完整性、转录组对基因组完整性等进行评估。同源四倍体紫花苜蓿基因组的破译对这一重要牧草后续的分子生物学研究具有重要意义。
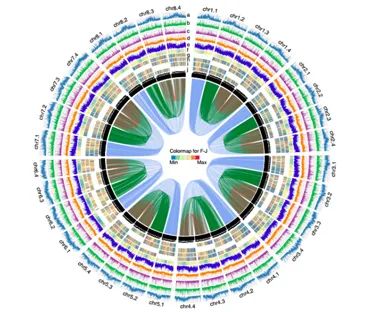
图1 紫花苜蓿基因组circos图
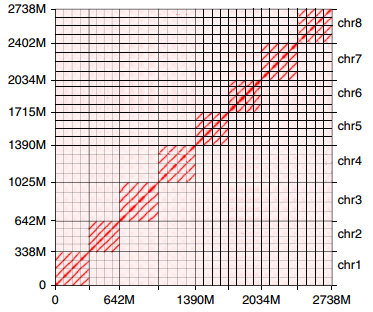
图2 紫花苜蓿Hi-C互作热图
进一步利用CRISPR/Cas9基因编辑系统对紫花苜蓿(Medicago sativa L.)中PDS基因(八氢番茄红素合酶基因)和PALM1基因(编码Cys(2)His(2) zinc finger transcription factor锌指转录因子;在二倍体蒺藜苜蓿M. truncatula中,其对复合叶形态发生具有重要作用)进行改造,进一步确定不同单倍型变异对紫花苜蓿白化和矮化及叶复合形态表型性状的影响。
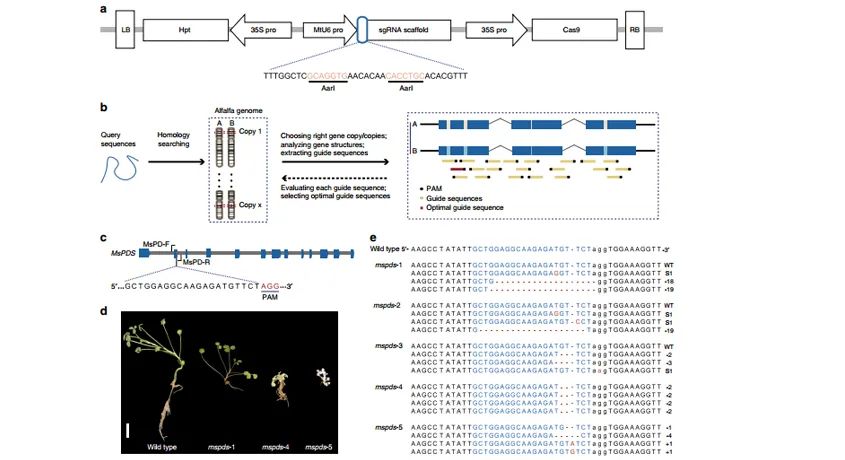
图3 CRISPR/Cas9介导的紫花苜蓿基因编辑研究
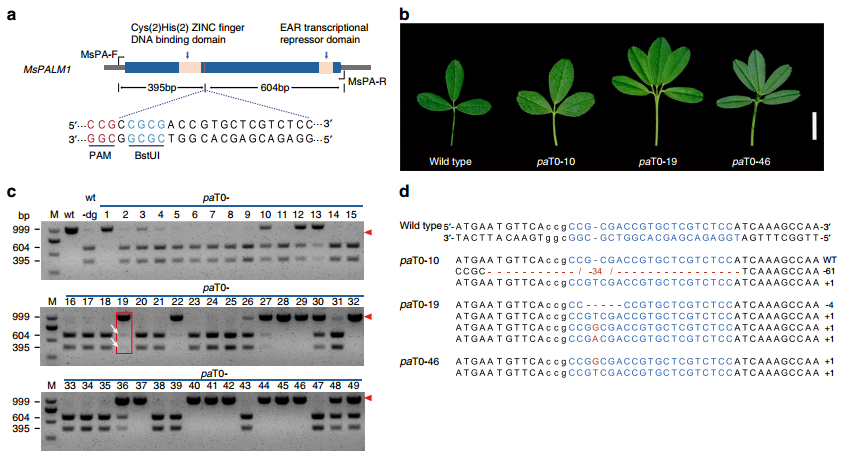
图4 紫花苜蓿MsPALM1的基因编辑研究
文章中利用了基因组三代测序技术,获得染色体级别的紫花苜蓿基因组的同时,攻克了同源多倍体物种基因组组装难题,组装获得了4个单倍型紫花苜蓿基因组,为后续基因编辑研究做了重要的铺垫,进而通过新技术CRISPR/Cas9基因编辑对控制相应表型性状基因进行改造,对于紫花苜蓿后续分子生物学研究具有重要而深远的意义。
02单细胞测序技术(10x Genomics)
单细胞测序,简而言之,就是获取单个细胞遗传信息的测序技术,其中10 X Genomics单细胞转录组测序平台具有广泛的应用。该技术利用微流控、油滴包裹和barcode标记等技术来实现高通量的细胞捕获技术,能够一次性分离、并标记500–10000个单细胞,从而获得每个细胞的3’端的转录组信息。主要用于细胞分型和标记因子的鉴定,从而实现对细胞群体的划分与细胞群体间基因表达差异的检测,此外该技术还可以预测细胞分化与研究发育轨迹,在当下疾病、免疫、肿瘤领域以及组织、器官、动植物发育研究中发挥越来越重要的作用。
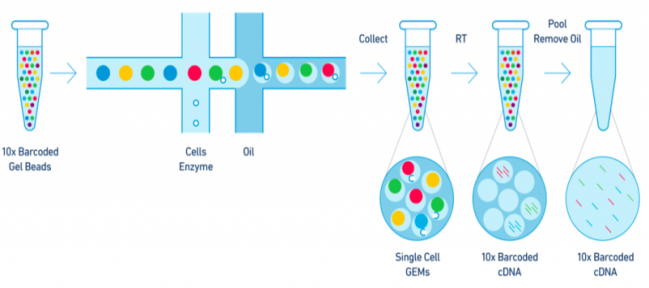
10x Genomics技术工作原理图
技术应用:基因组De novo(PacBio +Hi-C)+10xGenomics单细胞转录组测序

文中首先利用三代利用~80×PacBio RS+~100×Hi-C辅助组装等策略测序并拼装了准染色体水平的高质量安德爱胜蚓基因组,组装获得基因组大小1.3 Gb(Scaffold N50≈111Mb),基因组完整性92.1%(BUSCO),鉴定了31,817个蛋白编码基因,同时发现,蚯蚓基因组中重复序列LINE2转座元件可能在蚯蚓再生中扮演重要调控角色。
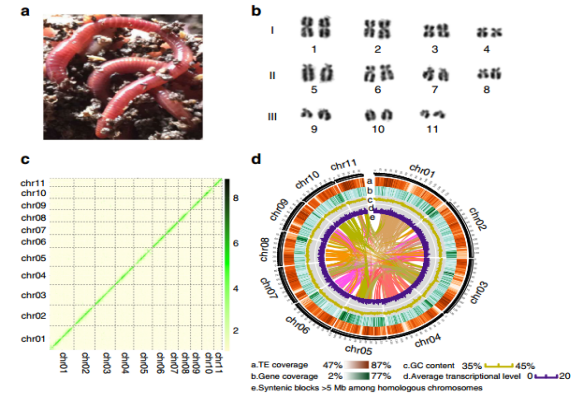
图 5 蚯蚓基因组组装
进一步利用10xGenomics单细胞转录组测序深度解析蚯蚓再生机制,蚯蚓再生早期72小时后损伤愈合部位细胞的高比例组分是干细胞,且多能干细胞在蚯蚓再生早期过程中具有重要作用。此项研究提供了一些蚯蚓再生的候选分子细胞学机制。
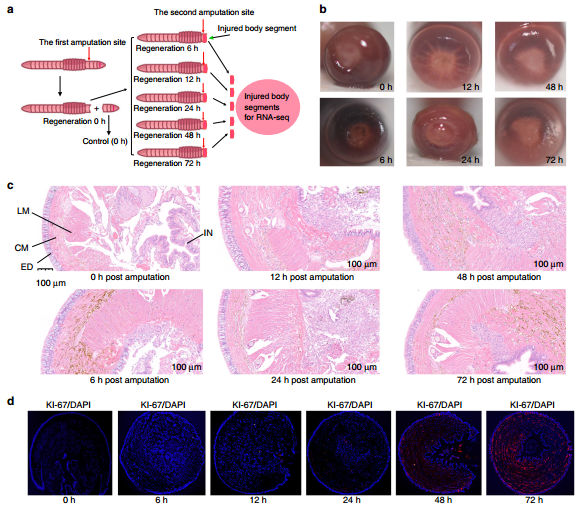
图6 蚯蚓再生过程表型组分析
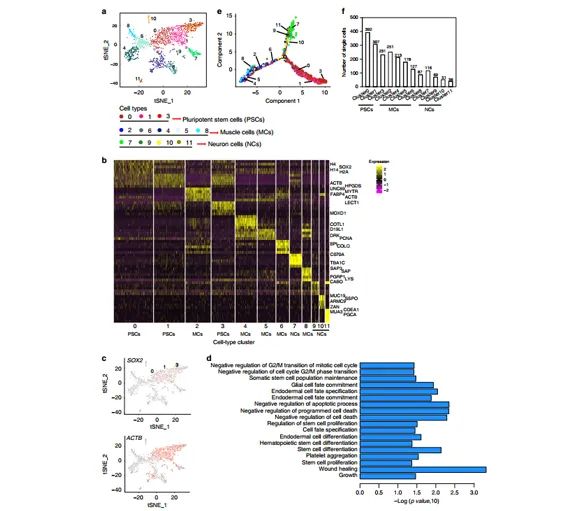
图7 蚯蚓再生过程单细胞转录组研究
文章中利用了三代测序技术及Hi-C辅助基因组组装技术,获得染色体级别的蚯蚓基因组,基因组组装连续性一般,但结合基因组序列信息及单细胞转录组研究解析了土壤生态系统重要成员无脊椎动物蚯蚓再生的分子细胞学机制,提出蚯蚓能够作为研究再生生物学或者再生医学的一个新的模型。
03表观遗传学研究
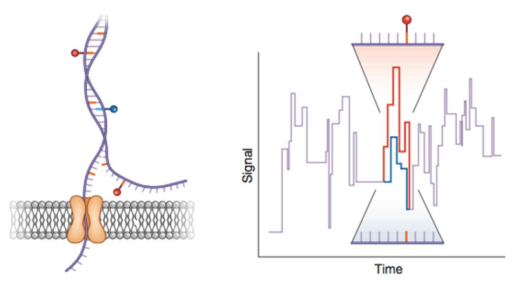
三代Nanopore DNA甲基化检测原理(Michael C Schatz,et al. 2018.)

PacBio对粳稻Nip与籼稻93-11基因组更新:利用PacBio对Nip和93-11进行测序及Illumina平台进行gap填充和误差校正后,Nip基因组大小为380.7 Mb(contig N50=16.97 Mb),93-11基因组大小为396 Mb(contig N50=9.64 Mb);进一步通过SMRT(单分子实时测序系统Single MoleculeReal Time)测序数据,在Nip和93-11中分别检测出704875个和784912个6mA位点。
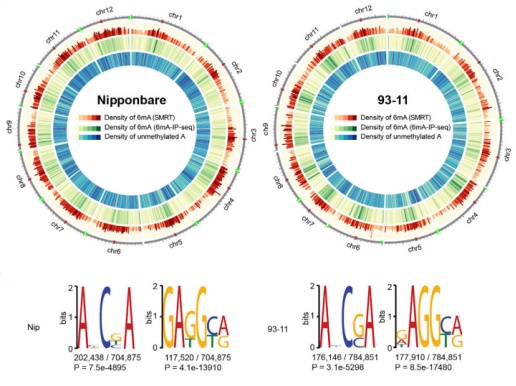
图8 水稻中6mA甲基化分析
文章中进一步通过RNA-seq分析表明在Nip和93-11中,在6mA位点有大量的高表达基因甲基化,大量未甲基化基因在低水平表达。6mA甲基化基因的表达水平显著高于非6mA基因;Nip对冷和盐胁迫表现出更大的耐受性,而93-11对热胁迫表现出更大的耐受性。6mA水平的变化在Nip和93-11之间表现出显著的差异。水稻6mA含量与耐冷性呈负相关,与耐盐性和耐热性呈正相关。






 京公网安备 11011302003368号
京公网安备 11011302003368号