The Architecture of SARS-CoV-2 Transcriptome SARS-CoV-2的转录组结构
发表期刊:Cell
发表时间:2020年5月
IF:38.637

研究背景
COVID-19是由一种被称为严重急性呼吸综合征冠状病毒2(SARS-CoV-2)的乙型冠状病毒引起的。SARS-CoV-2与SARS-CoV、中东呼吸综合征冠状病毒(MERS-CoV),分别具有80%和50%的同源性,这些病毒在人群中的爆发表明冠状病毒具有跨越物种壁垒并在人与人之间传播的非凡能力。
这篇文章结合了两种测序方法,Direct RNA sequencing (DRS)和sequencing-by-synthesis (SBS),明确地绘制了SARS-CoV-2的亚基因组RNA(subgenomic RNAs),ORF和转录调控序列(TRS)。同时发现了许多与常规TRS介导的聚合酶跳跃不同的非常规RNA融合事件。发现了RNA修饰位点,并测量了gRNA和sgRNA的poly(A)的长度。
研究方法
1.实验材料:
2.实验方法:
研究结果
1. SARS-CoV-2的转录组结构
用MinION纳米孔测序仪进行了DRS测序,获得了879,679个reads(1.9 Gb)(图1A)。大部分(65.4%)的reads定位于SARS-CoV-2,表明病毒转录本占据主导地位,而宿主基因的表达被严重抑制。已知DRS无法对末端12 nt进行测序,因此序列的5’末端缺失了12 nt(图1B)。病毒基因组3’端的覆盖度明显高于5’端,这也反映了RNA直接测序从RNA3’端开始测序的特点。病毒RNA中前导序列(72nt)的普遍存在导致在5’末端出现一个显著的峰。在sgRNA的leader-body junction位点检测到覆盖度的明显下降。此外,观察到反映出非常规的“拼接”事件的reads,这样的融合转录本导致5’末端覆盖度的增加(图1B,插图)。
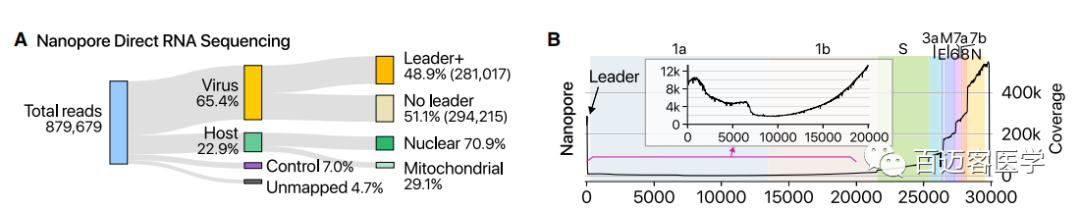
图1 Direct RNA seq数据分析图
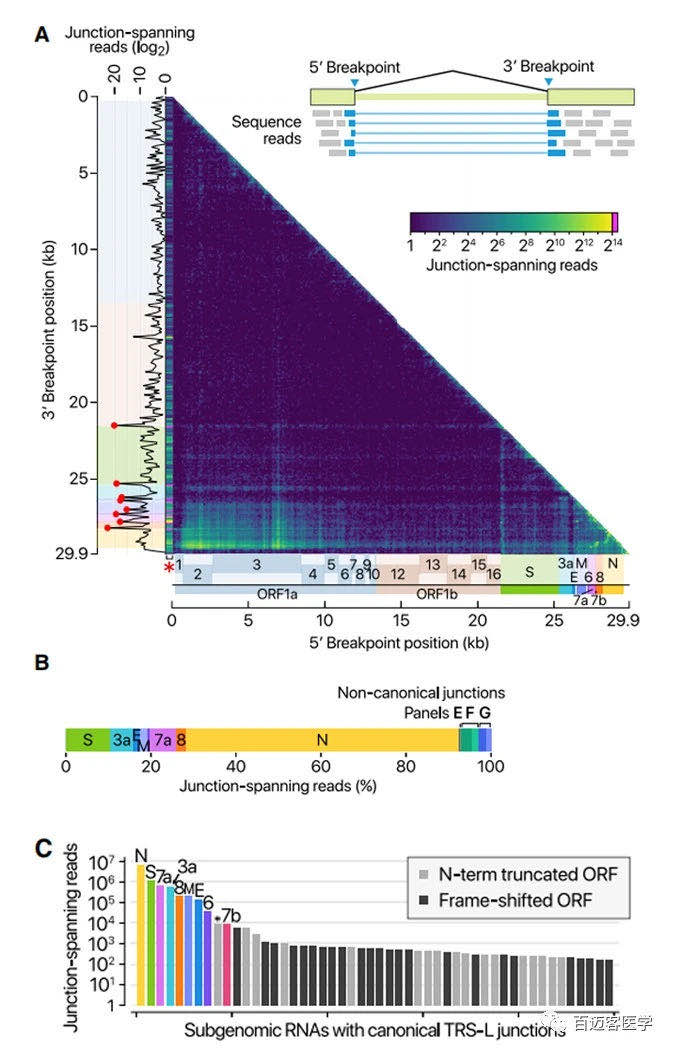
用DNB-seq确认和检查CoV基因组的junctions。在junctions处绘制了5’和3’断点,并通过计算跨越junctions的reads来估算融合频率(图2A)。如所预期的,leader代表了最突出的5’ 位点(图2A,x轴上的红色星号)。TRS-B被检测为3’位点的顶端(图2A,y轴上的红点)。这些结果证实,SARS CoV-2使用标准的TRS介导的模板转换机制进行不连续转录,以产生主要的sgRNA(图2B)。跨越junctions reads的定量比较显示,N RNA是表达最丰富的转录本,其次是S,7a,3a,8,M,E,6和7b(图2C)。
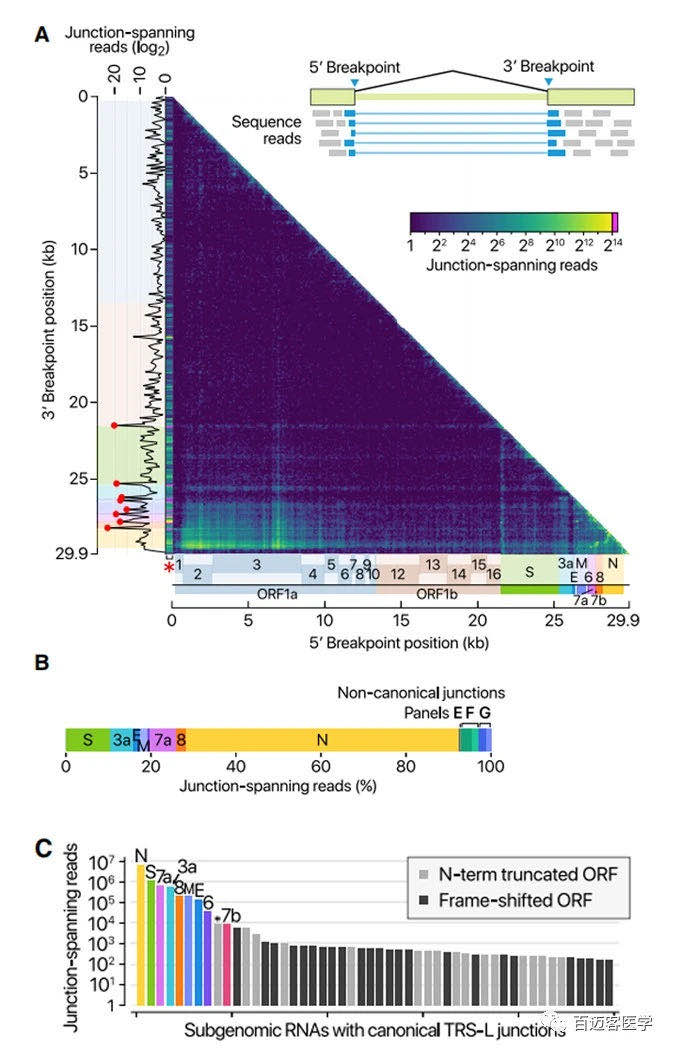
图2 病毒亚基因组RNA及其重组位点图
ORF10仅由DNB数据中的一个reads表示(占病毒junction-spanning reads的0.000009%),并且DRS数据完全不支持它。ORF10与已知蛋白没有显著同源性。因此,ORF10可能不被表达。总而言之,SARS-CoV-2表达了9种规范性sgRNA(S,3a,E,M,6、7a,7b,8和N)和gRNA。

图3 SARS-CoV-2基因组结构示意图
SARS-CoV-2除了具有预期结构和长度的经典sgRNA(图4D),结果还显示出许多次要的junction sites(图4E–4G)。这种融合事件有三种主要类型。第一组中的RNA在位于ORF或UTR中间非预期的3’位点,前导序列(leader)与下游编码序列(body)结合(图4E,TRS-L-dependent noncanonical)。第二组显示了与前导序列没有相似性的序列之间的远距离融合(图4F, TRS-L-independent distant)。最后一组局部融合,这导致较小的缺失,主要发生在结构基因和辅助基因中,包括S ORF(图4G,TRS-L-independent local recombination)。

图4 典型和非典型融合事件图
2. SARS-CoV-2的poly(A)长度
由于纳米孔DRS基于RNA的单分子检测,因此它提供了独特的机会来检查单个RNA分子的多个转录组特征。本文使用自己开发的软件根据DRS数据来测量ploy(A)尾的长度(Y. Choi and H.C., unpublished data)。使用此软件测量发现,与其他CoV一样,SARS-CoV-2 RNA带有ploy(A)尾(图5A–5B)。病毒RNA尾的中位长度为47 nt。全长gRNA的尾巴比sgRNA更长。值得注意的是,sgRNA具有两个种群:一个在30 nt处的小峰和一个在45nt处的主峰(图5B,箭头)。之前研究发现 bovine CoV mRNA的ploy(A)尾长度在感染过程中发生了变化,在这项研究中观察到的30 nt的短尾可能代表易于衰变的老化RNA。与宿主核基因组编码的mRNA不同,病毒RNA具有均匀的长度分布(图5C)。

图5 Poly(A) tail长度图
3. SARS-CoV-2的RNA修饰
研究人员用DRS数据检测了SARS-CoV-2的表观转录组学。病毒RNA修饰在40多年前就已描述过(Gokhale and Horner,2017)。为了明确研究RNA修饰,作者通过体外转录病毒序列生成了阴性对照RNA,并对阴性对照进行了直接RNA测序。注意到阴性对照和病毒转录本之间的离子电流之间的差异非常有趣(图6A)。至少有41个位点显示出实质性差异(频率超过20%),表明存在潜在的RNA修饰位点。值得注意的是,某些位点显示的频率取决于sgRNA的种类。图6A–6C,S RNA上比N RNA修饰的位点更多,与S RNA相比ORF8 RNA上的修饰频率更高。此外,修饰的碱基(图6D,右)的停留时间比未修饰的碱基(图6D,左)的停留时间长,表明修饰会干扰RNA分子过孔速度。
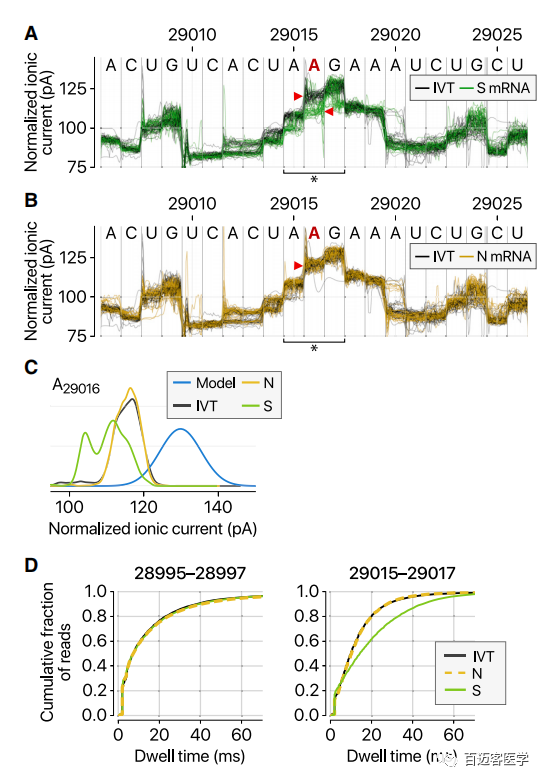
图6 RNA修饰位点图
在41个潜在的修饰位点中,最常见的motif是AAGAA(图7A和7B)。在整个病毒基因组中都发现了“AAGAA-like”motif的修饰位点(包括AAGAA和其他富含A/G的序列),尤其是在基因组28,500-29,500处特别丰富(图7C)。病毒长的转录本(gRNA,S,3a,E和M)比短RNA(6、7a,7b,8和N)的修饰频率更高(图7D),表明该修饰是特定于某些RNA种类。因为DRS可以同时检测单个分子上的多个特征,所以作者交叉检测了poly(A)尾长和内部修饰位点。有趣的是,修饰的RNA分子的poly(A)尾比未修饰的短(图7E)。这些结果表明内部修饰和3’尾之间存在联系。由于poly(A)尾巴在RNA周转中起重要作用,因此很容易推测所观察到的内部修饰与病毒RNA稳定性控制有关。
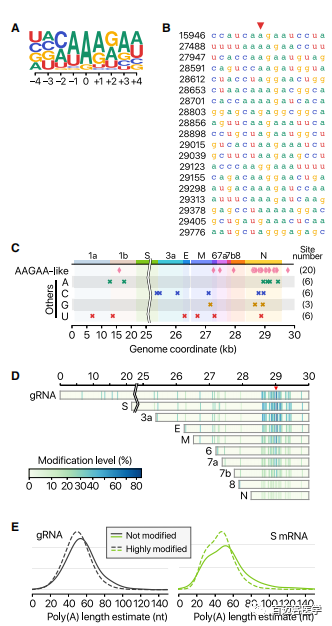
图7 检测到的RNA修饰差异调节图
讨论
在这项研究中,作者描述了SARS-CoV-2的转录组和表观转录组。对sgRNA和ORF的明确定位为病毒蛋白功能研究,复制机制以及致病性中宿主-病毒相互作用的提供前提。与RNA病毒一样,冠状病毒也经常重组,这可能会使它们迅速进化,从而改变其宿主/组织特异性和药物敏感性,在这项研究中检测到的频繁融合可能为变异提供基础。新的ORF可以作为辅助蛋白,调节病毒复制和宿主免疫反应。RNA修饰也可能有助于感染组织中的病毒存活和免疫逃逸。ORF和RNA修饰是SARS-CoV-2特有的还是在其他冠状病毒中保守的,有待检查。对它们的分布和功能意义的比较研究将有助于我们更全面地了解SARS-CoV-2和冠状病毒。







 京公网安备 11011302003368号
京公网安备 11011302003368号