Nanopore测序错误率相对于二代平台高,所以我们不推荐其做snp分析,但这不代表高深度ONT全基因组重测序不能用于snp分析。本期小编为大家分享一篇利用高深度ONT全基因组重测序进行snp分析以进行临床应用的案例。

【研究背景和方法】
Nanopore长读长测序在人类基因组测序方面主要优势集中于基因组组装及结构变异检测方面。由于其碱基错误率较高,其在临床应用所需的单核苷酸变异(SNV)检测方面存在困难。
为了评估纳米孔测序在临床人类基因组学的应用潜力,作者利用便携式MinION三代测序仪测序了2个人类基因组:基因组参考样本NA12878,增加其测序深度,以评估和校准三代nanopore变异检测方法;然后对伴有严重免疫失调的共济失调性全血细胞减少综合征患者进行测序,以解决与分子遗传学诊断相关的2种新生蛋白编码变异的染色体定相(phasing)相关问题。
【研究结果】
1.对参考样品进行MinION全基因组测序
GM12878人B淋巴细胞采用PCR扩增和6kb片段筛选的文库制备方案,共计获得45,740,123条reads(图1a),平均读取长度为6373bp(图1b)在流动细胞中是一致的,并且基于测序文库的物理大小选择非常接近预期。总数据量为273.4Gb,每个flow cell的平均产量为3.7Gb(图1c)。总计42,924,782个高质量clean reads的比对率为99.3%,唯一比对率为88.8%。
比对上的reads平均碱基替换SNV发生率为12.7%(与参考碱基不同的频率),平均缺失率为4.7%(参考序列中碱基缺失的频率),平均插入率为3.2%(图1d)。作者还评估了不同碱基识别算法对reads水平z确性的影响,发现Albacore v2.0.2实现了*低的未过滤替换错误率和缺失错误率,而其他方法具有较低的插入错误率。
平均每个碱基覆盖深度(不包括缺失)为81.7X(图1e),其中90.4%的基因组区域被至少40个reads覆盖。9.6%人基因组区域覆盖深度降低(<40×),反映文库制备方案的PCR步骤中的扩增偏差(图1f)。
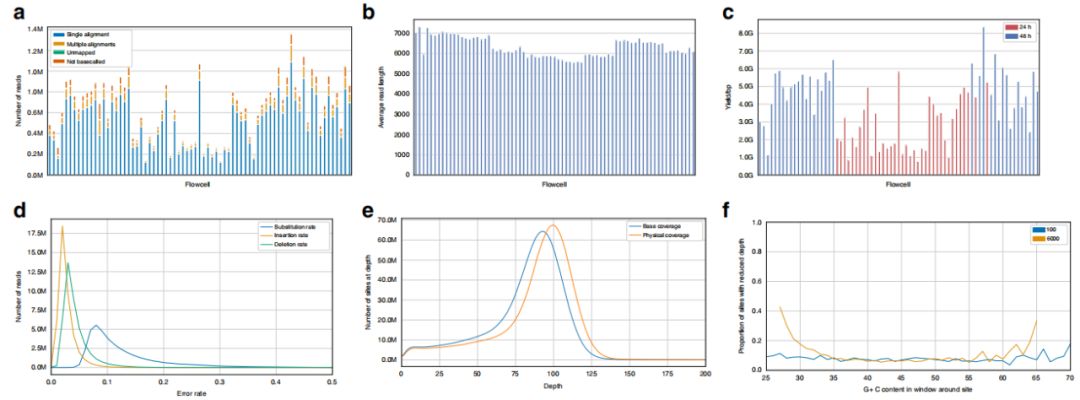
图1
2.NA12878中的单核苷酸变异SNV检测
使用multi-platform Genomes in a Bottle (GIAB)作为黄金标准真实数据集评估ONT检测SNV的z确性。NA12878样本22号染色体数据运行FreeBayes方法检测SNV,选择获得*佳F1分数的参数,与GIAB参考变异检测集相比,实现了99.9%的总体一致性z确度,并且观察到12.8%的错误发现率(FDR)和14.4%假阴性率(FNR),结合创造了86.4%的F1分数(表1)。在全基因组水平使用上述参数,获得了10.9%的FDR,12.5%的FNR和88%的F1分数。
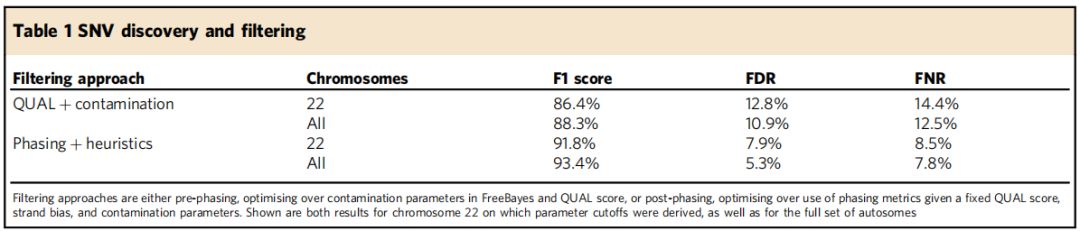
表1
为了更好地理解变异检测错误的潜在来源,作者注释了变异检测位点,其中包含一系列关于参考序列和跨越位点的reads注释。这些包括接近均聚物重复区域、较低的覆盖深度、链偏好和存在大量短缺失的reads覆盖区域(图2左)。表明,假阳性(FP)和假阴性的主要驱动因素是均聚物和低覆盖率。此外,使用高质量评分阈值(QUAL)来维持可接受的FDR会产生许多假阴性。
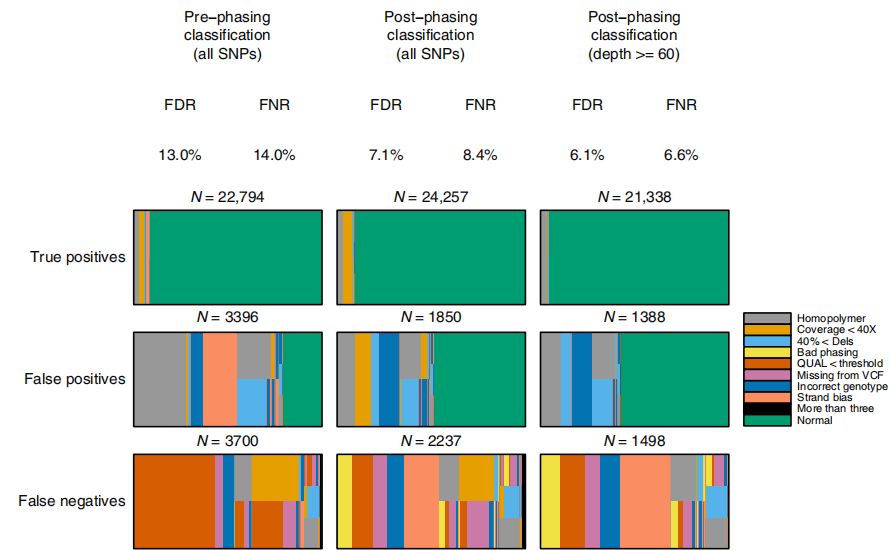
图2
初始变异集中的大部分假阳性基因型是杂合基因型。使用ONT数据的好处是跨越多个杂合位点的长reads提供了纠正此问题的机会。当reads被分成代表亲本单倍型的2组时,预期真阳性变异等位基因只固定在一个定相组(亲本单倍体)存在,而假阳性变异预期在组之间均匀分布。据此,作者开发了单样本、基于reads、无参考panel的定相算法。
使用过滤器改进变异检测,通过phasing和注释过滤器(Post−phasing classification)显著改善变异检测,*佳结果F1评分为92.2%,FDR为7.1%,FNR为8.5%(表1,图2中)。进一步考虑覆盖深度>=60X的假定变异位点(基因组的85%)时,观察到F1得分改善至93.6%,FDR为6.1%,FNR为6.6%(图2右),这意味着减少或消除覆盖深度偏差源(如PCR)的操作改进在提高z确性方面可以发挥一定作用。(百迈客目前ONT全基因组重测序和ONT全基因组甲基化测序建库过程正是PCR-free建库–direct-DNA建库,一是可减少覆盖深度偏好,有利于提高变异检测z确度;二是可以保留碱基修饰信息,同时检测甲基化修饰等信息)
在推定的致病LOF变异(功能缺失突变:本文针对终止密码子获得和剪接位点突变)中,与全基因组真阳性突变(173/788782,0.02%)相比,FPs(假阳性突变,69/45219,0.15%)富集,但FPs在高度不耐受LOF突变的基因(pLI>0.90,17 FP对20 TP)与LOF突变耐受基因(pLI <= 0.10,46 FP对122 TP)中成比例地富集。
在每条read随机碱基替换错误和无基因组扩增偏差的理想化模型下模拟NA12878数据集,与实测数据比较,表明均聚物缺失错误累积导致缺失变异检测错误,纳米孔测序中增加的测序覆盖深度以减小均聚物相关FDR,目前受到基因组范围的in-read缺失率的限制。
3.NA12878样本突变定相
基因型是通过母本或父本单倍型遗传的,但大多数基因分型方法,会产生非定相基因型检测,即无法区分单倍型。基因型定相很有意义,除了上述促进变异检测z确性的改进之外,还能够进行许多遗传分析,比如临床用途中解决多个杂合LoF变异的共分离和鉴定新生突变的起源亲本。作者开发的新型定相算法相比于其他算法,具有更低的错误率,其定相√确度类似于使用非常大的参考panel从SNP基因分型阵列数据定相常见变异所获得的定相√确度。
4.NA12878样本大片段结构变异检测
大的结构变异相对于snp和indel少见,但其对罕见疾病的影响可能甚至比目前估计的更大,因为现有分析检测这些突变存在技术困难。采用Sniffles检测22号染色体SV变异,共计检测到82个,其中22个是在GIAB真实数据集中存在的,之后通过ONT、Illumina和PacBio reads覆盖数据来判断剩余的60个SV:其中21个SV被Pacbio检测到或reads强烈支持,31个SV仅ONT reads明显证实,但PacBio reads很少或不支持。ONT特异性检测SV可能代表其他技术遗漏的真实缺失、由PCR扩增产生的假象或在NA12878细胞系的细胞培养期间发生的亚克隆缺失。作者发现目前ONT平台允许检测大的缺失,灵敏度在60%-91%(21/35和32/35)。
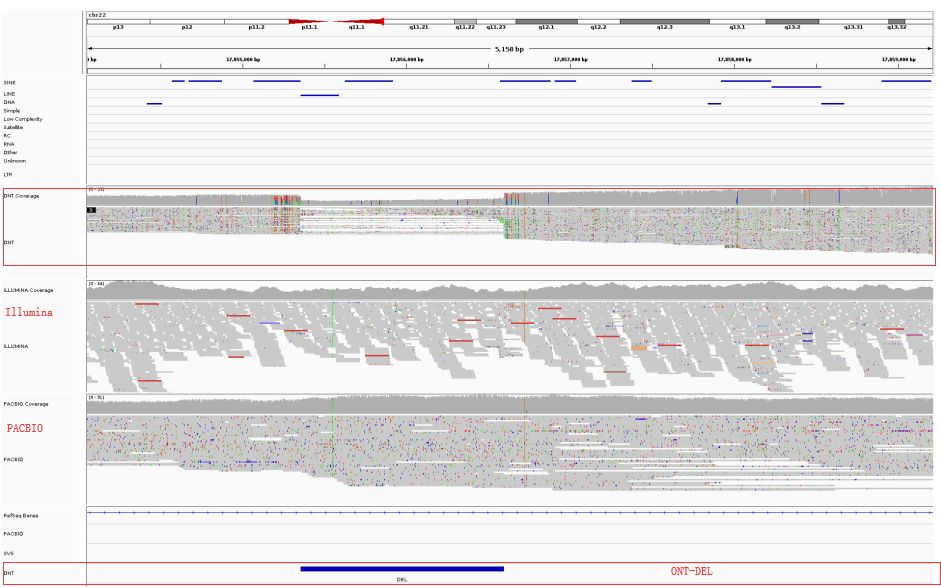
仅ONT检测到的缺失突变示例
5.使用MinION对临床样品进行全基因组测序
鉴于长reads可成功地检测杂合变异,作者试图使用全基因组纳米孔测序来解决具有不确定的免疫调节病症的个体基因组临床问题。简而言之,女性患者最初在婴儿期出现复发性感染、低丙种球蛋白血症、血小板减少症和轻度贫血,并且在儿童时期出现慢性炎症,在成年早期出现进行性神经系统症状。
患者及其父母组成的核心家系3个样本Illumina平台全基因组重测序(PE 126bp)结果:发现了84个高置信度的新生SNV,一个接近预期范围上限的数字,这与受孕时的父母年龄(母亲是38岁,父亲39岁)一致。其中3个变异预测为导致蛋白序列改变,2个位于SAMD9L基因蛋白质编码区中。该基因中罕见的杂合变异最近涉及常染色体显性遗传性共济失调性全血细胞减少综合征(OMIM:#159550),并且有证据表明造血组织的出生后逆转可能与较轻微的疾病表现相关。虽然这2个非同义突变(c.1076 G>A和c.3353 A>G; p.R359Q和p.Y1118C,NM_152703.3)位于同一个外显子中,但它们相距2277 bp,所以不能直接使用Illumina reads进行定相;其附近缺乏遗传的杂合变异也阻止将突变定相于亲本单倍型。解释这个问题对于解释每个等位基因的致病潜力非常重要,解决此类问题的能力与类似情况下的生殖决策直接相关。
全血样本Nanopore全基因组重测序结果:在34个R9.4 MinION flowcell中共计产生122 Gb数据量(16,692,656 reads,约40X),比对率为99.1%。通过上述在NA12878样本确定的变异检测和定相方法进行分析。
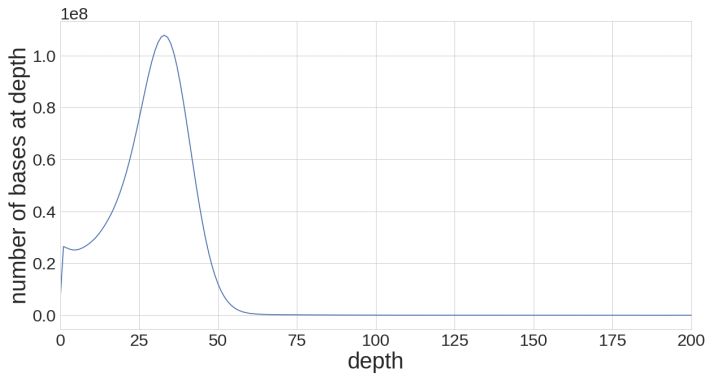
ONT数据基因组覆盖深度统计
不出所料,ONT数据也鉴定到了具有预期杂合基因型的c.1076 G> A和c.3353 A> G变异。使用ONT reads对其他附近变异进行定相,以确认新生变异的遗传和来源(图3)。这2个新生突变被定相于一个199kb的block内,33条reads(6.1-18.9kb)跨越2个新生突变位点,11条reads包含新生突变等位基因,8条reads包含2个参考等位基因,表明突变的等位基因是顺式的(来自于同一条染色体)。(NA12878数据中没有reads跨越包含2个位点的突变等位基因。)使用一系列等位基因特异性PCR实验证实来自ONT reads的新生等位基因的单倍型构象。相位区中的侧翼位点表明,父系遗传的单倍型出现了新生变异(图3)。
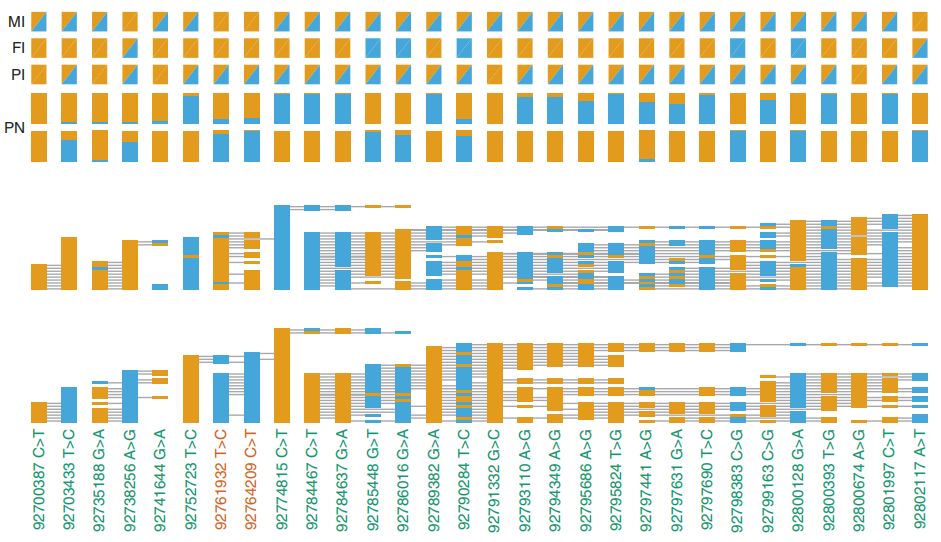
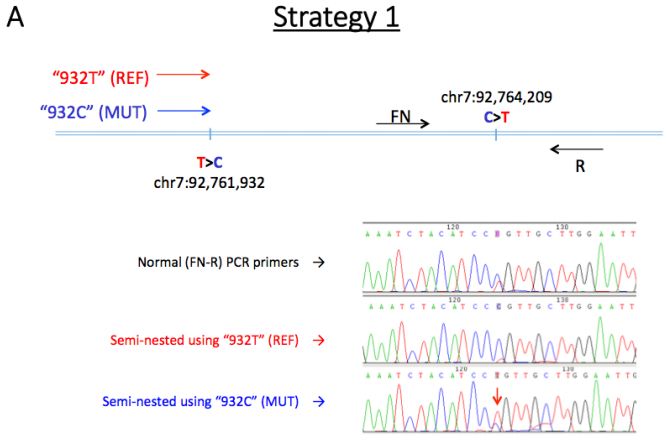
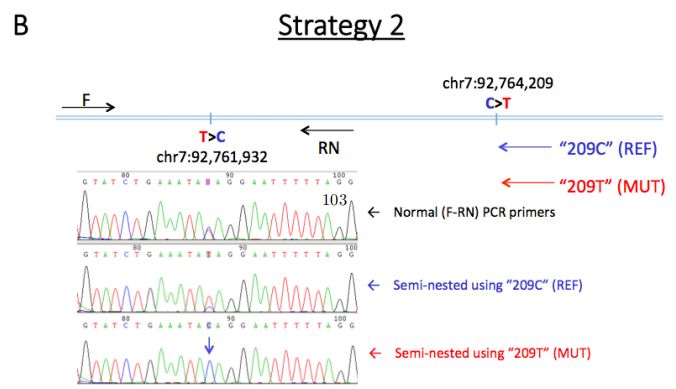
图3及等位基因特异性PCR结果
注:前3行为未定相母亲(MI),父亲(FI),先证者(PI)基因型,第4行为先证者单倍型Phased proband genotypes (PN)。蓝色=alt ,橙色=ref。PN下面2行为单倍型1(母系遗传)或单倍型2(父系遗传)对应的reads,其中对于每条read,碱基是矩形,reads跨度以水平线显示。间隙代表gap(缺失)。底部显示物理位置,感兴趣的位点为红色。基于GRCh37 NM_152703.3, 92761932 T>C对应于c.3353 A>G,92764209 C>T对应于c.1076 G>A。
【讨论】
该研究首次详细评估了ONT测序对人类样本的变异检测和基因分型、染色体定相(单倍型分析)的z确性。虽然很有希望,在总共107个MinION flowcell中对这2个人类基因组进行测序是一项重大任务,在技术和计算等方面具有挑战。最近商业化推出的PromethION是一种更高通量的纳米孔测序仪,自带数据处理功能,有望解决人类基因组规模数据中的许多挑战。(百迈客与Oxford Nanopore公司合作-斥巨资引进Nanopore全测序平台)最后,虽然变异检测的总体z确性仍存在局限性,但该工作突出了错误上下文,这些错误上下文将受益于基本检测、reads比对和一致性变异检测方法的改进,并说明了将ONT应用于临床目的的途径。
【小编碎语】
随着nanopore测序技术的发展更新,比如ONT内测的R10芯片75X达到一致性质量值Q50,比如新的”flip-flop”碱基识别软件可将R9一致性z确性提升至Q42等。总之,ONT长读长测序错误率down down down,测序通量up up up,测序价格low low low,三代取代二代指日可待。谁说鱼(长读长)和熊掌(z确度)不可兼得呢。
Bowden R, Davies R W, Heger A, et al. Sequencing of human genomes with nanopore technology[J]. Nature communications, 2019, 10(1): 1869.
文献原文下载:
https://international.biocloud.net/zh/article/detail/31015479







 京公网安备 11011302003368号
京公网安备 11011302003368号