Hi-C是2009年提出的染色体构象捕获结合高通量测序的一种技术(Erez Lieberman-Aiden, et al,Science),是衍生于染色体构象捕获技术(3C)的高通量技术,实现了全基因组范围内的染色体片段间的相互作用的捕获检测。作为染色体三维构象与互作研究的利器,Hi-C技术广泛应用于肿瘤/疾病发生发展机制、分化发育机制、畸变机制等研究。

同样源于3C的用以研究染色体互作的技术还包括4C(“circular 3C”或“3C-on-Chip”)、5C(Chromosome conformation capture carbon copy) 、ChIA-PET(chromatin interaction analysis by paired-end tag sequencing) ,不同的3C衍生技术的区别在于捕获的连接片段检测和定量方式。
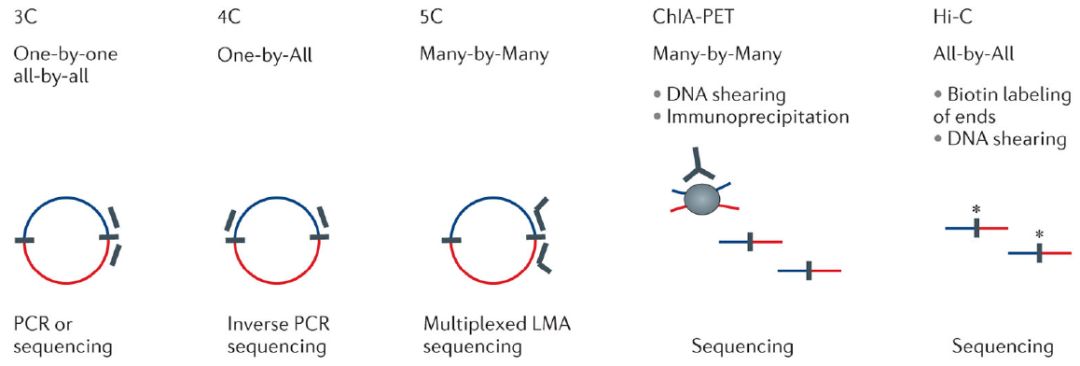
3C:经典的3C实验中,通过基因座特异性引物PCR检测单个连接产物,大多数3C通常仅能分析几十到几百Kb染色质之间的相互作用,通量低,费时费力——one vs one。
4C:使用反向PCR产生单基因座的全基因组相互作用图,研究已知DNA片段(bait)与全基因组未知DNA片段之间的互作——one vs all。
5C:基于3C的基本原理,结合连接介导的扩增 (ligation-mediated amplification,LMA)来增加3C检测的通量,识别两组大量位点之间并行的数百万个相互作用,例如一组启动子和一组远端调控元件之间的互作——many vs many。
ChIA-PET:对感兴趣的蛋白质结合位点之间的远程互作进行全基因组分析。
Hi-C:提供了一个真正全基因组范围的相互作用图谱(该图谱的分辨率取决于测序的深度)——all vs all。
Hi-C之技术流程
Hi-C的主要原理是将空间结构临近的DNA片段进行交联,并将交联的DNA片段富集,然后进行高通量测序,对测序数据进行分析,即可揭示染色体片段间的交互信息,阐述染色体三维构象。
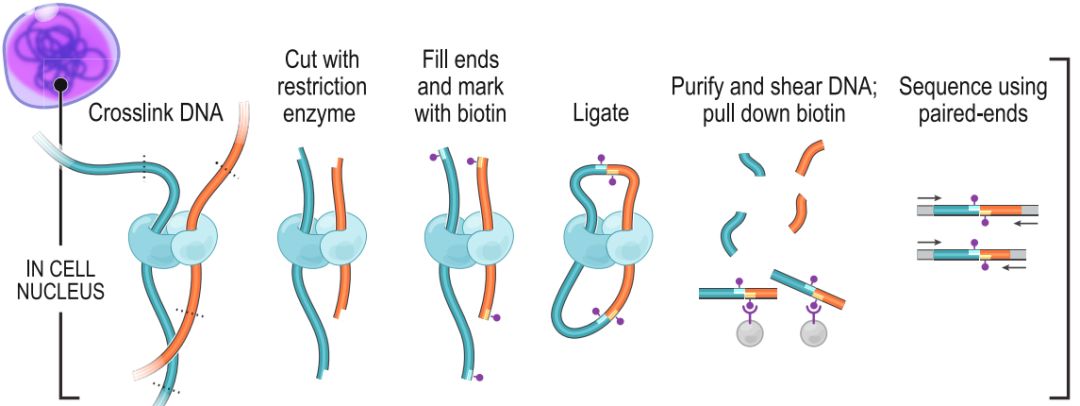
(Rao S S,et al.,Cell)
Hi-C与普通二代测序(如全基因组重测序等)最大的区别在于前期建库时对构象的固定与捕获,Hi-C文库制备流程主要包括甲醛交联、细胞裂解、内切酶酶切、末端修复及生物素标记、片段连接、捕获带生物素标记的片段、文库质检等步骤。Hi-C文库质量受多种因素影响,如细胞裂解剧烈程度、细胞裂解期间的蛋白酶抑制剂含量等,Hi-C文库质量直接影响后续的有效数据产出。
因而Hi-C技术难度较高的主要难点就在于建库,大多数公司在建库这一环节就已经被排除门外,有些公司虽然能做但也可能需要多次建库。百迈客Hi-C标准建库流程已申请国家专利,我们致力于将Hi-C打造成类似于转录组一样技术成熟的产品,成为功能基因研究的好帮手。
百迈客一直保持着近100%的建库成功率,即使是次生代谢物种类较多的建库难度大的植物,百迈客的一次建库成功率在97%以上,不仅如此,凭借一定的技术优势和项目经验,BMK已经帮助客户解决了某些Hi-C“疑难杂症”,并助力客户的科研进展。
合格的Hi-C文库对于提升有效数据量具有重要作用,因此有效Hi-C数据量的高低会直接反映Hi-C文库建库质量的高低,主要从Valid Interaction Pairs含量对Hi-C数据进行评估。Invalid Interaction Pairs主要主要包含自连类型、末端悬挂类型等。Valid Interaction Pairs在30%以上即认为数据合格,根据已有项目经验,百迈客动物样品文库的有效数据最高达86.68%。
Hi-C之研究内容
通过Hi-C技术可以获得全基因组范围内的互作信息,得到染色体三个层级的三维结构:A/B compartment、拓扑相关结构域(TAD)、染色质环(loop)。
A/B compartment
染色质划分为A、B两种compartments:
A compartments:常染色质,松散染色质状态、高基因密度、转录活跃区域,富集转录活性相关的表观遗传标记(例如H3K4me3)。
B compartments:异染色质,压缩染色质状态、低基因密度区域、转录抑制区域,富集无转录活性的表观遗传标记(例如H3K27me3)。
A/B compartments具有组织、时期、状态特异性,在不同组织或不同时期或疾病状态间能够发生转换,这种转换和基因表达调控有一定关系。当染色体上某区域发生A/B compartment转化,会影响其中的基因的转录活性,通常B compartment转换为A compartment的区域相关基因大多表达上调,而A compartment转换为B compartment的区域相关基因则下调。
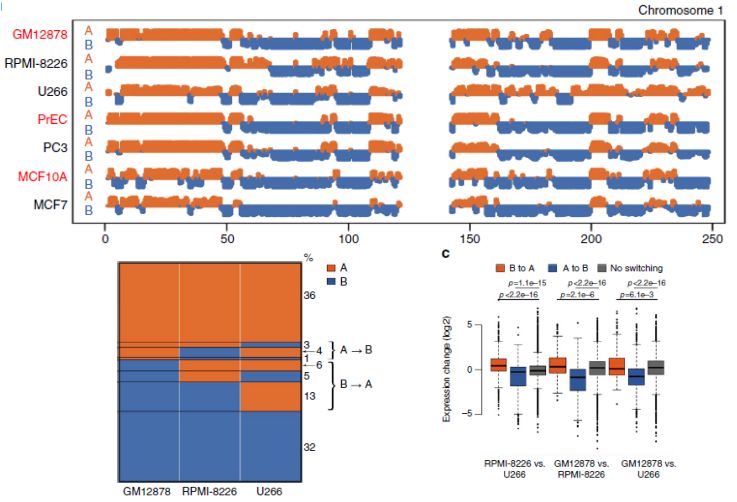
肿瘤细胞系与正常细胞系间A/B compartment转换(Pengze Wu1,et al,NC)
拓扑相关结构域(TAD)
拓扑相关结构域(topologically associating domains,TAD)是一段具有折叠结构的DNA序列,此区域内部的互作频率会显著高于毗邻的两个区域之间的互作频率,TAD是基因组在空间结构中的基本组织形式。TAD结构在不同时空下(组织、发育阶段、状态等)具有一定的保守性,同时也有一些动态变化。
TAD边界有明显的界限,通常存在大量的绝缘子、管家基因等,其内部是一个独立的调控单元,内部的基因存在协同表达特征;TAD边界具有很有很重要作用,将边界部分删除后会使得基因调控变得紊乱,导致原来沉默的基因被转录,而原来应该转录的则沉默了。
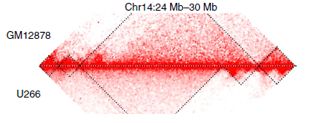
肿瘤细胞系与正常细胞系的TAD(Pengze Wu1,et al,NC)
染色质环(loop)
DNA在CTCF等蛋白质的介导下形成远距离互作,这种结构通常称为loop结构。通过Hi-C可以检测到线性距离很远而空间距离被拉至很近的DNA片段,即peak位点,它们之间的交互频率往往高于线性上相邻的片段,以peak位点来确定loop位置。
同样loop结构在不同时空下(组织、发育阶段、状态等)会有一些动态变化,且通过loop锚定的位点中包含启动子、增强子、沉默子等,当发生loop结构的动态变化,如新形成或消失,在一定程度上会影响基因的调控。
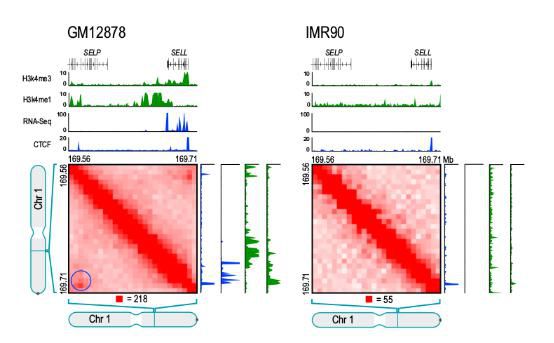
loop与基因转录开/关(Rao S S,et al.,Cell,2014)
如果您的科研项目有任何问题,欢迎点击下方按钮咨询我们。








 京公网安备 11011302003368号
京公网安备 11011302003368号