Circos图是在基因组相关的分析结果展示中非常常见的一类图型。由于Circos图中展示的信息量大,图形十分美观,很多文章中都会绘制类似的图形。从图形的内容来看,Circos图实际是将多个类型的图片,包括散点图、直方图、折线图、热图等,经过变形以后叠加显示在染色体上。本质上是多个图形的组合。
在R语言中,RCircos这个软件包可以实现快速生成Circos图片。接下来,我们就来看看怎样用R来绘制圈图。
1、基础绘图
# 加载RCicos,如果第一次使用,需要先安装
install.packages('RCircos')# 读取R包
library(RCircos)# 导入内建人类染色体数据
data(UCSC.HG38.Human.CytoBandIdeogram)
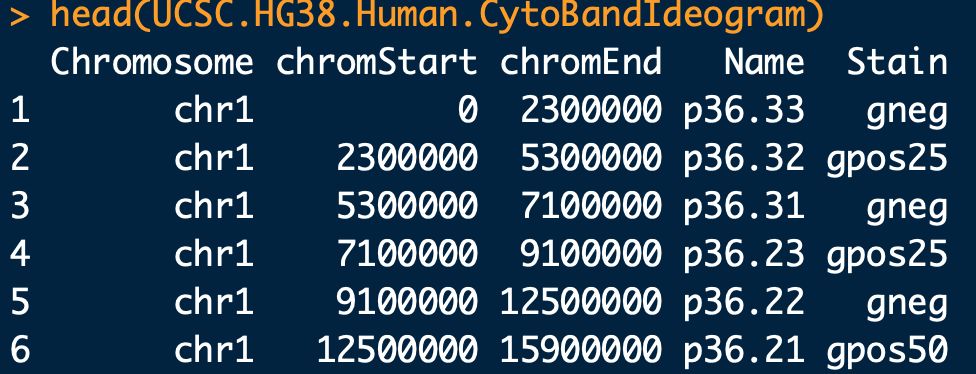
# 数据内容如上图;第一列 染色体编号;第二列 染色体片段起始位点;第三列 染色体片段结束位点;第四列 染色体片段编号;第五列 染色体片段颜色
# 这个数据是RCicos内置的人类染色体信息,第四列和第五列信息用于展示染色体的核型。可以省略。
# 设置染色体数据
cyto.info <- UCSC.HG38.Human.CytoBandIdeogram
RCircos.Set.Core.Components(cyto.info, chr.exclude=NULL,tracks.inside=10, tracks.outside=0 )
# chr.exclude=NULL; 设置不显示的染色体,如 c(1,3)
# tracks.inside=10; 设置内部环形个数
# tracks.outside=0; 设置外部环形个数
# 绘制染色体图形,默认方法显示染色体名称。
RCircos.Set.Plot.Area()
RCircos.Chromosome.Ideogram.Plot()
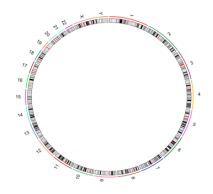
这样仅包含染色体的圈图就生成了:
2、绘制包含数据的图形
# 导入内建人类染色体数据
data(UCSC.HG19.Human.CytoBandIdeogram);
#设置不显示的染色体,如 c(1,3)
chr.exclude <- NULL;
#设置染色体数据
cyto.info <- UCSC.HG19.Human.CytoBandIdeogram;
#设置内部环形个数
tracks.inside <- 10;
#设置外部环形个数
tracks.outside <- 0;
# 导入上面四个基本参数
RCircos.Set.Core.Components(cyto.info, chr.exclude, tracks.inside, tracks.outside);
# 列出所有绘图参数
RCircos.List.Plot.Parameters()
# 定义要生成的图像文件
pdf(file="RCircosDemoHumanGenome", height=8, width=8, compress=TRUE);
RCircos.Set.Plot.Area();
# height和width指定生成图片的长和宽,compress指定生成的图片是否需要压缩
# 绘制染色体图形
RCircos.Chromosome.Ideogram.Plot();
3、染色体上添加基因名称与连线
# 加载内置的RCircos.Gene.Label.Data数据集
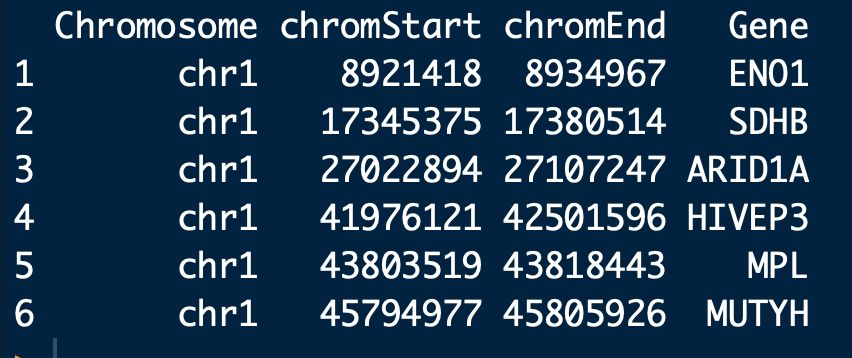
# 数据内容如上图,第一列 染色体编号,需要与第一步导入的染色体数据一致;第二列 基因在染色体片段起始位点;第三列 基因在染色体片段结束位点;第四列 基因名
# 指定内容在内侧的环形还是外侧的环形生成
side <- "in";# 指定内容在第几个环形生成
track.num <- 1;# 绘图
RCircos.Gene.Connector.Plot(RCircos.Gene.Label.Data, track.num, side);
# 在染色体上添加基因名称, 指定内容在第几个环形生成
name.col <- 4;# 指定基因名在数据的第几列
track.num <- 2;# 绘图
RCircos.Gene.Name.Plot(RCircos.Gene.Label.Data, name.col,track.num, side);
4、添加热图类型的环形
data(RCircos.Heatmap.Data);
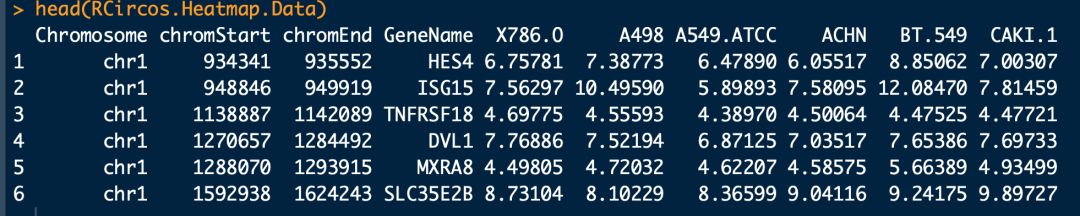
# 这个数据集中是6个不同样本的基因表达量数据,数据内容如上图,第一列 染色体编号,需要与第一步导入的染色体数据一致;第二列 基因在染色体片段起始位点;第三列 基因在染色体片段结束位点;第四列 基因名;第五列及以后 基因在不同样本中的表达量
# 指定以第6列数据生成热图
data.col <- 6; # 指定内容在第5个环形生成
track.num <- 5;# 指定内容在内侧的环形生成
side <- "in";# 绘图
RCircos.Heatmap.Plot(RCircos.Heatmap.Data, data.col, track.num, side);
5、添加散点图类型的环形
data(RCircos.Scatter.Data);
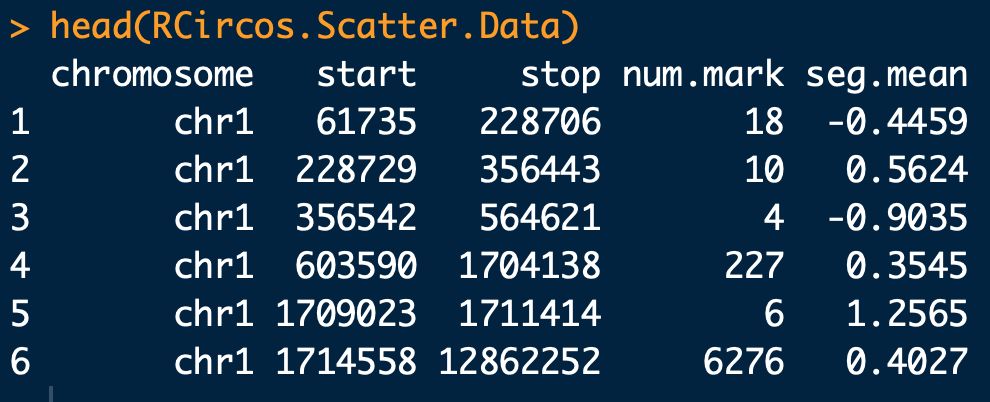
# 这个数据集是拷贝数变异的数据,数据内容如上图,第一列 染色体编号,需要与第一步导入的染色体数据一致;第二列 突变位点在染色体片段起始位点;第三列 突变位点在染色体片段结束位点;第四列 突变位点拷贝数;第五列 突变平均置信度
# 指定以第5列数据图形中散点纵坐标
data.col <- 5;# 指定图形在第6个环形生成
track.num <- 6;# 指定图形在内侧环形生成
side <- "in";# 指定数据大于1的点以红色显示,小于数据1的点以蓝色显示
by.fold <- 1;# 绘图
RCircos.Scatter.Plot(RCircos.Scatter.Data, data.col,track.num, side, by.fold);
6、添加折线图类型的环形
data(RCircos.Line.Data);
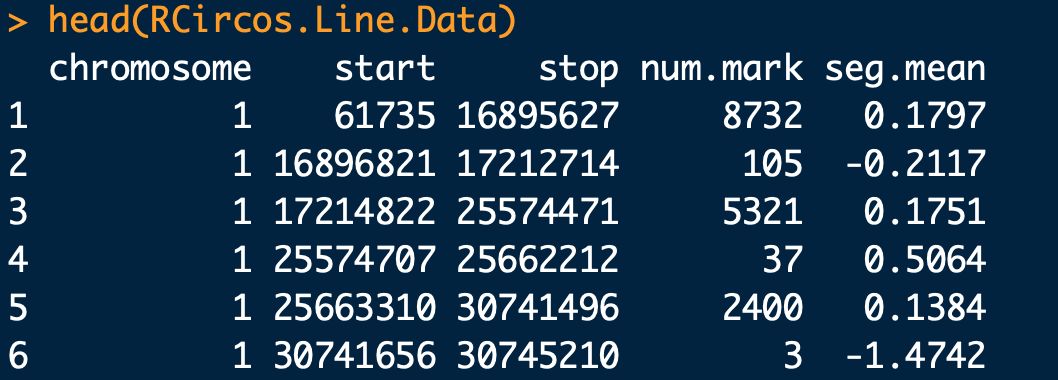
# 由于这个数据集的染色体编号与第一步导入的染色体数据不一致,需要在编号前添加chr
RCircos.Line.Data$chromosome <- paste(‘chr’,RCircos.Line.Data$chromosome,sep = ”)
# 指定以第5列数据做为图形中拆线的纵坐标
data.col <- 5;# 指定图形在第7个环形生成
track.num <- 7;# 指定图形在内侧环形生成
side <- "in";# 绘图
RCircos.Line.Plot(RCircos.Line.Data, data.col, track.num, side);
7、添加直方图类型的环形
# 加载内置的RCircos.Histogram.Data数据集
data(RCircos.Histogram.Data);
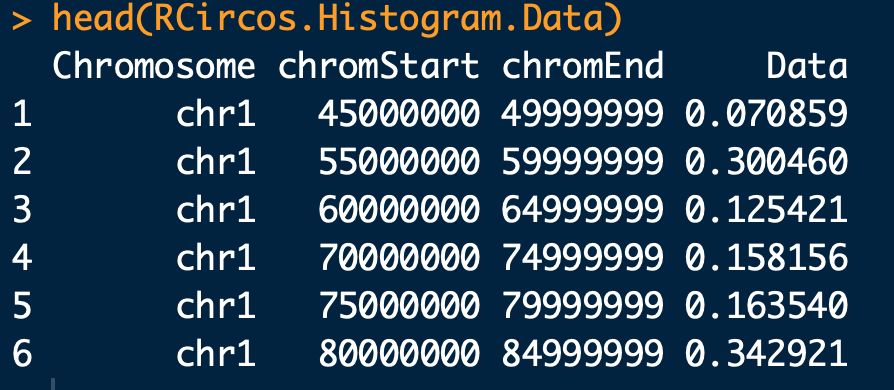
# 这个数据集是染色体片段突变频率的数据,数据内容如上图,第一列 染色体编号;第二列 染色体片段起始位点;第三列 染色体片段结束位点;第四列 染色体片段突变频率
# 指定以第4列数据做为图形中直方的纵坐标
data.col <- 4;# 指定图形在第8个环形生成
track.num <- 8;# 指定图形在内侧环形生成
side <- "in";# 绘图
RCircos.Histogram.Plot(RCircos.Histogram.Data, data.col, track.num, side);
8、添加网络图类型的环形
# 加载内置的RCircos.Tile.Data数据集
data(RCircos.Tile.Data);
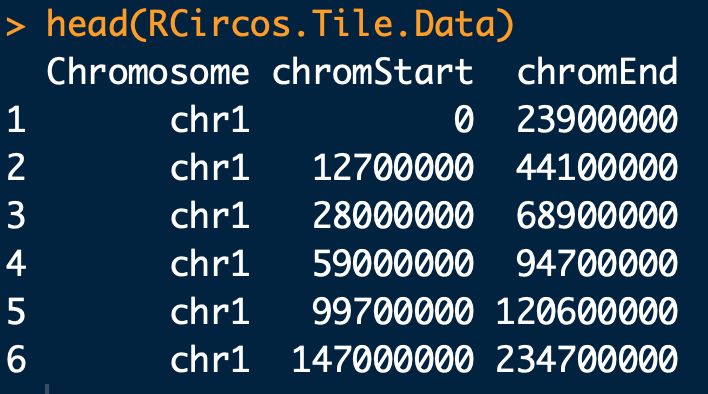
# 数据内容如上图,第一列 染色体编号;第二列 需要绘制的网格在染色体片段的起始位点;第三列 需要绘制的网格在染色体片段的结束位点
# 指定图形在第9个环形生成
track.num <- 9;# 指定图形在内侧环形生成
side <- "in";# 绘图
RCircos.Tile.Plot(RCircos.Tile.Data, track.num, side);
9、添加一个或多个基因间的联系曲线
# 加载内置的RCircos.Link.Data数据集
data(RCircos.Link.Data);
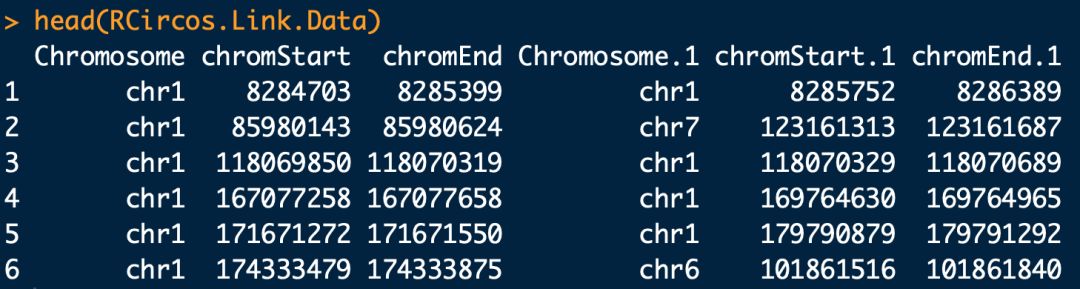
# 数据内容如上图,第一列 染色体编号;第二列 联系位点在染色体片段的起始位点;第三列 联系位点在染色体片段的结束位点;第四列 联系位点在所在的另一个染色体编号;第五列 联系位点在所在的另一个染色体片段的起始位点;第六列 联系位点在所在的另一个染色体片段的结束位点
# 指定图形在第11个环形生成
track.num <- 11;# 绘图
RCircos.Link.Plot(RCircos.Link.Data, track.num, TRUE);
# 加载内置的RCircos.Ribbon.Data数据集
data(RCircos.Ribbon.Data);
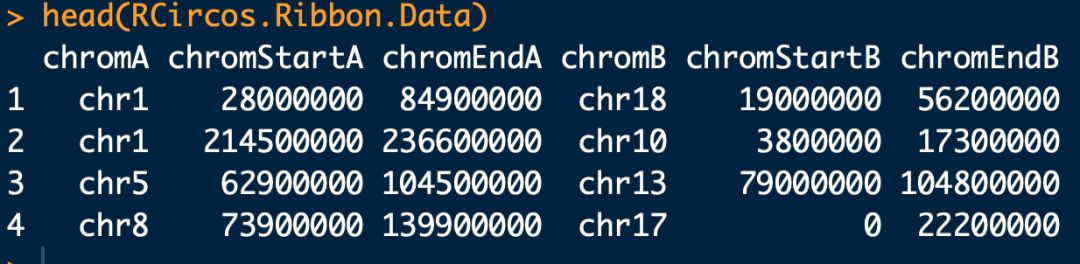 # 绘图
RCircos.Ribbon.Plot(ribbon.data=RCircos.Ribbon.Data, track.num=11, by.chromosome=FALSE, twist=FALSE);
# 绘图完成之后关闭图形设备,保存图片。可以在工作目录下找到生成的图片,默认是PDF格式
dev.off();
# 绘图
RCircos.Ribbon.Plot(ribbon.data=RCircos.Ribbon.Data, track.num=11, by.chromosome=FALSE, twist=FALSE);
# 绘图完成之后关闭图形设备,保存图片。可以在工作目录下找到生成的图片,默认是PDF格式
dev.off();
最终绘图效果如下图:
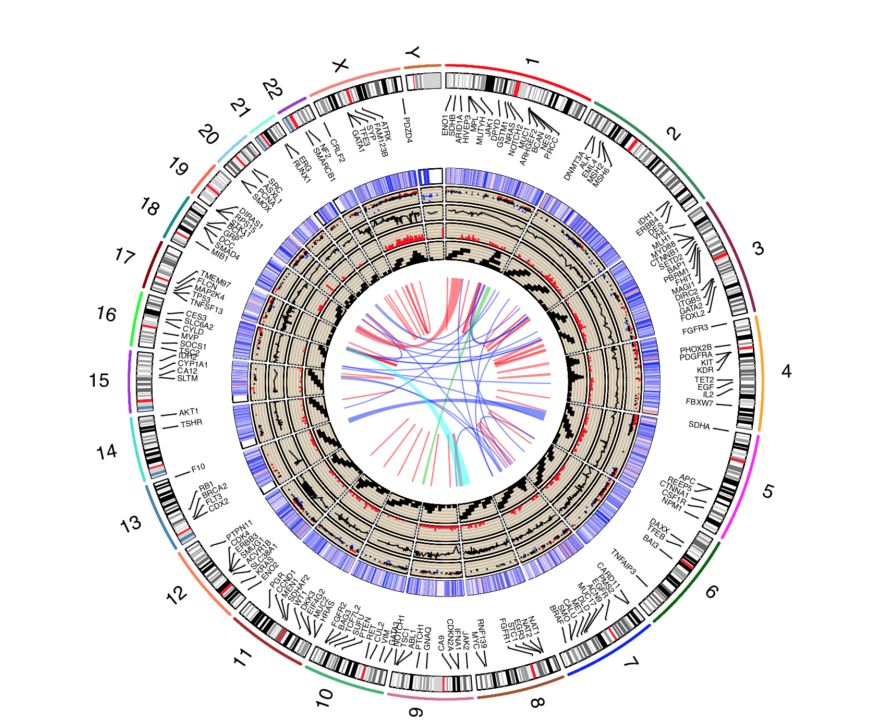
以上就是一个使用数据生成多个环形组合图的过程。我们还可以根据自己的需要添加更多的类似图形。只需要保证数据的结构一致即可。同样,也可以以上面的代码为模板,把其它数据代换进去生成全新图片。
如果您对这个绘图还有其他问题,欢迎点击下方按钮联系我们,我们将免费为您设计文章思路方案。









 京公网安备 11011302003368号
京公网安备 11011302003368号