叶绿体在光合作用以及激素、维生素、类胡萝卜素、氨基酸以及脂肪酸等重要物质的合成代谢中扮演重要角色,而叶绿体的发育受到核基因与叶绿体基因协同互作调节。迄今为止,大部分有关叶绿体发育的生理及遗传调控研究都集中于叶片,而对于果实中调控叶绿体发育的研究几乎为零。果实中同样存在光合元件并且十分活跃,然而,一般认为果实中大部分光合产物是由叶片转运而来。
番茄一直被视为研究果肉发育的模式植物,然而,大多研究集中于果实成熟的调控。有一些基因被认为调控果实叶绿体发育,例如,GLK2是叶片与果实组织叶绿体发育的正调控因子,通过激活光合相关基因的表达来调控叶绿体发育;在野生型番茄中,SlGLK2及其他光合、叶绿体相关基因呈现梯度表达,在幼嫩果实中常呈现“绿肩”表型。另一个相关转录因子APRR2-LIKE在调控叶绿体发育中,与SlGLK2表现相似功能。其他一些正向或负向调控因子,如BEL-LIKE HOMEODOMAIN 11,SlARF10,DET1等。
辣椒在幼果果色及叶绿素含量方面存在丰富自然变异,控制这种变异的第一个基因是APRR2-LIKE,其功能缺失会降低叶绿素含量并出现白色幼果。Brand et al., 2012利用遗传群体定位到两个控制叶绿素含量的主效QTL,pc8.1与pc10.1,研究发现CaGLK2与pc10.1共分离,并调控叶绿素含量,暗示CaGLK2是pc10.1的靶标基因,而对于pc8.1的靶标基因还不清楚。
今天给大家分享一篇由以色列与美国科学家合作完成的一项研究“The zinc-finger transcription factor CcLOL1 controls chloroplast development and immature pepper fruit color in Capsicum chinense and its function is conserved in tomato”。该研究对QTL pc8.1(即pc1)进行解析,利用BSA-Seq、BSR-Seq、交换单株筛选、DEGs分析、CRISPR等方法定位并证明辣椒锌指转录因子CcLOL1是此QTL的靶标基因。研究发现,该基因以果实特异性方式控制叶绿体大小与叶绿素含量,并且在番茄果实中它的同源基因是保守的。

图1. 亲本:C. annuum 1154与C. chinense PI 152225(Brand et al., 2012)
材料与方法
1. 群体构建:1154(深绿;C. annuum),PI 152225(淡绿;C. chinense);86-45(BC4F3 NIL,含来自PI152225的pc1区段)(图 1.及图 2.)
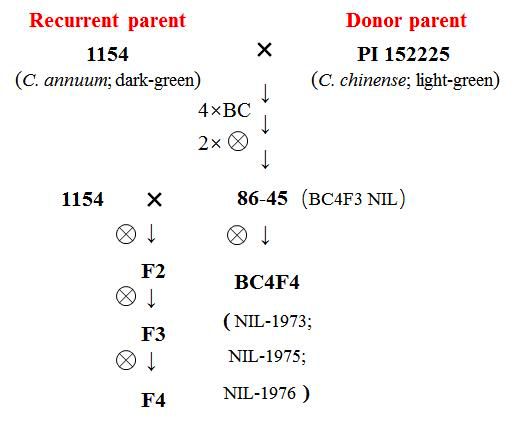
图2. 群体构建流程图
2. BSA-Seq:F2群体(1154 × NIL 86-45)混池——30个(淡绿果色的叶片)+30个(深绿果色的叶片),每个混池测序深度20×,PE150,Illumina HiSeq 4000;参考基因组—— CM334 v. 1.55 (Kim et al., 2014)
3. BSR-Seq:1154与PI 152225以及2个F2群体(1154 × NIL 86-45)极端混池——每个亲本或混池各取15个单株,每个单株取3个幼果,设3个生物学重复;SE60,Illumina HiSeq 2500;参考基因组—— CM334 v. 1.6 (Kim et al., 2017)
4. QTL-NILs差异表达分析: NIL-1973, NIL-1975, NIL-1976与亲本 (1154与PI 152225);取样时间为授粉后10天与30天, 3个生物学重复;Illumina HiSeq 4000 single-read sequencing technology
5. 色素含量、白利度及成熟果实重量考察
6. 叶绿体结构观察:Leica SP8激光共聚焦显微镜 (Leica, Wetzlar, Germany);检测叶绿体面积与单位面积叶绿体数目
7. CRISPR/Cas9:敲除番茄基因Solyc08g077060 (tomato ortholog of CcLOL1),获得基因敲除株系lol1CR-25、lol1CR-43、lol1CR-57与lol1CR-58
8. 品种:C. annuum accessions——1154;C. annuum var glabriusculum accessions——Cora, Nayoi and 1202;C. chinense accessions——CA4, USDA 162, 170, 174-3, 164-3, 180-2, PI 159236, 187-2, 4, 165-2
结果与分析
BSA-Seq分析(pc1区域)
为确定pc1在基因组中的具体位置,本研究利用F2群体(1154 × NIL 86-45)极端混池进行BSA-Seq分析,通过与参考基因组CM334 v. 1.55比对,混池间共检测出14805个SNPs,其中,有8235个SNPs分布在Chr.1上; 另外还有5670个SNPs位于4条未被组装的scaffolds上(scaffold1362, scaffold1381,scaffold1659与scaffold799),有趣的是,这些scaffolds上的番茄同源基因与辣椒Chr.1存在共线性。SNPs在Chr.1上分布特点是从0M-16M SNP数目逐渐增加,到17M后SNP数目突降,暗示渗入的QTL等位区段终止于这个位置。以上结果表明,pc1定位在Chr.1的SNP峰值区或者富含SNPs的未被组装的scaffolds上(图3.a)。
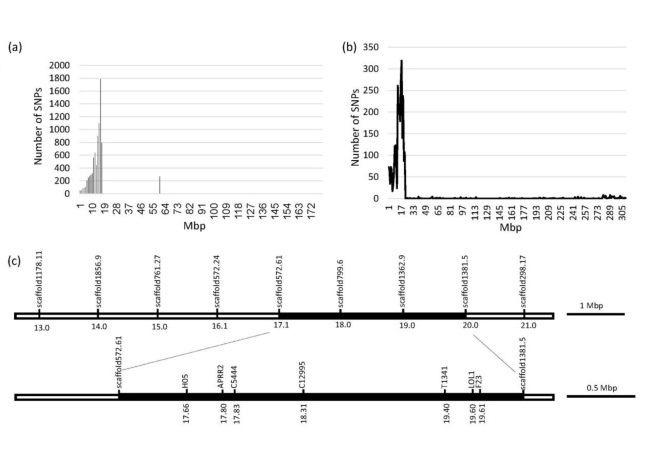
图3. 混池间SNPs分布(a. BSA-seq与b. BSR-seq)及pc1区域物理图谱(c.)
重组分析(pc1区域)
为进一步精细定位pc1,本研究对624个F2(1154 × NIL 86-45)单株标记分型以筛选QTL区域内的交换单株。基于前期研究,标记T1341与QTL peak连锁最为紧密 (Brand et al., 2012),作者又在T1341附近开发出6个标记,在标记F23与H05间,共筛选出34个F4交换单株,可代表6种基因型组合,横跨Chr.1:5cM的区间。为排除pc1与pc10互作的影响,作者将所有交换单株的CaGLK2固定为1154等位基因。结果分析暗示,F23与LOL1是与QTL连锁最为紧密的标记,因为line 15含PI 152225最短的导入片段且叶绿素含量显著低于1154,而line 260在这2个标记间含1154等位且与1154叶绿素含量没有显著性差异。此外,lines 53与260在APRR2-like位点含有PI 152225等位,叶绿素含量却与1154没有显著差异,暗示此基因(APRR2)在本研究中的遗传背景下不影响叶绿素含量(表1.)。
CM334 v. 1.6 (Kim et al., 2017)已将上述4个scaffolds(scaffold1362,scaffold1381,scaffold1659与scaffold799)整合进Chr.1中,通过pc1区域的标记比对,F23到H05区间大小为2Mbp。
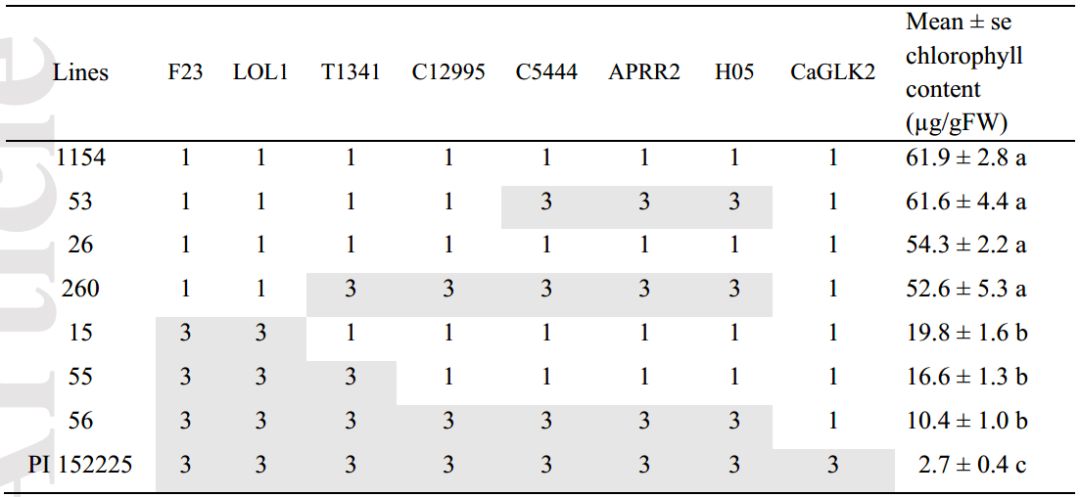
表1. pc1区域标记基因型与辣椒幼果叶绿素含量(双亲与6个重组系)
pc1 以果实特异性方式调控叶绿体区室大小与叶绿素含量
为确定pc1对果实发育的影响,本研究对line 1154(深绿NIL)与15(淡绿NIL)的叶绿体参数进行考察。叶绿素含量测定结果显示,1154与15在绿色辣椒幼果中存在显著差异(图 4.a,b),而在叶片中没有显著差异(图 4.c);激光共聚焦显微镜观察发现,与1154相比,line 15绿色幼果的叶绿体较小且具有较少的叶绿体数目(图 4.d,e)。这些结果暗示pc1通过调控叶绿素含量以及叶绿体大小来影响辣椒幼果颜色。
为考察幼果期叶绿体发育差异是否会影响成熟期果实特征,作者又比较了1154与15的几个成熟果实特征。结果显示,果实重量与白利度在两者间并无不同(图 4.f,g),然而,1154比15拥有更高的类胡萝卜素素总量(图 4.h)。
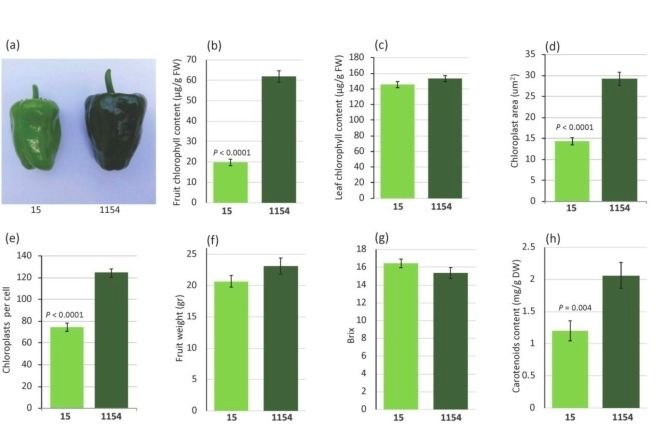
图 4. QTL- NILs(pc1)叶绿素含量、叶绿体大小以及成熟果实特征.
BSR-Seq分析(pc1区域)
为识别QTL区候选基因,解析其分子及细胞学机制,本研究对双亲(1154与PI 152225)及两个极端混池(1154 × NIL 86-45)进行转录组分析。BSR-Seq得到21.2,21,21.9与21.2 M个reads(1154,dark-green bulk,light-green bulk与PI 152225),共分析得到4114个纯合SNPs,其中2264个SNPs定位在Chr.1。SNPs分布峰值区间位于Chr.1:8 Mbp区间(即,13-21 Mbp;CM334 v.1.6),峰值中心落在18 Mbp(图3b,c),而在21.6 Mbp的位置SNPs数目突降。BSA-Seq (图 3a)与BSR-Seq (图 3b)比较发现,QTL峰值位点与SNP突降位置出现一个迁移,这是因为所用参考基因组不同所致,前者是CM334 v. 1.55(Kim et al., 2014),后者为CM334 v. 1.6 (Kim et al., 2017)。
差异表达候选基因分析(pc1)
与淡绿色亲本与混池对照相比,在1154与深绿色混池间共有240个基因发生上调表达,284个基因下调表达。
综合考虑重组交换(表1.),BSA-Seq与BSR-Seq(图3.)分析,本研究将QTL区间锚定在2.2 Mbp(19.4-21.6 Mbp),此区间共包含51个基因,其中,有11个基因在混池间发生差异性表达。深绿色混池中3个上调表达基因可能与叶绿体发育相关,因而被视为候选基因进行下一步分析。经过分析,第一个基因CA00g61750与第二个基因CA00g77840或与叶绿素含量不相关或表达量无显著差异,因而被排除;第三个基因CA00g77830,锌指转录因子LOL1,是拟南芥胁迫诱导细胞死亡的正调控因子,并且它的水稻同源基因过表达可提高叶绿素b含量,此外,CA00g77830在绿色组织高度表达,而在成熟果实中表达减少,暗示此基因在调控叶绿素含量方面发挥重要功能。
第四个差异表达基因CA00g77800,编码MIP1蛋白,在深绿色混池及亲本中呈下调表达。MIP1是否调控叶绿素含量并不清楚,但是拟南芥数据库STRING蛋白互作网络预测显示LOL1与MIP1存在互作。此外,MIP1在叶片与茎中表达量很低,在果实中表达量相对较高,并在成熟果实中达到最高,然而,在成熟期表达水平不变。
pc1 参与调控辣椒幼果的光合与氧化还原过程
为研究pc1参与影响的细胞学过程,本研究对混池间983个差异表达基因进行GO富集分析并根据生物过程进行分类(图5)。最显著的GO term过程与光合作用及氧化还原有关,这两个过程包含184个DEGs,被分成17个亚类,其中最大的一类是氧化还原过程。这一类基因与叶绿素合成下调有关,例如,POR,CAO与HEMA1,还与叶绿素降解基因上调有关,例如,CA00g73130,暗示叶绿素合成与降解受这个QTL调控。另一类重要基因,包含Chlorophyll a/b-结合蛋白的多个成员基因(CA01g17090,CA02g12050,CA02g12060,CA02g12070等),在淡绿混池中下调表达,它们参与光合、蛋白发色团及光刺激响应等过程。另外,若干与碳固定有关的Rbc家族基因在淡绿混池中也呈现下调表达。
多个DEGs的上调或下调表达与氧化胁迫或细胞氧化还原稳态相关,例如,CA03g06870 (Superoxide dismutase),CA10g19020 (Thioredoxin),CA00g63370 (Dihydrolipoyl dehydrogenase)等。此外,其它重要过程包括碳水化合物代谢、脂质代谢、次级代谢、类黄酮合成以及DNA复制。
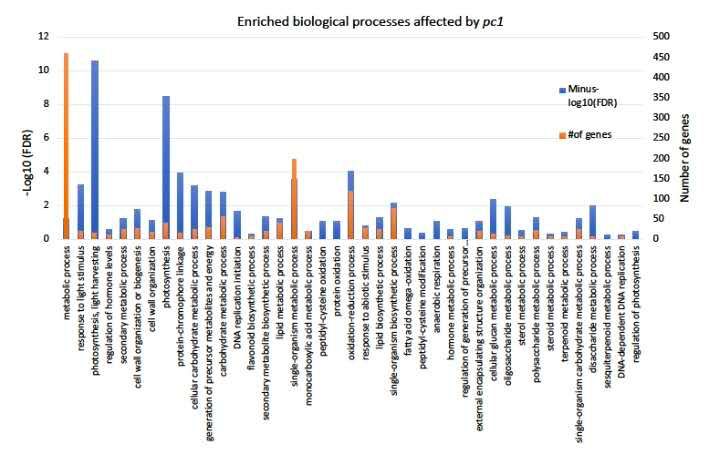
图 5. 混池间差异表达基因的GO生物过程分类(FDR ≤ 0.05).
CcLOL1与CcMIP1在C. chinense PI 152225中发生移码突变
对1154与PI 152225中的CcLOL1 ORF测序分析发现,在PI 152225中26bp处存在单碱基C的插入,引起移码突变,在第19个氨基酸处造成提前终止,在line 15中包含同样的突变;而在C. annuum与C. annuum var glabriusculum中虽然叶绿素含量变异显著,但是它们的CcLOL1 ORF序列均与1154相同。因此,作者又进一步分析了CcLOL1在12个品种中表达情况与叶绿素关系,然而,两者之间的相关系数并不显著。
对1154与PI 152225中的CcMIP1 ORF测序分析发现,在PI 152225中570 bp处存在32 bp的插入,引起移码突变,在第209个氨基酸处造成提前终止。除了C.annuum var glabriusculum lines Nayoi与1202在相同位置有32 bp插入之外,其它品种CcMIP1序列均与1154一致。
C. chinense幼果果色变异遗传位点等位多样性分析
由于在PI 152225中CcLOL1与CcMIP1及CaGLK2都是突变等位,因此作者又对另外10个C. chinense品种测序分析这3个基因以及CcAPRR2以求获得等位多样性并分析出它们与叶绿素含量的关系。结果暗示极端淡绿果色的3个基因(CcLOL1、CcAPRR2、CaGLK2)均是突变等位,而C. annuum 1154在这些位点都是野生型等位基因。对于叶绿素含量中等的一些品种,3个基因其中有1个为突变等位:CcLOL1 (164-3,180-2,PI 159236与CA4),CaGLK2(lines 187-4与USDA 162)与CcAPRR2(line 187-2)。深绿果色品种165-2突变CcAPRR2后,会引起叶绿素含量降低;而被诱变后幼果叶绿素含量变化并不一致,暗示CcMIP1其独立于CcLOL1。
CRISPR/Cas9敲除番茄基因SlLOL1 导致幼果呈淡绿色
为证明CcLOL1参与调控叶绿体发育与叶绿素含量,本研究利用CRISPR/Cas9技术敲除番茄栽培种MP1中的辣椒同源基因Solyc08g077060(SlLOL1)获得突变体。首先设计两个gRNAs靶定exon 2,两者相距91 bp;然后遗传转化番茄MP1,经阳性苗筛选,检测,最终获得4类突变体lol1CR-25,lol1CR-43,lol1CR-57与lol1CR-58,或缺失,或插入,经T1与T2代测序验证获得纯合突变体(图 6.a,b)。
与MP1相比,突变体幼果颜色明显变浅,尤其是近端肩区(图6.c);并且无论是近端区还是远端区,果实中叶绿素含量显著降低(图 6.d,e)。然而,在成熟叶片中叶绿素含量没有变化,SlLOL1以果实特异性方式调控叶绿素含量。为了检测叶绿素含量降低是否是由叶绿体变小所致,作者分析了lol1CR-43与MP1幼果近肩区果皮中叶绿体面积与数目。结果显示,lol1CR-43中叶绿体显著变小,也就是说突变体中叶绿体面积与数目明显低于MP1(图 6.g,h)。
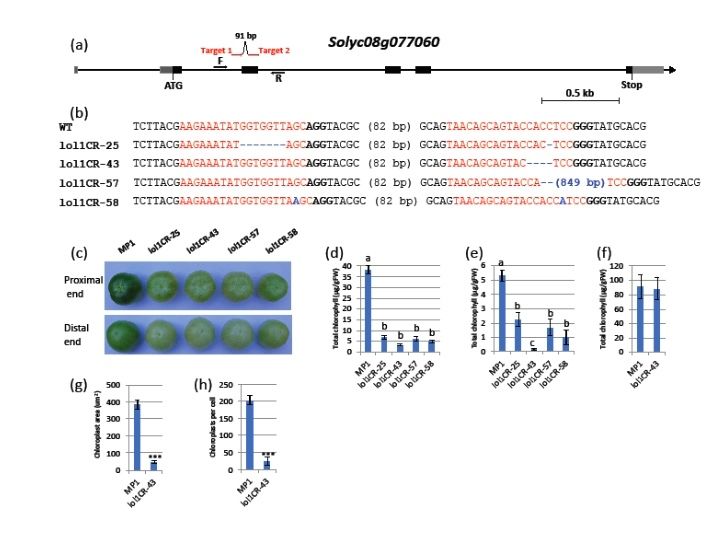
图 6. CRISPR/Cas9敲除番茄基因SlLOL1导致幼果叶绿素含量降低
pc1与pc10 互作影响辣椒幼果叶绿素含量
为分析pc1与pc10对叶绿素含量的影响,本研究利用QTL-NILs 1976,1975与1973(分别突变pc1,pc10以及pc1pc10)进行比较分析。与1154相比,NIL-1975 and NIL-1976 叶绿素含量分别降低49%与41%,而NIL-1973降低33%;双QTL突变NIL-1973效应比期望值(90%)显著小,暗示pc1与pc10间的互作呈现非加性效应(图7.a,b)
为检测CcLOL1与CaGLK2表达水平是否相互影响,作者分析了不同NILs在幼果发育的两个时期CcLOL1与CaGLK2以及CcAPRR2与CcMIP1的表达情况。结果显示,CcLOL1的表达水平不受CaGLK2 (NIL 1975)突变的影响,并且在NILs 1976与973中表达水平相似;相似地,CaGLK2的表达不受CcLOL1与CcMIP1 (NIL 1976)突变的影响,并且在NILs 1975与1973中表达水平相似;这些结果暗示了CcLOL1与CaGLK2的表达彼此独立。CcAPRR2在NILs 1975与1973授粉后30天表达上调,但是在NIL 1976中不受影响,暗示CaGLK2对CcAPRR2有抑制作用。需要说明的是, CcMIP1与CcLOL1紧密连锁(相距77 Kbp),因此两个基因突变等位一起被回交到NILs 1973与1976中。此外,CcMIP1仅在NILs 1973与1976授粉30天后上调表达,且在混池间呈现相似表达模式,说明此基因表达不受CaGLK2影响。
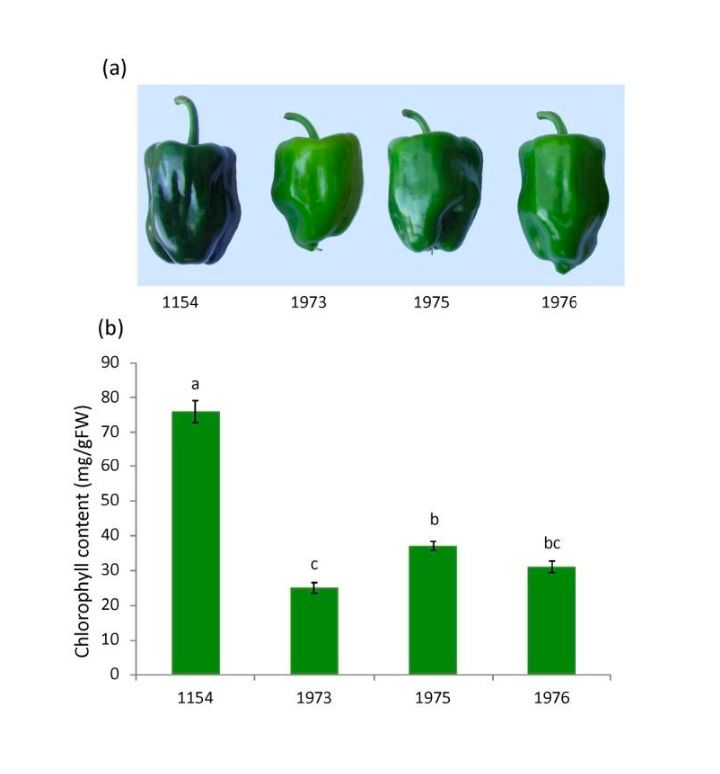
图 7. pc1与pc10-NILs幼果叶绿素含量(分别突变CcLOL1与CaGLK2).
结论
1. CcLOL1即为pc1靶标基因,调控辣椒果实中叶绿体大小与叶绿素含量变异
2. CcLOL1主要调控C. chinense幼果果色变异,而对于C. annuum调控能力有限
3. CcLOL1,CaGLK2与CcAPRR2是高度分化的并与C. chinense与C. annuum var glabriusculum幼果果色相关
4. pc1与pc10功能彼此互补,而CcLOL1与CaGLK2的表达互不影响
研究思路总结
利用近等基因系与轮回亲本杂交获得F2,然后混池测序,即BSA-Seq与BSR-Seq,结合候选区域交换单株筛选,寻找重组断点,转录组分析差异表达基因,分析与目标性状相关候选基因,同时分析品种间候选基因序列变异与表型变异之间的关联性,接着,CRISPR敲除候选基因验证功能,最后,考察表型相关指标与QTL或基因互作模式之间的相关性。
如果您的科研项目有问题,欢迎点击下方按钮咨询我们,我们将免费为您设计文章方案。
参考文献:
Borovsky, Y., Monsonego, N., Mohan V. et al. (2019) The zinc-finger transcription factor CcLOL1 controls chloroplast development and immature pepper fruit color in Capsicum chinense and its function is conserved in tomato. Plant J. doi: 10.1111/tpj.14305.
Brand, A., Borovsky, Y., Meir, S. et al. (2012) pc8.1, a major QTL for pigment content in pepper fruit, is associated with variation in plastid compartment size. Planta, 235, 579-588.
Brand, A., Borovsky, Y., Hill, T. et al. (2014) CaGLK2 regulates natural variation of chlorophyll content and fruit color in pepper fruit. Theor. Appl. Genet. 127, 2139–2148.
Kim, S., Park, M., Yeom, S.-I. et al. (2014) Genome sequence of the hot pepper provides insights into the evolution of pungency in Capsicum species. Nature Genet. 46, 270-278.
Kim, S., Park, J., Yeom, S.-I. et al. (2017) New reference genome sequences of hot pepper reveal the massive evolution of plant disease-resistance genes by retroduplication. Genome Biol. 18, 210.









 京公网安备 11011302003368号
京公网安备 11011302003368号