2018年11月,浙江大学蒋立希、吴德志团队联合新加坡国立大学俞皓教授团队与百迈客合作,完成了991份源自世界39个国家(地区)的油菜种质资源基因组测序并研究其生态差异。相关研究成果“Whole-genome resequencing of a world-wide collection of rapeseed accessions reveals genetic basis of their ecotype divergence”于2018年11月23日发表在国际知名期刊 Molecular Plant 杂志上。
英文题目:Whole-genome resequencing of a world-wide collection of rapeseed accessions reveals genetic basis of their ecotype divergence
中文题目:全球油菜种质资源全基因组测序揭示了其生态型差异的遗传基础。
研究单位:浙江大学作物科学研究所
发表时间:2018年11月23日
发表杂志:Molecular Plant
影响因子:9.326
研究背景
油菜(Brassica napus L.)是世界上食用油和富含蛋白质的牲畜饲料的重要来源。甘蓝型油菜B.napus (AACC)起源于距今不到7500年的两个二倍体祖先白菜(B.rapa(AA)(n=10))和甘蓝(B.oleracea(CC)(n=9))之间的种间杂交。尽管油菜的进化历史比亲本品种短,但它已经适应了不同的气候区和纬度,并形成了三个主要的生态型群,即冬季型、半冬季型和春季型。
为了在相应的环境中生存,适时的开花至关重要,并且花期对油菜的生命周期、产量和种子质量起着至关重要的作用。在模式植物拟南芥中,多条开花途径响应环境和发育信号,并汇聚到FLOWERING LOCUS T (FT)的转录调控上,其蛋白质充当移动的花原信号,从叶子移动到茎尖分生组织以触发开花。FT的表达被光周期途径中的CONSTANS(CO)激活,并且被FLOWERING LOCUS C (FLC)抑制,其mRNA表达被春化和自主途径同时抑制。
虽然已经报道了油菜的冬季、半冬和春季生态型的遗传变异,但不同的生态类型在甘蓝型油菜中的差异的关键等位基因变异尚不完全清楚。
材料与方法
材料及来源:来自39个国家的991个甘蓝型油菜种质资源,包括658个冬季类型、145个半冬季类型和188个春季类型。
测序平台: Illumina HiSeq XTen
参考基因:‘Darmor-bzh’; ‘Tapidor’
分析内容:系统发育与种群结构分析;PCA、LD分析;基因流分析;选择性清除分析;花期性状GWAS;花期相关SNPs进行RT-qPCR分析。
研究结果
991份甘蓝型油菜种质资源的再鉴定
与参考基因组(“Darmor-bzh”)相比,在991个油菜基因组中检测到556万个SNP和186万个InDels。(表1)。并检测了它们在甘蓝型油菜基因组区域的分布。比较A和C亚基因组之间的SNP分布显示,A亚基因组中的SNP频率(7.9 SNPs/kb)高于C亚基因组中的SNP频率(5.9 SNPs/kb)。并将过滤后鉴定出的2753575个可信的SNPs(缺失数据<50%,MAF>5%)用于后续分析。

表1 不同生态型油菜基因组多态性及变异
991个群体的群体结构和基因组变异
991份油菜大致与它们的地理分布相对应,即分别是欧洲、亚洲和欧亚大陆/北美(图1A)。系统发育、种群结构和主成分分析(PCA)结果表明,991份油菜品种可分为三组(K=3)(图1BCD),大致相当于冬季、半冬季和春季生态类型,说明冬、半冬和春季生态型在遗传上存在差异。与参考基因组“Darmor-bzh”相比,分别在冬、半冬和春季材料寻找SNPs与InDels。冬型的核苷酸多样性(π=8.42×10-4)低于半冬型(π=10.16×10-4)和春型(π=9.92×10-4),表明在冬季类型中低频SNPs的存在主要是由于“Darmor-bzh”(也是一种冬型种质资源)的mapping所致。
进一步分析连锁不平衡在整个基因组中的表达,发现冬、半冬和春季生态型SNPs之间的物理距离(最大值的一半)分别为11.27 kb(r2=0.30)、24.55 kb(r2=0.32)和17.40 kb(r2=0.31)。(图1E)LD衰变的大小在不同染色体和不同亚基因组之间变化很大。在冬型、半冬型和春型基因组中分别检测到100、5460和631个大的LD结构(>10 kb),表明冬型油菜的遗传重组频率高于其它类型油菜的遗传重组频率,从而破坏了基因组LD结构。此外,在A亚基因组中,LD衰变速率比在C亚基因组中更快,这表明A亚基因组中的遗传重组频率高于C亚基因组中的遗传重组频率(图1F)。
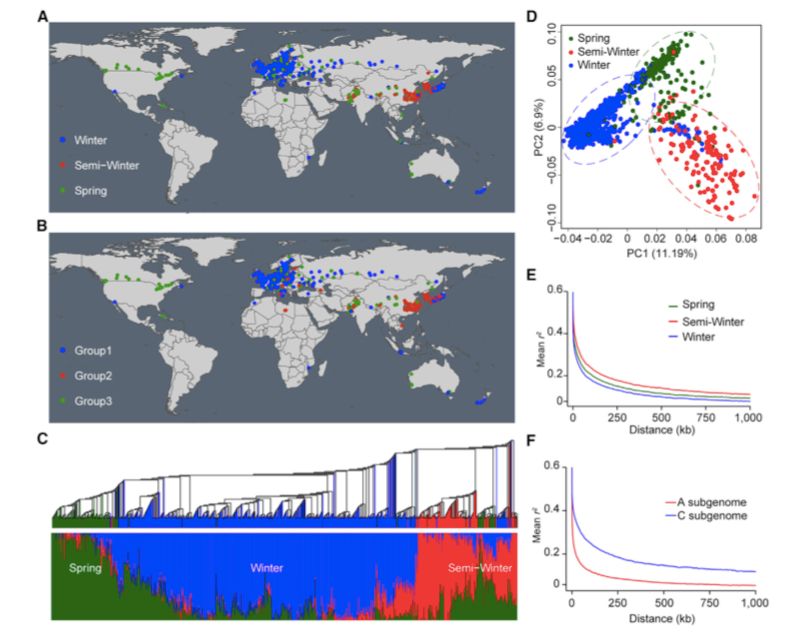
图1 991份油菜种质资源的分布、种群结构、PCA和LD衰变
主要种群间的等位基因漂移路径
进一步计算世界地图上显著的基因流数目之后,在冬型和春型油菜中发现了四个主要基因流(图2C)。冬型油菜的基因流从法国到俄罗斯(weight=0.5)、法国到瑞典(weight==0.3)、波兰到日本(weight==0.3)和日本到中国(weight==0.3),而春型油菜的基因流从法国到俄罗斯(weight==0.5)、法国到瑞典。经德国(weight=0.4),瑞典到加拿大(weight=0.2),加拿大到中国(weight=0.5)。没有显著的生态型漂移事件(inter-ecotype drift events)(图2 A、B)。
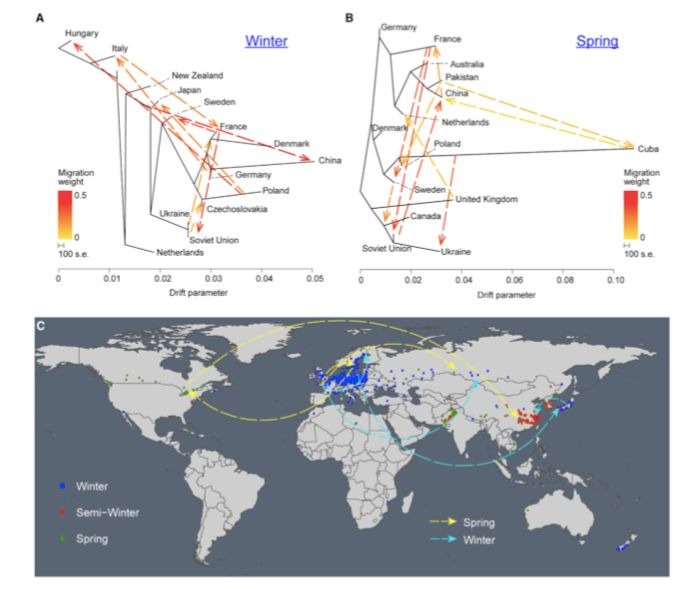
图2 基因漂移
甘蓝型油菜自然选择和人工选择产生的信号
为了确定自然选择和人工选择对历史上基因的改变,作者对三种生态型之间进行选择性清除分析。春冬季型、半冬和冬季型、春季和半冬型之间分别有61、56和64个选择性清除(表1;图4)。位于选择性清除区域上的基因可能发挥重要作用。在Chr.A10上发现了最强的信号,该信号是冬春型之间基因组变化的基础(图3A)。该区域有56个带注释的基因,包括FLC直系同源基因(BnaA10g22080D)和AIL6直系同源基因(BnaA10g21750D),编码AP2-like乙烯反应转录因子(ERF)。在含有377个带注释基因的Chr.A02上发现了另一个强选择性信号,包括FT (BnaA02g12130D)、FY (BnaA02g01670D)、CONSTANS-like (BnaA02g13920D)、ERF118 (BnaA02g13950D)和ERF118-like(BnaA02g13960D)以及组氨酸transporter-like 2乙烯转运体(BnaA02g13290D)。编码几个MADS-box基因的Chr.C01、C02、C08和C09上的一些区域显示出弱清除信号。发现在有1404个基因在冬春两季类型之间发生选择性清除,这些基因包括花期控制、乙烯生物合成和信号传导以及植物应激反应的基因。此外,在半冬型和冬型之间以及春季型和半冬型之间的种群分化的清除区域内共鉴定出1228和1370个基因。鉴定出许多与开花时间调控相关的基因,如CONSTANS-like和MADS-box基因,或植物激素信号传导基因,如ERFs以及生长素和赤霉素诱导基因。GO分析表明,这些基因涉及多种生物学过程、细胞成分和分子功能。

图3 三种油菜生态型之间的选择性清除信号
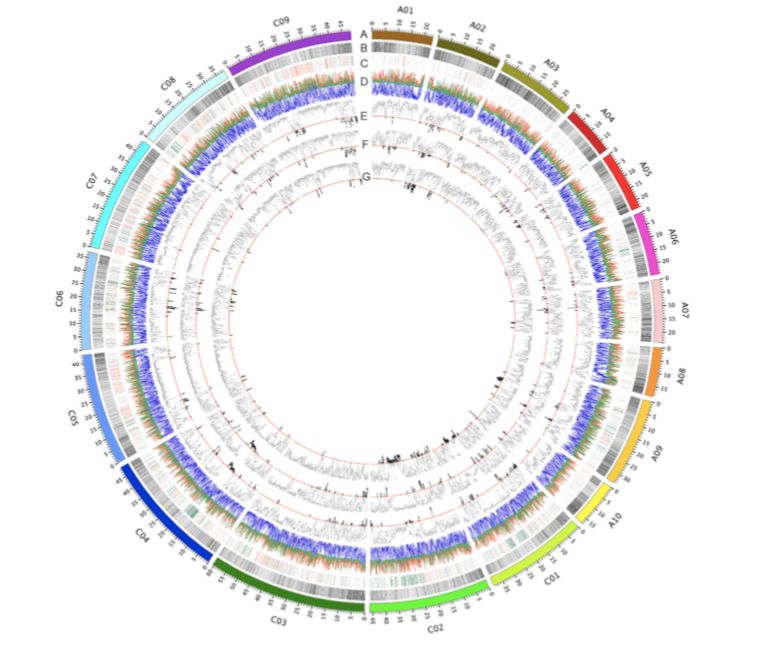
图4三种生态型之间的遗传多样性和选择性信号circos图
甘蓝型油菜开花时间相关基因的鉴定
开花期是油菜品种驯化和育种的重要性状之一,也是油菜三个生态型的重要区别之一。观察了不同品种之间的开花时间变化(125-191 d)。曼哈顿试验表明,在Chr.A02的4-7 Mb区域,许多SNPs与开花时间显著相关(-log10P>6)(图3B和图5)。在6.37~6.38 Mb区域内鉴定了拟南芥FT(BnaA02g12130D)的直系同源序列,其中的SNPs与开花时间密切相关(图5)。对100 kb(6.37~6.38Mb)序列进一步的LD分析表明,与开花时间相关的SNPs位于FT直系同源起始密码子的上游~3 kb(6372995~6375936 kb)(图5A和5B)。

图5 Chr.A02上开花时间从约6.37 Mb-6.38 Mb的SNPs
BnaA10g22080D是拟南芥FLC的一个假定的同源基因,在Chr.A10的14.99-15.01 Mb之间的区域内被鉴定(图3B)。GWAS和LD分析表明,BnaA10g22080D的5’调控区的SNPs与品种间开花时间的变化显著相关(图6A和6B)。

图6 Chr.A10染色体上约14.99-15.00 Mb的花期SNPs
与“Tapidor”基因组进行比对
为了验证上述结果的可靠性(即种群结构、选择信号和开花时间的GWAS分析),通过将reads mapping到另一个“Tapidor”参考基因组进行call SNPs。共获得553万个SNPs和192万个InDels。群体结构分析和PCA分析结果与mapping “Darmor-bzh”参考基因组(简称SNP-Darmor)的结果相同。比较了LD衰变率之间的“SNP-Darmor”和“SNP-Tapidor”,得到一致的结果。对开花时间的选择性清除分析和GWAS分析证实,A02上的FT同源性(BnaA02g12130D)和A10上的FLC同源性(BnaA10g22080D)与开花时间变化显著相关。从而得出:利用群体遗传学方法,如PCA、LD率、选择性信号和GWAS,对两个SNP集(SNP-sets)进行分析得出相同的结论。
生态型差异对应的SNPs鉴定
FT和FLC直系同源基因编码区的核苷酸序列在所有三种生态类型中都相当保守。在BnaA02g12130D的5’上游地区32个SNPs中,10个SNPs在这三种生态类型之间有特异性差异。95±2%的冬型种质在BnaA02g12130D起始密码子上游上具有与参照(“Darmor-bzh”)相同的核苷酸,而在85±6%的春型种质中,相应位点的核苷酸以不同的方式改变,如鸟嘌呤(G)向胞嘧啶(C)转化以及腺嘌呤(A)向鸟嘌呤(G)的转化。只有22±3%的半冬型品种与春型品种有相同的变化,其中40%的半冬型品种与春型品种有相似的T到C的变化。半冬型品种表现出与春型品种相似的核苷酸变化(图5C)。
FLC直系同源基因推测的启动子区(BnaA10g22080D)中各种SNPs的频率在三种生态类型之间有很大差异(图6C)。为了研究生态型特异性SNPs与甘蓝型油菜FT或FLC转录是否相关,用FT (BnaA02g12130D)和FLC (BnaA02g12130D)的特异性引物对各种FT和FLC同源物的表达水平进行了比较。结果和已发表的转录组数据一致表明:春季型品种叶片中BnaA02g12130D的表达水平显著高于其他两种生态型(图5D),而BnaA10g22080D在春型品种叶片中的表达在所有三个生态型中都最低(图6D)。与春、冬型品种间FT和FLC同源基因表达显著差异相比,半冬型品种与其它两种类型品种间的表达差异较小(图5D和6D)。这些结果表明,生态型特异性SNPs与FT和FLC同源物的表达水平相关,这有助于三种生态型间形成不同的开花时间。
在春与冬、春与半冬、半冬与冬的选择性清除分析下,推测有12~15个乙烯基因。在Chr.A02上仅推测ERF119直系以及Chr.A10上推测的AP2-like ERF (AIL6)直系显示与生态型差异相对应的有区别的SNPs。这些生态型特异性SNPs与已发表的转录组数据中的ERF119和AP2-like ERF(AIL6)的转录水平相关,这使三种生态型表现出不同的营养生长期。
总结
该研究对991份油菜天然种质资源进行了遗传多样性的测序和鉴定。通过对两个不同的参考基因组,即“Darmor-bzh”和“Tapidor”基因组的定位读数产生的两组SNPs的群体遗传学分析,得出了一致的结论。遗传多样性和连锁不平衡参数表明甘蓝型油菜两个亚基因组的进化是不对称的。选择性扫描分析揭示了与调控植物发育和应激反应的各个方面相关的基因的遗传变化。并对影响开花时间的SNPs进行了探索。
整个研究科学而严谨,为油菜分子标记的筛选提供了重要依据,为油菜分子标记在油菜育种中的应用奠定了基础。
如果您的科研项目有问题,欢迎点击下方按钮咨询我们,我们将免费为您设计文章方案。








 京公网安备 11011302003368号
京公网安备 11011302003368号