研究疾病发生机制?
DNA序列变异、基因表达变化、非编码RNA调控机制、DNA甲基化修饰…
除了这些,我们还能做什么?
当然是基因组表观层面的三维构象啦
用三维的视角,你会发现不一样的风景
作为染色体三维结构与互作研究的利器,Hi-C技术可揭示染色体片段间的互作信息,阐述染色体三维结构,常常与WGS、WGBS、RNA-seq等技术联合,从DNA线性序列、表观修饰、三维构象、转录调控等多组学层面,研究疾病发生发展机制。
今天给大家介绍的这篇文章中,作者利用Hi-C技术揭示了染色体三维结构在心力衰竭中的作用,构建了发育和疾病发生中表观基因组的变化模型。
 研究背景
研究背景
心力衰竭是一种毁灭性的疾病,虽然心力衰竭是一种多器官疾病,但它是由心肌细胞生物学的变化驱动的,这种复杂的细胞功能障碍的基础是基因表达的变化,而细胞核内基因组的正确折叠决定了适当的基因表达和细胞功能,先前的研究已经证明了组蛋白修饰酶和这些修饰的识别酶在其中发挥重要作用。但是心肌细胞表观基因组的基本折叠原理还未被揭示,基因组的三维结构是否会导致心血管疾病还有待确定。
在此研究中,作者使用两种疾病模型成年鼠进行Hi-C,研究心力衰竭小鼠中全基因组范围内的三维结构特征,由于转录组分析是表观基因组结构变化的功能解读,因此作者结合RNA-seq获得基因表达图谱,以揭示基因组三维结构在心力衰竭中的作用。
研究方法
材料:正常对照成年小鼠(CTRL),两个不同的疾病模型–压力超负荷性心肌肥厚成年小鼠(TAC)、染色体结构蛋白CTCF敲除成年小鼠(CTCF-KO),分离心肌细胞。
研究结果
疾病模型小鼠病理表型
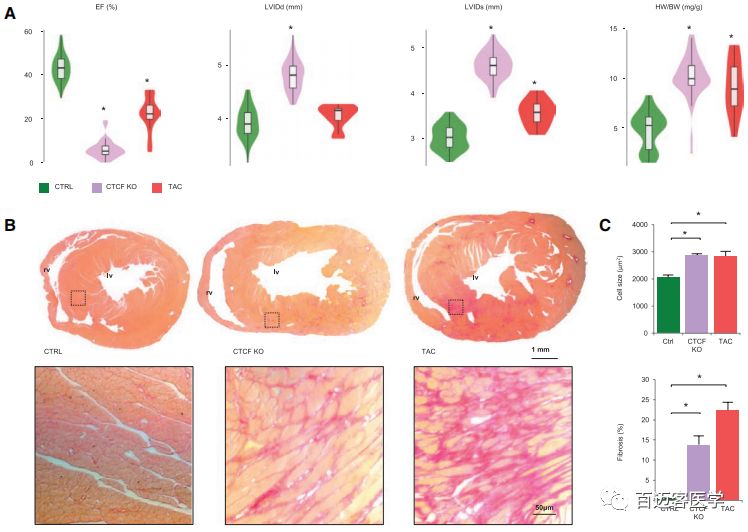
对CTCF-KO小鼠的表型呈现明显的心肌病,失去CTCF会导致射血分数受损、左心室扩张以及器官和细胞水平的肌肉肥厚。而TAC小鼠也表现出肥厚,并伴有较轻微的心室大小和射血分数的变化,这表明CTCF-KO和TAC小鼠代表了不同的病理生理模型。
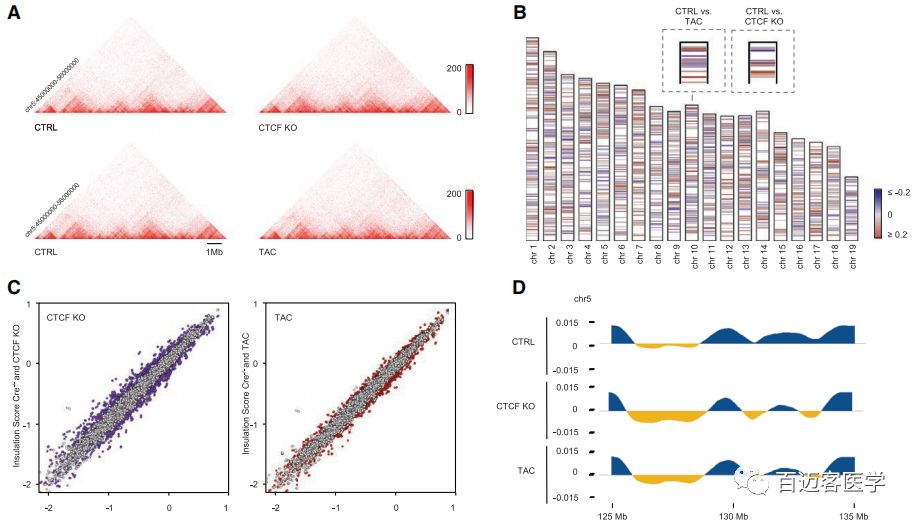
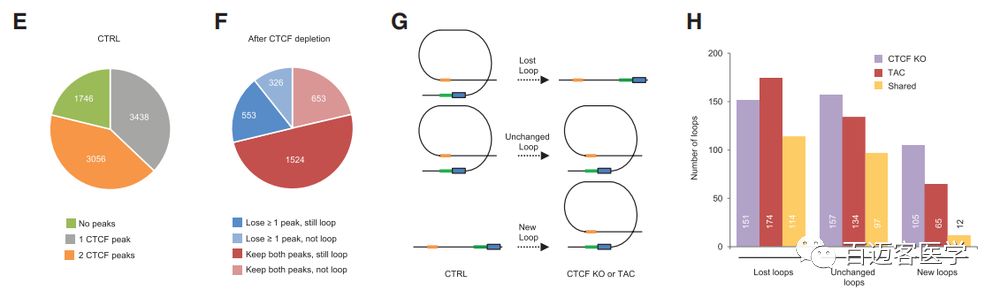
CTRL、CTCF-KO、TAC小鼠的TAD结构分析显示,三组的TAD数量分别为3686、3746和3705个,虽然整个基因组的TAD边界数量变化小于2%,但是边界强度发生了不同的变化。此外,CTCF-KO、TAC小鼠的染色体均有新的TAD边界,包括发生边界的转移和新边界的形成。
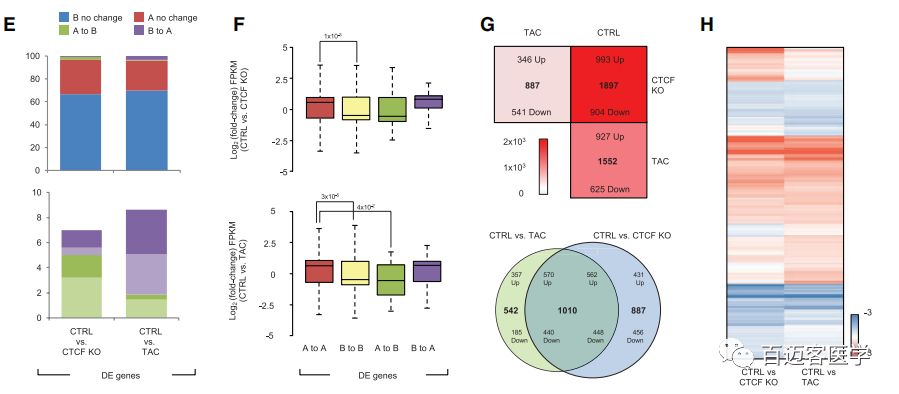
CTCF的缺失或TAC对染色体的A compartment和B compartment的影响较小,而其中发生compartment转换的区域与基因表达呈正相关,即从A compartment到B compartment转换区域的基因表达下调,从B compartment到A compartment转换区域的基因表达上调。CTCF-KO、TAC小鼠间存在41%的共有差异表达基因,两者都表现出已知的标志基因激活。ChIP-seq检测到CTCF靶向基因的转录起始位点,而这些基因富集在与心脏病相关的通路中。
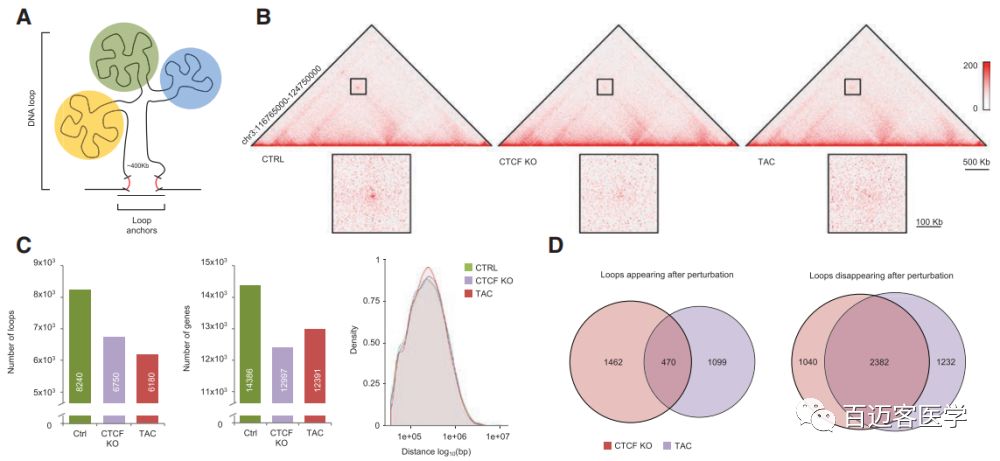
疾病心肌细胞的染色体长距离loop特征
CTCF-KO、TAC小鼠的染色体长距离loop大小没有明显变化,但loop总数量减少。相比于对照组,CTCF-KO和TAC中消失的长距离loop中有51%两者共有的,而新出现的loop中只有15%是两者共有的;在疾病细胞中丢失的loop数大于新形成的loop数。在疾病细胞中发生重组(丢失/新形成)的loop中,富集与心血管功能相关通路中的基因。此外,重组loop中亦包含大量增强子–启动子loop,其中在疾病细胞中丢失的loop富集与心脏病理相关的基因。
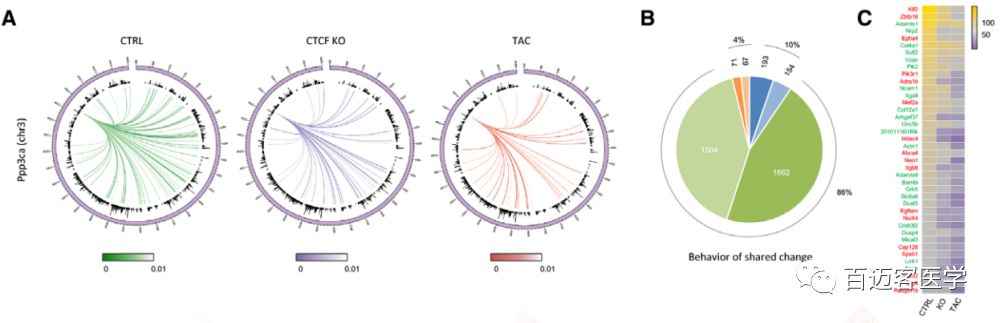
增强子-基因互作的改变
存在互作的9194个表达基因中,60%的基因在CTCF-KO和TAC中的互作变化方向相同。而剩余的40%基因中大多数(75%)是在某一组疾病细胞中虎作没有发生变化的,25%是互作变化在两组中相反的。在CTCF-KO和TAC中互作改变相同且差异表达的3651个基因中,绝大多数(86%)的局部互作减少(图4B)。这些发现支持了这样一种趋势,即更多的动态染色质互作影响基因表达,两组疾病细胞与对照相比,它们的基因表达变化方向更可能趋向一致。互作数量top 40的差异表达基因在疾病细胞中的互作持续下降,如与钙依赖信号和心肌肥厚相关的基因Ppp3ca。
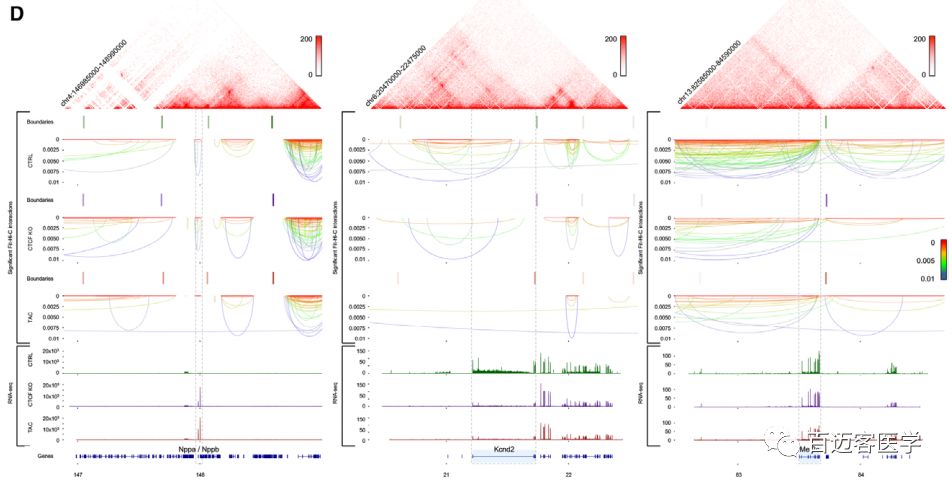
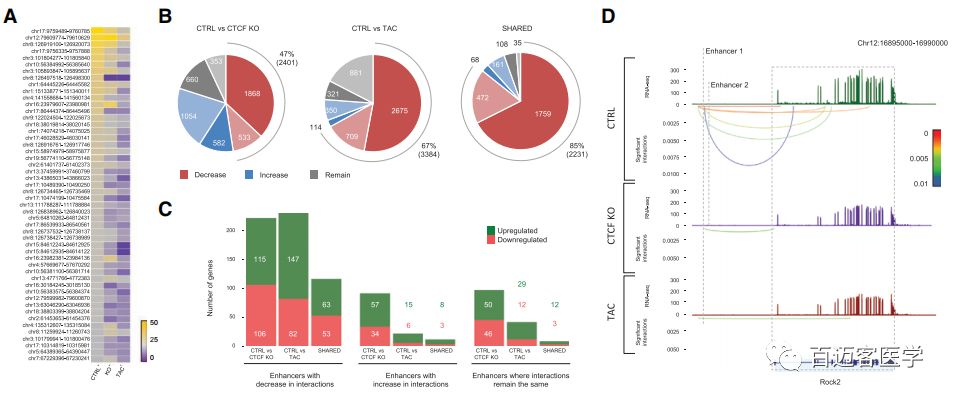
在先前鉴定的成人心肌细胞增强子中,其中有115050个再本研究中存在互作。这些增强子的互作在疾病细胞中减少。事实上,其中47%在CTCF-KO中表现出了互作减少,67%在TAC中表现出互作减少。两种疾病细胞中共有的增强子中,85%在两者中均出现互作减少。且两种疾病细胞中基因表达变化与其增强子-基因互作的减弱有关,且受影响的基因在心脏病理通路中富集,如Rock2。
小结
正常对照成年小鼠(CTRL)和两个不同的疾病模型(TAC、CTCF-KO)小鼠的Hi-C结果揭示了5 kb分辨率下心肌细胞的基因组相互作用,得到了染色体内和染色体间的互作事件,并为未来的心脏表观基因组研究提供了依据。压力超载或CTCF缺失选择性地改变了TAD的边界强度和A/B compartment、基因组可及性。心力衰竭涉及致病基因周围染色质互作稳定性的降低。此外,压力超负荷或CTCF缺失重塑了心脏增强子的长距离互作,导致这些功能元件周围局部染色质互作显著减少。
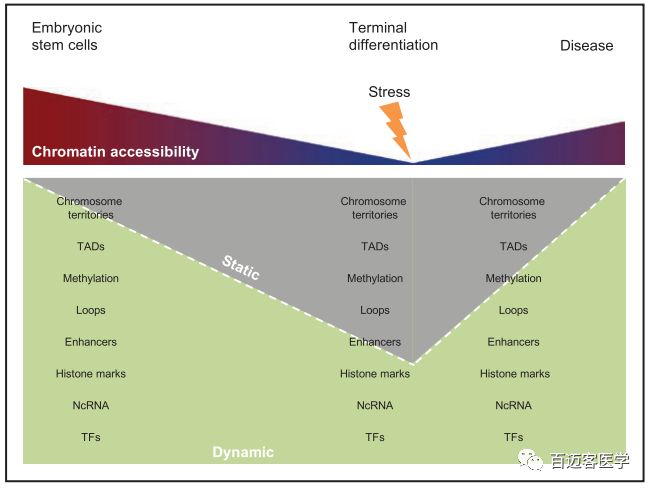
作者以此研究为基础,结合前人研究结果,构建了发育和疾病发生中表观基因组的变化模型(如下图)。发育过程中伴随着染色质的变化,使最终分化的细胞具有稳定的转录组,而疾病破坏了这种平衡,通过影响染色质结构、增强子-基因loop、组蛋白修饰、DNA甲基化等,将基因组的特定区域转变为更动态的构象。
参考文献
Rosa-Garrido M, Chapski D J, Schmitt A D, et al. High Resolution Mapping of Chromatin Conformation in Cardiac Myocytes Reveals Structural Remodeling of the Epigenome in Heart Failure[J]. Circulation, 2017.
如果您的项目有任何问题,欢迎点击下方按钮咨询我们。








 京公网安备 11011302003368号
京公网安备 11011302003368号